В данном практикуме для ранее выбранных животных (смотри практикум 1) реконструируется дерево по последовательностям малой РНК митохондриальных рибосом (12S rRNA) с помощью программы fastme.
Но для построения данного дерева были взяты 2 других вида: Scarturus williamsi и Panthera tigris altaica - вместо Scarturus elater (SCAEL) и Panthera tigris sumatrae (PANTS) соответственно, поскольку митохондриальные геномы этих ранее выбранных животных не были отсеквенированы полностью.
| Вид | Таксономия | Мнемоника |
|---|---|---|
| Scarturus williamsi | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Euarchontoglires; Glires; Rodentia; Myomorpha; Dipodoidea; Dipodidae; Allactaginae; Scarturus | SCAWI |
| Panthera tigris altaica | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Boreoeutheria; Laurasiatheria; Carnivora; Feliformia; Felidae; Pantherinae; Panthera; Panthera tigris | PANTA |



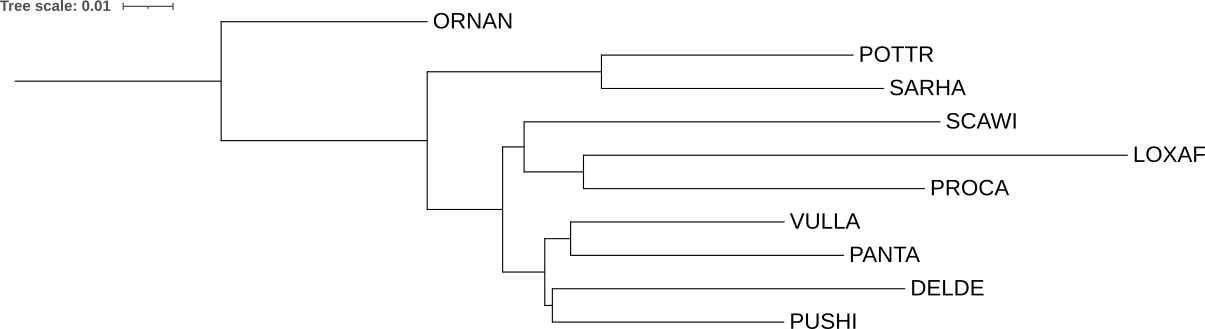
У построенного по нуклеотидным последовательностям дерева, если сравнивать с деревом из практикума 1 (это референсное дерево - Рис. 2.), есть 1 клада, которой нет на референсном дереве, это клада с Delphinus delphis (DELDE) и Pusa hispida (PUSHI), хотя PUSHI должен быть объединён с Vulpes lagopus (VULLA), те в свою очередь должны быть объединены с Panthera tigris altaica (PANTA), который правильно объединён с VULLA, а все эти животные глобально должны быть объединены в кладу с DELDE, при этом Scarturus williamsi (SCAWI) был правильно объединён с Procavia capensis (PROCA) и Loxodonta africana (LOXAF), но ошибочно отделён от большой клады с DELDE.
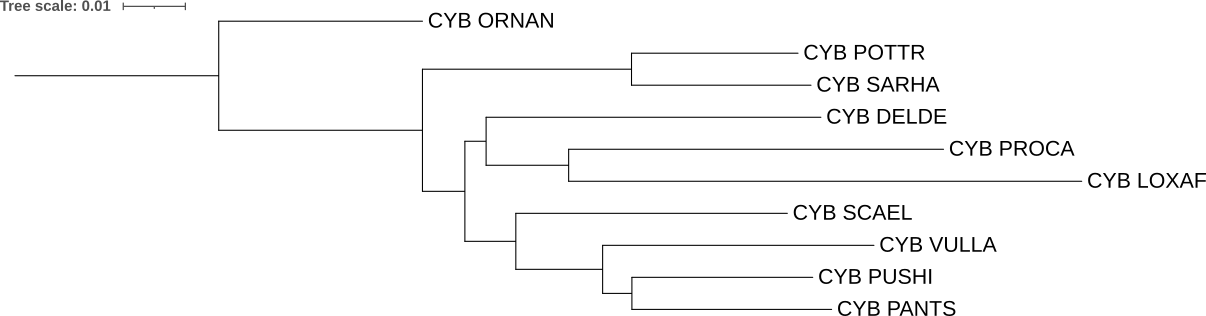
Если сравнивать с деревом, построенным по последовательностям цитохромов B с помощью программы fastme по модели p-distance (Рис. 3.), Delphinus delphis (DELDE) был ошибочно объединён в одну кладу с Pusa hispida (PUSHI), хотя DELDE должен быть объединён с Procavia capensis (PROCA) и Loxodonta africana (LOXAF) вместо Scarturus williamsi (SCAWI), который должен быть объединён с Vulpes lagopus (VULLA) и c объединёнными в одну кладу Panthera tigris altaica (PANTA) и PUSHI.
Если сравнивать с деревом, построенным по последовательностям цитохромов B с помощью программы iqtree (Рис. 4.), Delphinus delphis (DELDE) был ошибочно объединён в одну кладу с Pusa hispida (PUSHI), хотя DELDE должен быть объединён с большой кладой, где есть Scarturus williamsi (SCAWI), объединённые в одну кладу Procavia capensis (PROCA) и Loxodonta africana (LOXAF), Vulpes lagopus (VULLA), Panthera tigris altaica (PANTA) и PUSHI, при этом PUSHI и PANTA также должны быть объединены в одну кладу.
Было подобрано животное, которое служит внешней группой относительно ранее подобранных животных - это Aldabrachelys gigantea. Все выбранные в практикуме 1 животные относятся к классу Млекопитающих, а Aldabrachelys gigantea (гигантская черепаха) относится к классу Пресмыкающиехся.
| Вид | Таксономия | Мнемоника |
|---|---|---|
| Aldabrachelys gigantea | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Sauropsida; Sauria; Archelosauria; Testudinata; Testudines; Cryptodira; Durocryptodira; Testudinoidea; Testudinidae; Aldabrachelys | ALDGI |

После укоренения во внешнюю группу дерево, построенное по последовательностям 12S rRNA, не изменилось (сравни с Рис. 1.), то есть Ornithorhynchus anatinus, действительно, является внешней группой на дереве, построенном по нуклеотидным последовательностям 12S rRNA (смотри Рис. 1.).
Бутстреп метод проверки достоверности топологии дерева.
Из входного выравнивания делается, например, 100 бутстреп-реплик, каждая из которых представляет собой результат случайного выбора столбцов из выравнивания в том же количестве, что и в исходном выравнивании (при этом какие-то столбцы выбираются один раз, какие-то - два раза, другие - ни разу), далее для каждой из бутстреп-реплик строится дерево.
При проведении бутстрепа каждой ветви присваивается значение - поддержка - процент случаев присутствия ветви в 100 различных деревьях. Чем больше поддержка (ближе к 100), тем достовернее и надёжнее разветвление в данном узле.

Бутстреп выводит следующее: одна ветвь, имеющая значение 100, является достоверной, что, действительно, так и есть, если смотреть на референсное дерево (смотри Рис. 2.); 2 ветви тоже с высокими значениями (81 и 71), по мнению бутстрепа, также являются верными, что правда, судя по референсному дереву. Ветви со значениями 58 и 53 показаны как сомнительные ветви, хотя это странно, потому что на референсном дереве такие ветви существуют. Значения 38 и 26 у ветвей показывают, что данные ветви вряд ли достоверны, и опять же это верно, потому что на референсном дереве данных ветвей нет. Наверное, отдельно стоит обсудить ветвь по значением 54, не удивительно, что бутстреп показывает такое пограничное значение достоверности данной ветви, так как на референсном дереве Vulpes lagopus (VULLA) и Panthera tigris altaica (PANTA) находятся в одной кладе ещё вместе с Pusa hispida (PUSHI).