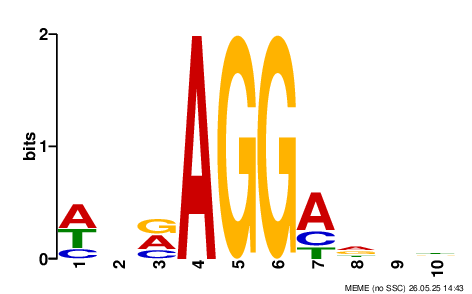
В данном практикуме будет произведён поиск последовательность Shine-Dalgarno (сайта посадки рибосомы на мРНК) в геноме бактерии Pseudomonas syringae, по которой ранее мной был написан мини-обзор.
Для работы понадобились следующие файлы, скачанные с NCBI: геном рассматриваемой бактерий (в формате fasta) и аннотацию этого генома (в формате gff).
На основе взятых файлов и скрипта, написанного Вячеславом Масленниковым, были созданы 3 файла:
На основе TRAIN.fasta с помощью MEME был проведён поиск мотива, длина которого составляет от 6 до 10 нуклеотидов, следующей командой:
meme TRAIN.fasta -dna -nmotifs 1 -minw 6 -maxw 10
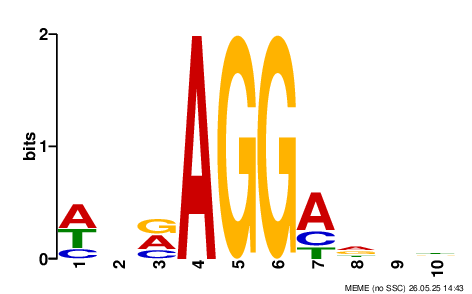
Полную выдачу можно посмотреть здесь.
Находка является значимой, так как E-value низкий (6.5e-008). Паттерн полученного мотива: WNVAGGADNK, его консенсус: ANGAGGAANT. Начало полученного мотива схоже с последовательностью Shine-Dalgarno AGGAGG.
Провела поиск найденного с помощью MEME мотива для двух групп контроля (POSITIVE.fasta и NEGATIVE.fasta) с помощью команд:
fimo --oc fimo_pos -thresh 0.001 ./meme_out/meme.txt POSITIVE.fasta
Результат работы можно посмотреть здесь.
fimo --oc fimo_neg -thresh 0.001 ./meme_out/meme.txt NEGATIVE.fasta
Результат работы можно посмотреть здесь.
Среди последовательностей из POSITIVE.fasta 725 являются значими находками из всех 5185 (приблизительно 14% от всех последовательностей), а среди последовательностей из NEGATIVE.fasta 132 являются значими находками из 5185 (2,5% от всех последовательностей). На мой взгляд, нельзя заявить, что найденный мотив является последовательностью Shine-Dalgarno для Pseudomonas syringae, потому что мало, где встречается, судя по количеству находок (даже не половина от всех последовательностей). Наверное, изначально стоило делать последовательности размером не 25 нуклеотидов перед старт-кодоном, а больше.