1. Кратко опишите один сигнал, закодированный в геноме
Вирусы не способны независимо реплицировать геном, поэтому они захватывают механизмы клетки-хозяина,
чтобы размножаться. Пикорнавирусы получают необходимые условия для эффективного синтеза белка с помощью
специфических последовательностей, известных как сайты внутренней посадки рибосомы (Internal Ribosome Entry Site - IRESs). Они либо стимулируют, либо подавляют
трансляцию и репликацию пикорнавируса. Некоторые IRESs неэффективны и требуют применения
ITAFs. Существует два типа ITAF, один способствует вирусной трансляции, зависящей от IRES, а второй тип ограничивает.
Пикорнавирусы IRESs подразделяются на пять типов в зависимости от используемой ими последовательности, ITAFs и факторов инициации.
На самом деле оно встречается и у эукариот. Собственно, как мы уже сказали, он нужен для трансляции. Собственно
это регуляторный участнок мРНК в 5'UTR, но также может встречаться в другом месте мРНК.
IRES связывается с 40S субъединицей рибосомы. После связывания он превликает фактор инициации трансляции, независимым от кэпа образом.
2. Для одного из сигналов в геноме найдите последовательности представителей, постройте PWM и оцените результаты поиска по этой PWM новых сайтов.
Я выбрала последовательность Шайна-Дальгарно, расположенную перед стартом трансляции генов в геноме бактерии Micrococcus luteus.
Я взяла референсный геном ASM1989091v1. Для выполения задания
был использован скрипт, любезно предоставленный Лизой Плешко.
На вход вход скрипт берет три файла: обучающую выборку, тестовую и
для отрицательного контроля.
Для начала были взяты последовательности, которые начинаются за 50 нуклеотидов до генов и заканчиваются старт кодоном.
Из них мы выделили 200 случайных и выровняли их в JalView с помощью программы Muscle. Выделили участок выравнивания (без гэпов)
с 41 по 57 нуклеотиды и получили последовательности сигнала в формате fasta. Получилась обучающаяя выборка - на ее основе
была создана матрица PWM.

Далее из тех же обрезанных кусочков (50 нуклеотидов до старт кодона) взяли 100 других - сформировали тестовую выборку.
А для отрицательного контроля мы у тех же генов (100 шт), что были в тестовой выборке, взяла 100 их первых нуклеотидов.
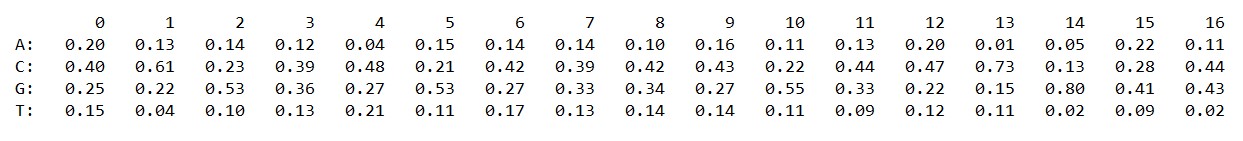
Выдача скрипта:
Консенсусная последоваетльность: CCGCCGCCCCGCCCGGC
Порог: 4.097
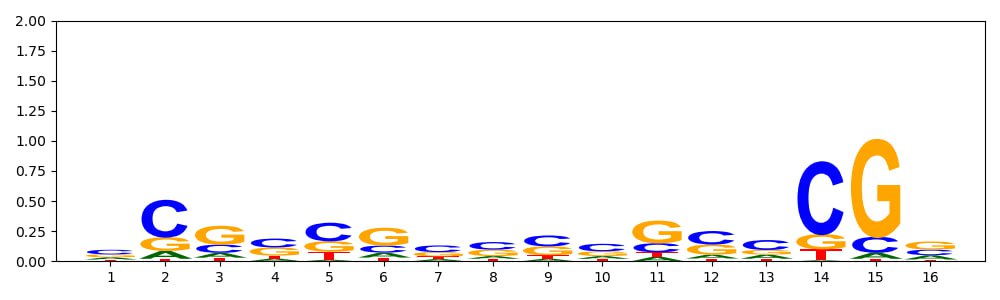
С помощью скрипта я построила LOGO-диаграмму для
этого сигнала. Результат, конечно, не порадовал. Все буквы практически одинакового размера, да еще и низкие.
Это значит, что информационное содержание у полученной матрицы достаточно мало и под нашу матрицу подходит много что,
даже то, что не является последовательностью ШД. Однако позиции 2, 14 и 15 достаточно специфичны относистельно остальных.
Порог веса равен 4.097. Вероятность случайно сгенерировать последовательность (с учетом встречаемости нуклеотидов)
с таким или большим весом равна 0.01. Далее мы посчитали веса последоавтельностей в каждой выборке и построили график их распределения.
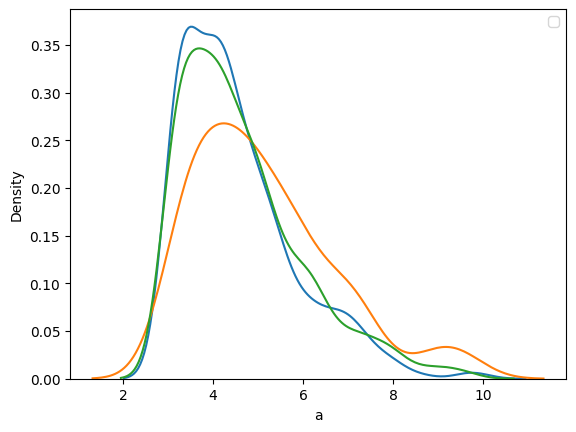
Так как pwm строилась на основе обучающей выборки - среднее значение веса послеовательностей в ней должна быть выше,
чем у тестовой и отрицательного контроля. Так и есть.
А вот у тестовой выборки иотрицательного контроля средние значения очень близки, из чего мы делаем вывод, что наша модель не
специфична: даже в тех местах, где ШД быть не может, определяет ее.
Большое спасибо за прочтение моего практикума! Хорошего Вам настроения!