A- и В- формы ДНК. Структура РНК
1.Построение модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
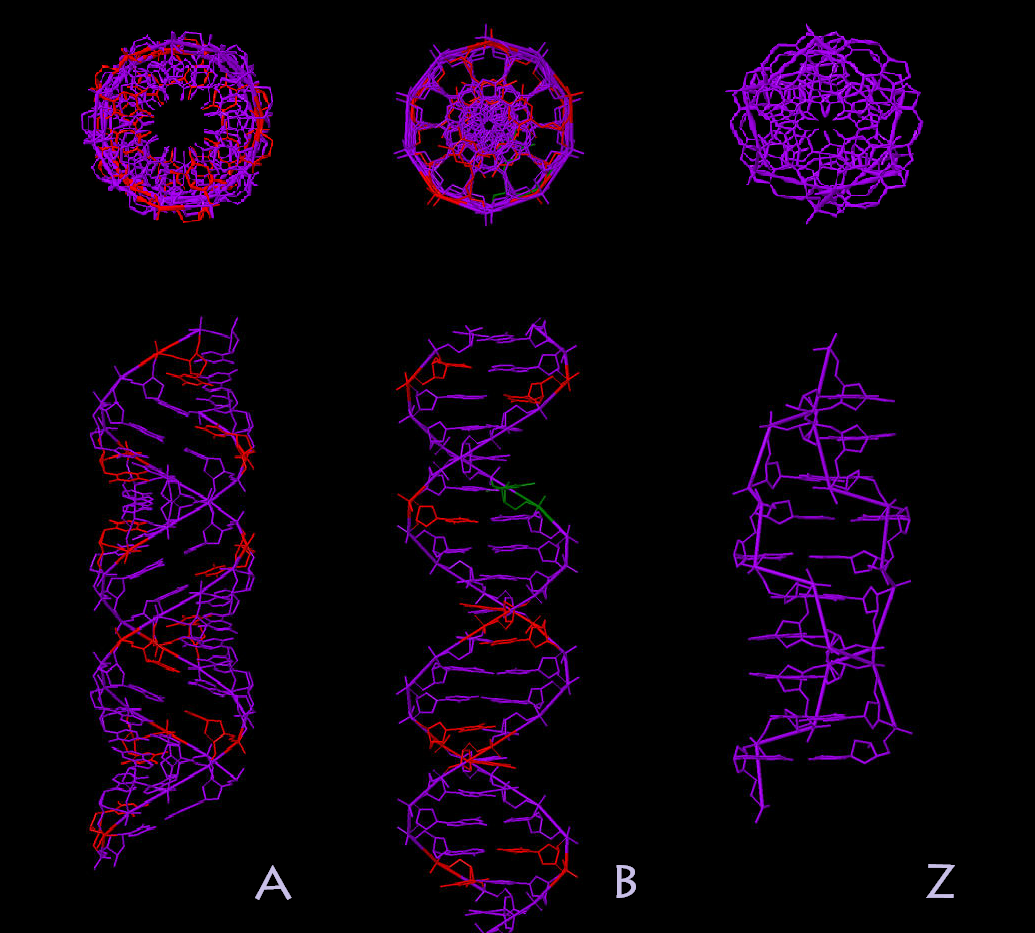
С помощью программы fiber пакета 3DNA были построены A-, B- и Z-формы дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "GATC".
fiber -a gatc_A.pdb fiber -b gatc_B.pdb fiber -z gatc_Z.pdb Полученные файлы: gatc_A.pdb gatc_B.pdb gatc_Z.pdb
A-,B-,Z- формы дуплекса ДНК
Пространственная организация ДНК при стандартных физиологических условиях представлена В-формой. A-форма образуется в средах с низким содержанием воды. А-ДНК представляет собой правозакрученную двойную спираль, на один оборот которой приходится 11 пар оснований. Плоскость пар оснований в А-ДНК находится под углом приблизительно 20° к оси спирали. В результате этих структурных изменений углубляется большая бороздка, в то время как малая бороздка становится почти незаметной. Реагенты, используемые при кристаллизации ДНК, как правило, дегидрируют ее, поэтому большинство коротких молекул ДНК кристаллизуются именно в А-форме.
Z-форма ДНК очень сильно отличается от В-структуры: самое главное отличие - это левозакрученная спираль. На один ее оборот приходится 12 пар оснований, сама молекула более узкая и удлиненная. Остов ДНК принимает зигзагообразную форму. Примеры последовательностей, образующих левозакрученную спираль - это чредующиеся пиримидиновые и пуриновые основания, особенно чредующиеся остатки С и G или 5-метил-С и G. Большая бороздка в Z-ДНК едва заметна а маленкая - узкая и глубокая.
По некоторым данным, короткие фрагменты Z-ДНК могут быть и у прокариот и у эукариот. Эти фрагменты могут играть роль в регуляции экспрессии некоторых генов или в генетической рекомбинации.
2.Сравнение структур 3-х форм ДНК с помощью средств JMol.
Упражнение 1.
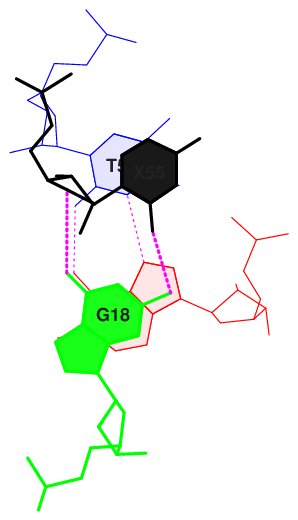
Для примера был взят тимин15.
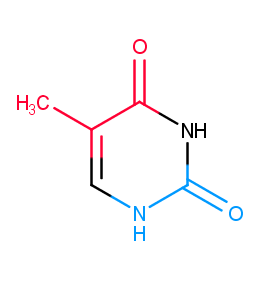

Упражнение 2.
С помощью программы Jmol измерим шаг спирали и длинны малой и большой бороздок для каждой из трех форм полученной последовательности ДНК.
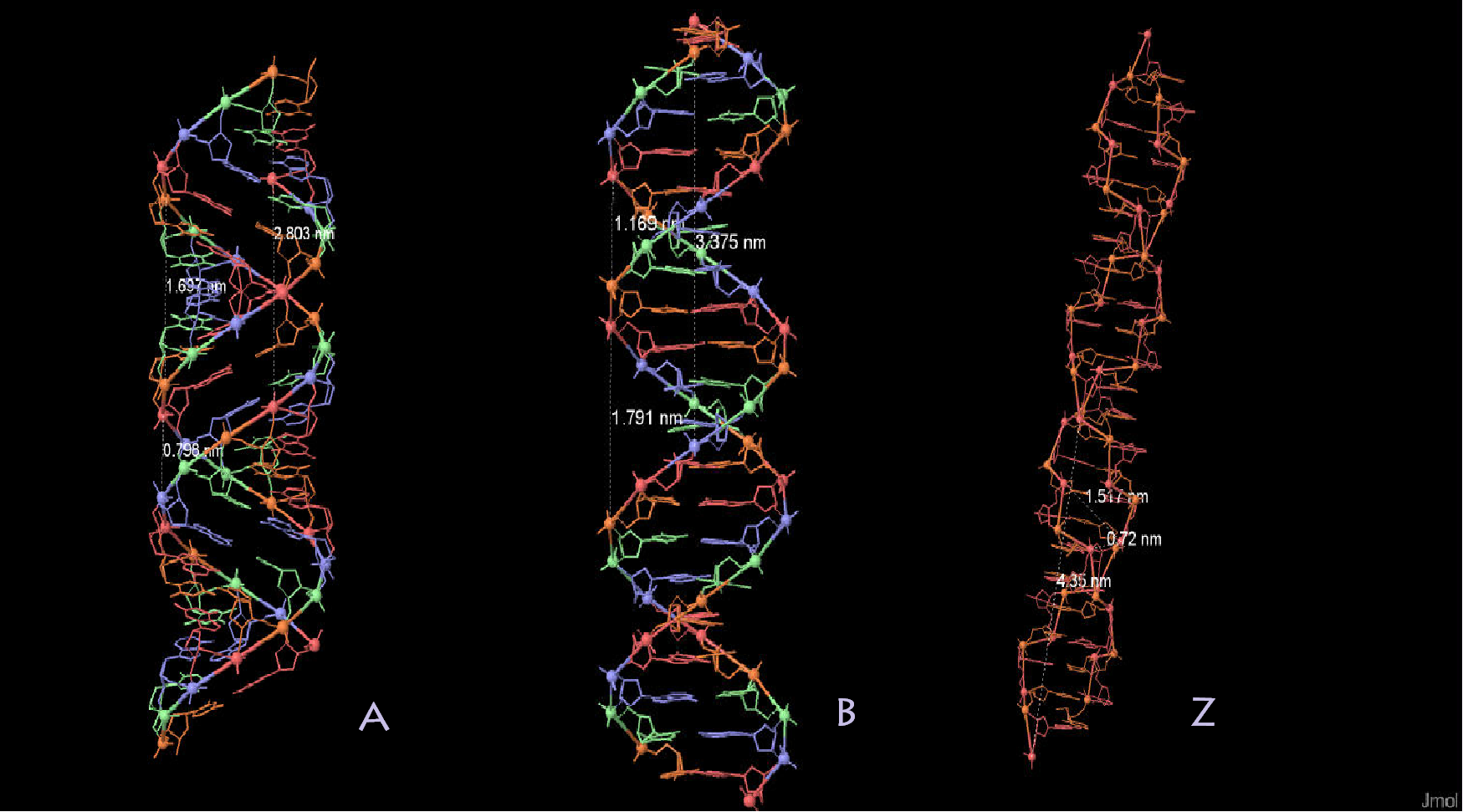
16,97 Å ( [C]36: B.P – [C]8: A.P)
17,91 Å ( [G]9: A.P – [C]28: B.P)
15,17 Å ( [G]33: B.P – [C]16: A.P)
7,2Å ( [G]19: A.P – [G]33: B.P)
Упражнение 3.
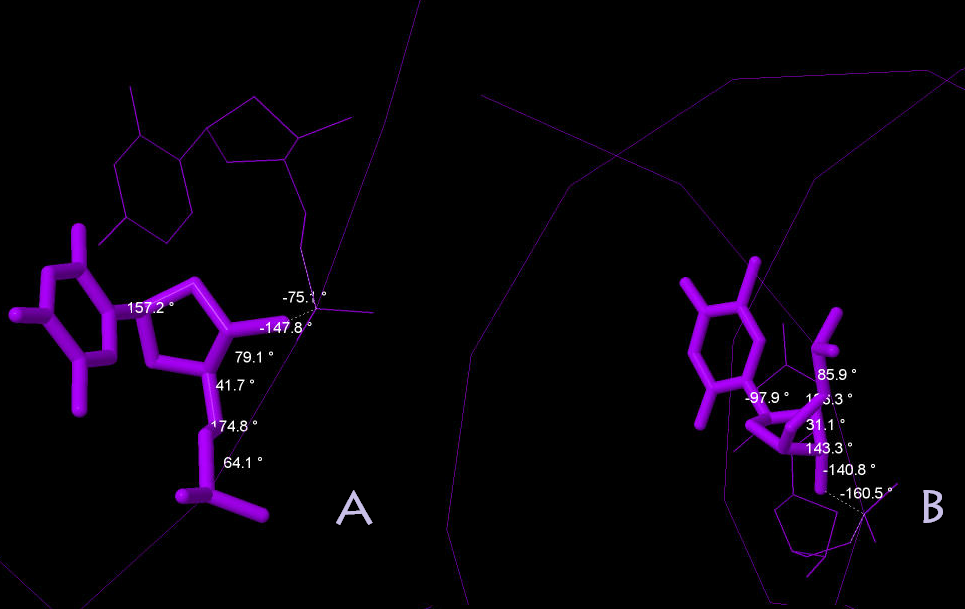
В программе Jmol измерим торсионные углы в тимине 3 для А-формы и В-формы ДНК.
α ( P-O'5) |
β (P-C4') |
γ (O'5-C3') |
δ (C'5-O3') |
ε (C'4-P) |
ζ (C3'-O5') |
χ (O4'-C2) |
|
A форма |
64.1 |
174.8 |
41.7 |
79.1 |
-147.8 |
-75.1 |
-157.2 |
А форма (презентация) |
62 |
173 |
52 |
88.3 |
178 |
-50 |
-160 |
В форма |
85.9 |
136.3 |
31 |
143.3 |
-140.8 |
-160.5 |
-97.9 |
В форма (презентация) |
63 |
171 |
54 |
123/131 |
155 |
-90 |
-117 |
Некоторые значения, приведенные для торсионных углов созданных структур, не коррелируют со значениями, представленными в презентации.
3.Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
На сайте PDB выполним поиск по идентификаторам 1CF7 и 1EVV, которые понадобятся нам для дальнейшей работы. В связи с тем, что пакет 3DNA работает только со старым форматом PDB, конвертируем файлы следующей командой: remediator --old ''1CF7_dna.pdb'' > ''1CF7_dna_old.pdb'' remediator --old ''1EVV_rna.pdb'' > ''1EVV_rna_old.pdb'' После вышеуказанных манипуляций приступим к анализу полученных данных. Упражнение 1.Для каждого gatc_A.pdb, gatc_B.pdb, gatc_Z.pdb выполним команды типа:
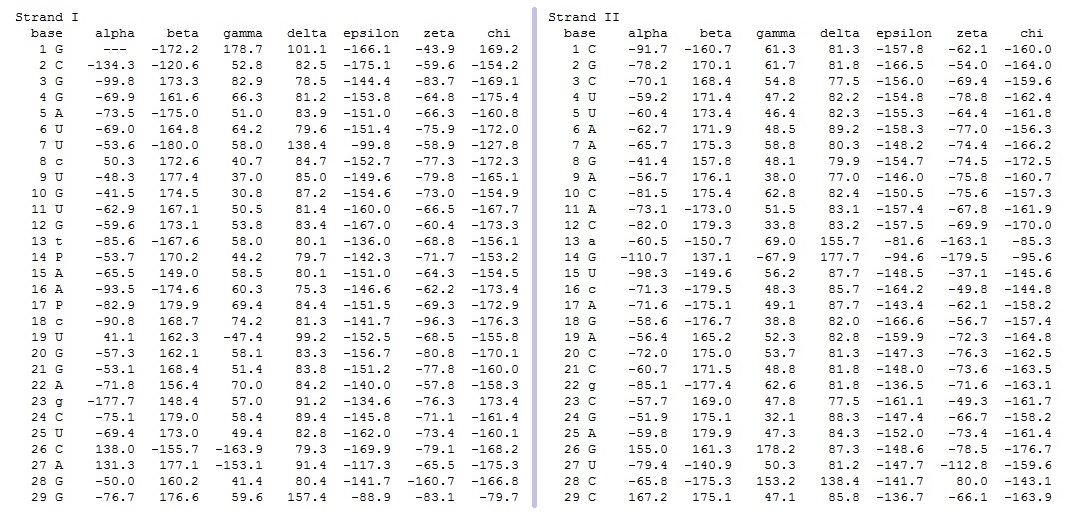
find_pair -t gatc_x.pdb stdout | analyze В результате чего мы получим файлы с подробным описанием параметров этих структур. Найдем в файлах gatc_A.out,gatc_B.out и gatc_Z.out значения торсионных углов. Если сравнить значения соответствующих торсионных углов в структурах A-, B- и Z-форм ДНК, то в наибольшей степени различаются значения следующих углов: у A- и B-форм - δ и χ; у A- и Z-форм - α; у A- и Z-форм - α, ξ и χ. Теперь применим команды find_pair и analyze к полученным тРНК и ДНК. Из данных о торсионных углах, представленных в файле 1EVV_rna_old.out, можно сказать, что по параметрам тРНК более всего похожа на А-форму ДНК.
Торсионные углы тРНКС помощью Excel определим среднее значение каждого из торсионных углов ДНК (не рассматривая краевые нуклеотиды). Из полученного файла Torsion.ods определиv номер самого "деформированного" нуклеотида, коим является аденин 14 из цепи 1. Деформированность можно определить как сумму разниц значения каждого угла и среднего значения по данному углу. Чем больше сумма, тем более деформирован остаток.
Упражнение 2.
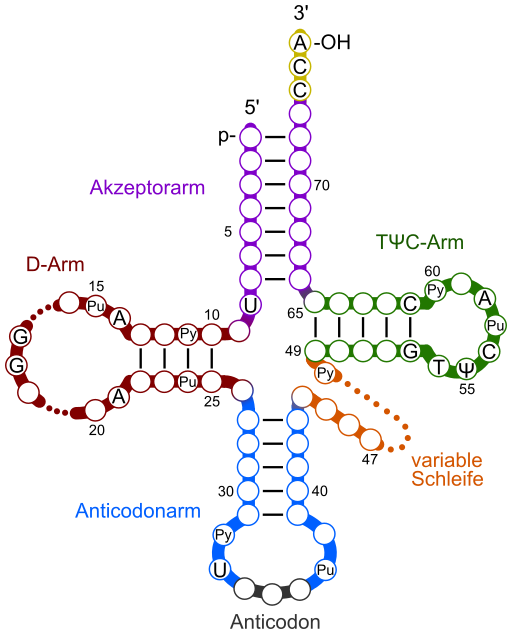
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями в нашей тРНК. Ниже представленна структура тРНК в общем виде и таблица с номерами нуклеотидов, образующих водородные связи (помечено *). Водородные связи, не относящиеся к стеблям, могут играть роль стабилизаторов в третичной структуре тРНК. Фиолетовым цветом выделен акцепторный стебль; зеленым - Т-стебль; красным - D-стебль и синим - антикодоновый стебль.Из полученных данных можно сделать следующие заключения:
- Акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72.
- Т-стебель состоит из участка 49-53 и комплементарного ему участка 61-65.
- D-стебель сосотит из участка 10-13 и комплементарного ему участка 22-25.
- Антикодоновый стебель состоит из участка 39-42 и комплементарного ему участка 28-31.
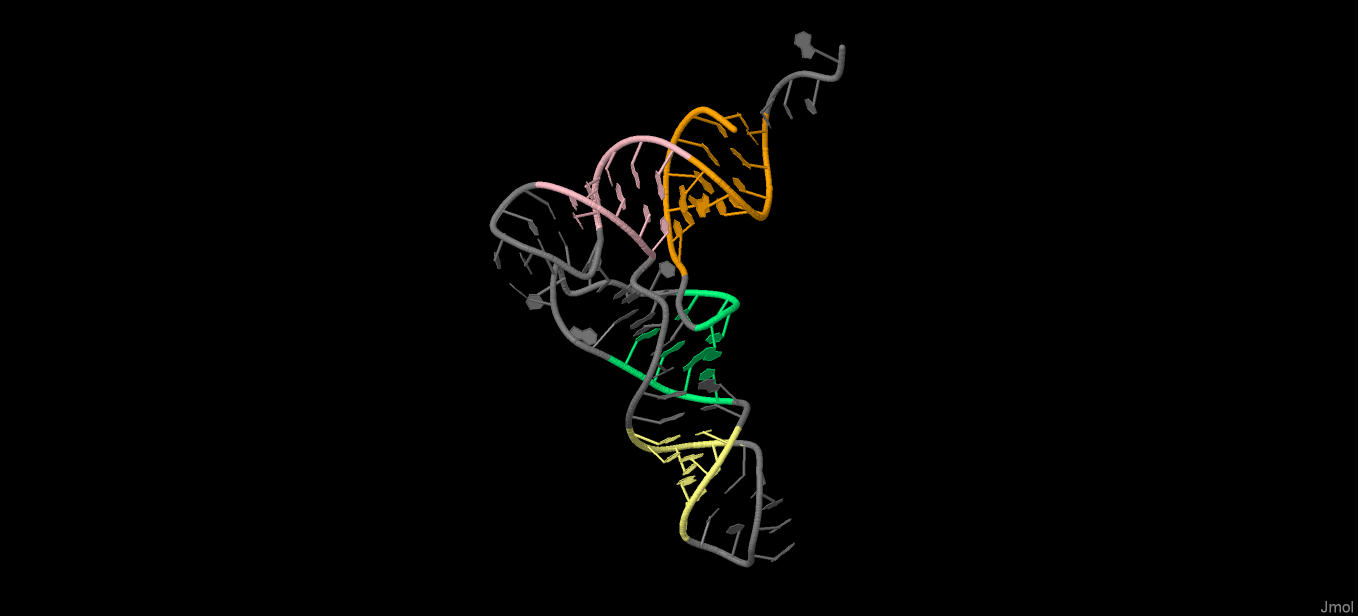
С помощью программы Jmol проиллюстрирован остов заданной тРНК, где оранжевым цветом выделен акцепторный стебель, зеленым — D-стебель, розовым — T-стебель и желтым — антикодоновый стебель.
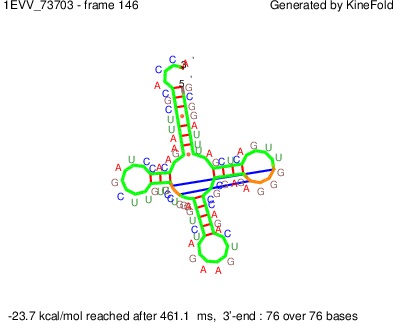
Зная последовательность тРНК, для сравнения мы также воспользовались программой KINEfold, которая осуществляет стохастическую симуляцию процесса фолдинга. Как видно, полученное изображение далеко от реальной структуры.
Возвращаясь к вторичной структуре заданной тРНК важно отметить:
- Структуру стеблевых дуплексов формируют 18 канонических пар оснований и 2 неканонические пары (G4-U69; P39-A31).
- Дигидроуридины присутствуют в D-петле.
- Тимидины отсутствуют в Т-петле.
- Вариабельная петля представлена 4-мя нуклеотидами: U47, 7MG46, G45 и A44.
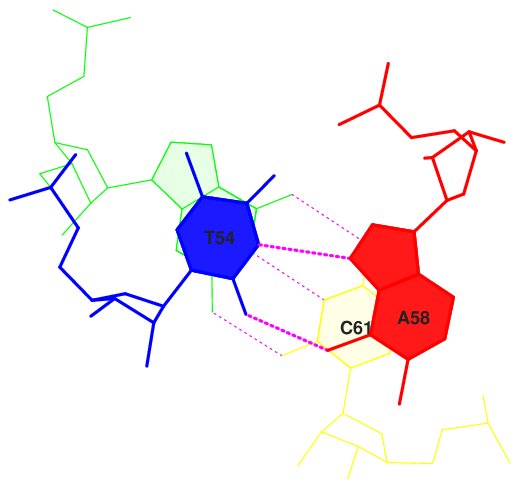
- В заданной тРНК есть несколько дополнительных водородных связей, стабилизирующих ее третичную структуру. Такие связи составляют 4 канонические пары (5MU54-1MA58; A36-U33; A14-U8; G15-C48) и 3 неканонические (PSU55-G18; A38-OMC32; A44-M2G26).
Упражнение 3.
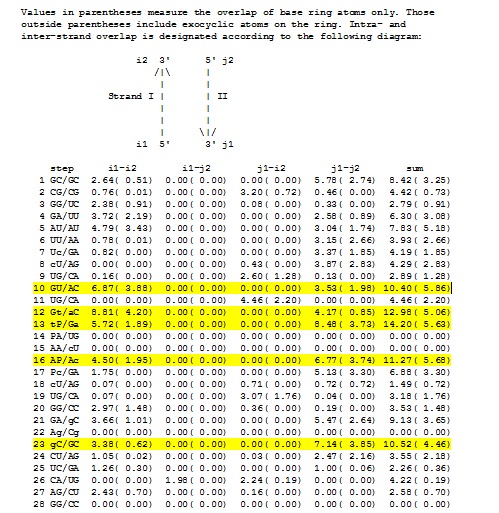
Вданном упражнении научимся находить возможные стэкинг-взаимодействия (связь между параллельными плоскостями оснований) в нашей тРНК. Для этого сначала исследуем участки перекрываний, информация о которых содержится в файле 1EVV_rna_old.out. Как видно из таблицы, для нашей тРНК существует около 5 участков с наибольшим перекрыванием и 4 с наименьшим.
Рассмотрим только 2 участка с наибольшим перекрывание (Section #0012 - Section #13). Для того, чтобы определить координаты искомых нуклеотидных остатков, между которыми предполагается стекинг-взаимодействие, воспользуемся командой типа:
ex_str -12 stacking.pdb step12.pdb ex_str -13 stacking.pdb step13.pdb Чтобы получить изображения воспользуемся следующими командами: stack2img -cdolt step12.pdb step12.ps stack2img -cdolt step12.pdb step12.ps