Обзор генома и протеома бактерии Streptomyces lincolnensis
РЕЗЮМЕ
Данная работа представляет собой мини-обзор генома и протеома бактерии Streptomyces lincolnensis. Интерес данного исследования заключается в возможности использования данного микроорганизма при производстве антибиотиков.
КЛЮЧЕВЫЕ СЛОВА: Streptomyces lincolnensis; геном; протеом; линкомицин.
1 ВВЕДЕНИЕ
Целью данного обзора является изучение генома и протеома Streptomyces lincolnensis.
Таксономия вида Streptomyces lincolnensis[1]:
Домен:
Тип:
Класс:
Порядок:
Семейство:
Род:
Вид:
Bacteria
"Actinobacteria"
Actinobacteria
Streptomycetales
Streptomycetaceae
Streptomyces
S. lincolnensis
Данная бактерия является анаэробной, грамположительной, филаментной (нитчатой). Образует хорошо развитый воздушный мицелий, позволяющий прикрепляться к субстрату и добывать органические вещества.[2] Обитает в почвах. Была впервые выделена в 1963 году.[3]
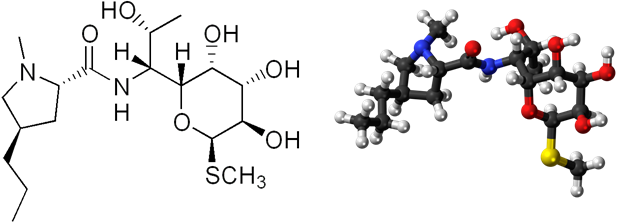
Род Streptomyces дал название антибиотику стрептомицин.[2] Все представители данного рода вырабатывают те или иные антибиотики, причём количество различных биологически активных веществ, производимых представителями рода, доходит до 8000.[4] В частности S. lincolnensis продуцирует линкомицин (Рис. 1) — антибактериальный антибиотик группы линкозамидов.[1]
Представители рода хорошо культивируются в питательной среде с pH~7.2 при комнатной температуре. Оптимальной для роста температурой является 28℃.[5, 6] Поэтому S. lincolnensis представляет большой интерес в биотехнологии и фармацевтическом производстве.
2 МЕТОДЫ
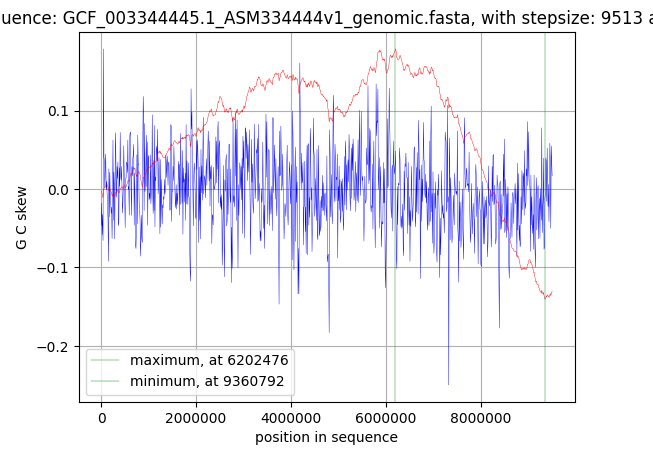
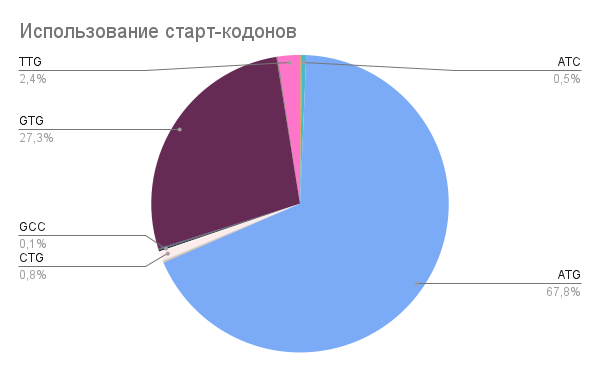
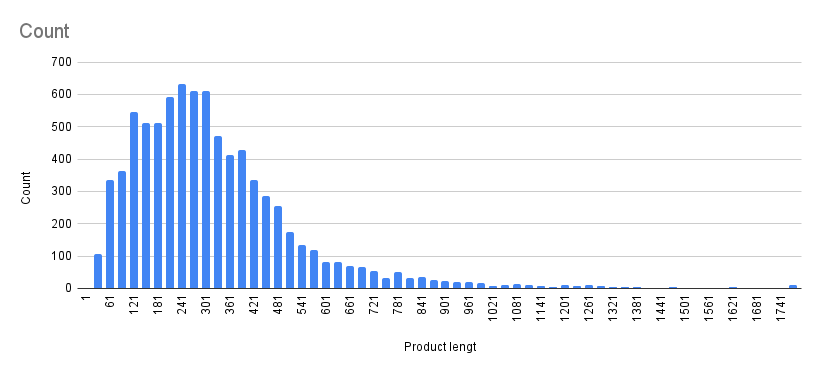
Все материалы, а именно файл с геномом бактерии, таблица его особенностей и файл с кодирующими последовательностями всех генов белков, были взяты из базы данных GenBank.[8] Для определения нуклеотидного состава генома (Таблица 1) и частоты использования кодонов и распределения генов по цепям ДНК использовался скрипт, написанный на Python. Для построения кумулятивного графика GC skew (Рис. 2) использован сервис Webskew.[9] Для построения диаграммы частоты использования старт-кодонов (Рис. 3), гистограммы распределения длин белков (Рис. 4), Таблицы 4 и Таблицы 5 было использовано приложение Google Таблицы.
3 РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЯ
Геном представлен одной кольцевой хромосомой, состоящей из 9 513 637 пар нуклеотидов.[10] В Таблице 1 приведено количество встреченных нуклеотидов и частота встреч. Причём количество нуклеотидов A приблизительно равно количеству нуклеотидов T, а количество С – количеству G, что означает, что второе правило Чаргаффа соблюдается. Также был определён GC-состав ДНК. Он равен 0.7106. Такой довольно высокий уровень содержания GC в молекуле ДНК свойственен бактериям типа Actinobacteria.[11]
| Нуклеотид | Количество | Частота |
|---|---|---|
| A | 1 378 642 | 0.1449 |
| C | 3 388 854 | 0.3562 |
| G | 3 371 840 | 0.3544 |
| T | 1 374 301 | 0.1445 |
На Рис. 2 представлен график GC-skew cumulative. Расчёт GC-skew в окне заданной ширины производится по формуле:
\begin{equation}GCskew = \frac{G-C}{G+C}\tag1\end{equation}где G и С – количество соответствующих нуклеотидов в окне. GC-skew cumulative позиции считается как сумма всех GC-skew, посчитанных ранее. Точка минимума на графике соответствует началу репликации – oriC. Её координата в районе 9 360 792 нуклеотида. Максимум на графике должен соответствовать точке терминации репликации – ter.
На графике максимум наблюдается в районе 6 202 479 нуклеотида. Однако точка ter ожидается на участке кольцевой ДНК, диаметрально противоположном oriC, т.е. верна формула:
\begin{equation}|oriC-ter|\approx\frac{SecquenceLength}{2}\tag2\end{equation}где SequenceLength – длина кольцевой ДНК. Тогда точка ter должна находится в районе 4 600 000 нуклеотида. Неверное определение точки ter, вероятно, вызвано наличием большого числа локальных минимумов и максимумов на графике GC-skew comulative.
Наиболее часто используемым стоп кодоном является TGA (Таблица 2). Таким образом подтверждается корреляция между GC-составом генома и частотами использования стоп-кодонов.[14] Также встречаются и нестандартные стоп-кодоны, среди них CGC, GCC, GTC наиболее часто, однако всё равно более чем в 40 раз реже, чем TAA. На Рис. 3 представлена круговая диаграмма частоты использования старт-кодонов. Старт-кодом чаще всего является ATG, однако также довольно часто встречаются старт-кодоны, получающиеся из ATG заменой одного нуклеотида. Вероятно подобные мутации незначительны, и не препятствуют инициации синтеза белка.
| Стоп-кодон | Количество | Частота |
|---|---|---|
| TAA | 439 | 0.0517 |
| TAG | 1 521 | 0.1791 |
| TGA | 6 401 | 0.7536 |
Распределение генов по цепям ДНК приведено в Таблице 3. Для генов белков вероятность получить такое же или большее различие равна ~0,00008, что является статистически значимым. Похожий результат наблюдается, если рассмотреть распределение по половинам цепей ДНК.
| Статистическая величина | Значение |
|---|---|
| Средняя длина | 336 |
| Стандартное отклонение | 223 |
| Медиана | 294 |
| Минимальная длина | 18 |
| Максимальная длина | 3 638 |
| Тип гена | Прямая цепь | Обратная цепь |
|---|---|---|
| Ген белка | 4 270 | 3 913 |
| Псевдоген | 115 | 108 |
| Ген РНК | 45 | 45 |
| Длина | Название белка |
|---|---|
| 3 638 | non-ribosomal peptide synthetase |
| 3 062 | non-ribosomal peptide synthetase |
| 2 696 | type I polyketide synthase |
| 2 577 | non-ribosomal peptide synthetase |
| 2 540 | non-ribosomal peptide synthetase |
| 2 525 | hybrid non-ribosomal peptide synthetase/ type I polyketide synthase |
На Рис. 4 представлена гистограмма распределения длин белков с шириной кармана равной 30 аминокислотным остаткам (а.о.). Наибольшее количество белков имеют длину около 241 – 270 а.о., второй пик соответствует длине 121 – 150 а.о. Также примечательно, что в протеоме имеется небольшое число очень больших белков, длина которых превышает среднюю в 8 – 10 раз (Таблица 5). Эти белки принимают участие в синтезе вторичных метаболитов.[12, 13] Не исключено, что именно эти белки могут участвовать в синтезе линкомицина.
ЗАКЛЮЧЕНИЕ
Изучение протеома и генома является одними из основных факторов при поиске и разработке лекарственных препаратов и методов лечения. В ходе данного мини-обзора были определены такие интересные особенности Streptomyces lincolnensis, как скачкообразно изменяющийся вдоль генома кумулятивный GC-skew, разнообразие в использовании старт-кодонов и стоп-кодонов и состав протеома.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Все сопроводительные материалы, в частности скрипт, написанный на Phython, и использованная в работе электронная таблица доступны по ссылкам: Открыть папку | скачать одним архивом
- Файл с геном бактерии открыть | скачать .fna
- Таблица особенностей генома бактерии открыть | скачать .txt
- Файл с кодирующими последовательностями открыть | скачать .fasta
- Скрипт, написанный на Python открыть | скачать .py
- Файлы выдачи скрипта:
- Электронная таблица открыть | скачать .xlsx
СПИСОК ЛИТЕРАТУРЫ
- 1🠕 2🠕 Wikipedia, Streptomyces lincolnensis
- 1🠕 2🠕 Wikipedia, Streptomyces
- 1🠕 Mason, D.J., A. Dietz and C. DeBoer. 1963. Antimicrobial Agents and Chemotherapy, 1962, pages 554 – 559.
- 1🠕 János Bérdy, Bioactive Microbial Metabolites
- 1🠕 The Bacterial Diversity Metadatabase BacDive, Streptomyces lincolnensis DSM 40355
- 1🠕 BacMedia, GYM STREPTOMYCES MEDIUM
- 1🠕 Wikipedia, Lincomycin
- 1🠕 GenBank, Index of /genomes/all/GCF/003/344/445/GCF_003344445.1_ASM334444v1
- 1🠕 Webskew
- 1🠕 Статистика сборки GCF_003344445.1_ASM334444v1б
- 1🠕 Lightfield J., Fram Noah R., Ely B. 2011. Across Bacterial Phyla, Distantly-Related Genomes with Similar Genomic GC Content Have Similar Patterns of Amino Acid Usage
- 1🠕 Wikipedia, Nonribosomal peptide
- 1🠕 Wikipedia, Polyketide synthase
- 1🠕 Ho, A.T. and Hurst L.D. 2021. Variation in Release Factor Abundance Is Not Needed to Explain Trends in Bacterial Stop Codon Usage