Сигналы и мотивы
Сигналы программируемого -1 сдвига рибосомальной рамки считывания (−1 PRF)
Программируемый сдвиг рамки считывания представляет собой явление, когда при трансляции рибосома сдвигается на +1 или -1 нуклеотид, что приводит к трансляции альтернативного белка с той же мРНК. Данное явление распространено среди вирусов, однако подобные мотивы также обнаружены у дрожжей (Saccharomyces cerevisiae). Сигналом является мотив N NNW WWH и специфическая вторичная структура мРНК (псевдоузел). [1]
Последовательность Козак в геноме человека
Последовательность Козак – это крестность ATG кодона, являющаяся сигналом старта транскрипции. Для выполнения данного практикума был использован скрипт, основанный на коде Смирнова Максима и Селифонова Игоря. Данный скрипт принимает на вход файл human-genes.tsv, содержащий таблицу со списком генов человека и их положением в геноме. В результате работы скрипта получаются следующие файлы:
- kozak-learn.fasta, kozak-test.fasta и pseudokozak1.fasta – файлы с последовательностями для обучения, тестирования и негативного контроля соответственно
- result.csv – позиционная весовая матрица с псевдоттсчетами (PWM), построенная на материале обучения
- ic.csv – матрица информационного содержания IC(b,j) для выравнивания из материала обучения
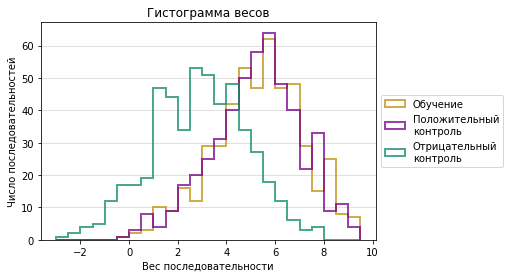
- hist.svg и hist.png – изображения гистограммы весов
- check.csv – таблица результатов проверки
В результате работы скрипта получено 100 последовательностей для обучения, 100 для тестирования и 100 последовательностей отрицательного контроля. На материале обучения построена следующая позиционная весовая матрица с псевдоттсчетами (PWM):
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.28 | -0.39 | -0.40 | -0.17 | 0.40 | 0.11 | -0.47 | 1.22 | -6.60 | -6.60 | -0.22 | -0.07 | -0.50 |
| T | -0.40 | -0.35 | -0.34 | -0.80 | -1.21 | -0.86 | -1.16 | -6.60 | 1.22 | -6.60 | -0.98 | -0.61 | -0.53 |
| G | 0.28 | 0.60 | 0.28 | 0.25 | 0.53 | -0.10 | 0.33 | -6.24 | -6.24 | 1.58 | 0.84 | -0.22 | 0.59 |
| C | 0.41 | 0.07 | 0.45 | 0.55 | -0.52 | 0.56 | 0.76 | -6.24 | -6.24 | -6.24 | -0.13 | 0.67 | 0.31 |
На основе данной таблицы были посчитаны веса всех последовательностей и построена гисторгамма распределения. Исходя из неё порог веса, выше которого находка считается правильной, был выбран равным 4 и составлена таблица результатов проверики.
Веса последовательностей из материала обучения и материала тестирования имеют сходные распределения, в то время как веса последовательностей негативного контроля заметно сдвинуты влево. Однако эти распеделения в значительной степени перекрываются, поэтому ошибки первого и второго рода при тестировании велики (30,4% и 23,6% соответственно). Причём уменьшее этих ошибок за счёт изменения порога не представляется возможным.
| Обучение | Положительный контроль | Отрицательный контроль | |
|---|---|---|---|
| Cигнал(+) | 389 (77.8%) | 382 (76.4%) | 152 (30.4%) |
| Cигнал(-) | 111 (22.2%) | 118 (23.6%) | 348 (69.6%) |
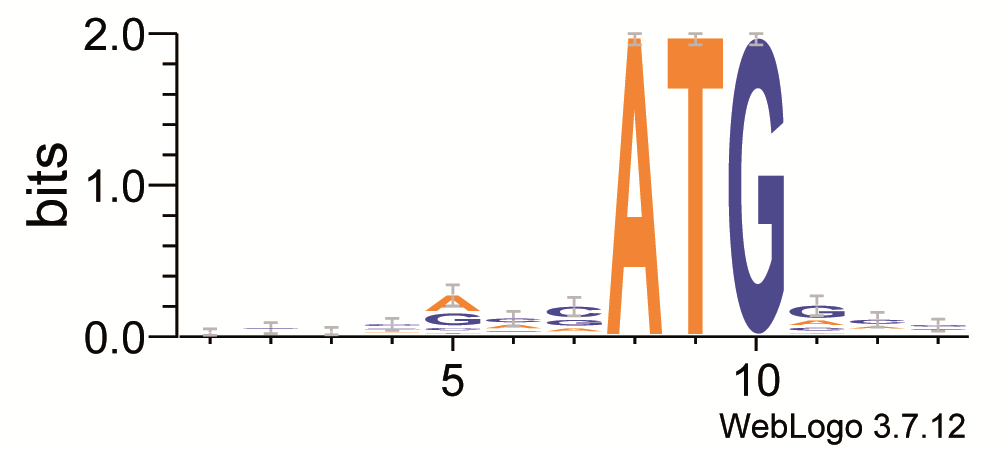
Далее была получена матрица информационного содержания и с использованием сервиса WebLOGO 3 построено LOGO.
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.09 | -0.11 | -0.11 | -0.06 | 0.26 | 0.05 | -0.13 | 1.76 | 0.00 | 0.00 | -0.08 | -0.03 | -0.13 |
| T | -0.11 | -0.10 | -0.10 | -0.15 | -0.15 | -0.16 | -0.15 | 0.00 | 1.76 | 0.00 | -0.16 | -0.14 | -0.13 |
| G | 0.11 | 0.32 | 0.11 | 0.10 | 0.27 | -0.03 | 0.14 | 0.00 | 0.00 | 2.29 | 0.57 | -0.05 | 0.31 |
| C | 0.18 | 0.02 | 0.21 | 0.28 | -0.09 | 0.29 | 0.48 | 0.00 | 0.00 | 0.00 | -0.03 | 0.39 | 0.13 |
| IC(j) | 0.09 | 0.13 | 0.10 | 0.16 | 0.28 | 0.16 | 0.34 | 1.76 | 1.76 | 2.29 | 0.31 | 0.17 | 0.17 |
Сайты GAATTC в геноме Escherichia coli
Для выполнения данного задания была использована референсная сборка генома Escherichia coli str. K-12 substr. MG1655 (GCF_000005845.2) и написан скрипт, в результате которого было найдено 646 сайтов GAATTC в геноме, при этом ожидаемое число таких сайтов в геноме равно 1097 (с учётом GC-состава). Данное отличие является статистически значимым, так как \(\begin{equation}\text{p-value} = 1.735\cdot10^{-49}\end{equation}\) (использовано биномиальное распределение).