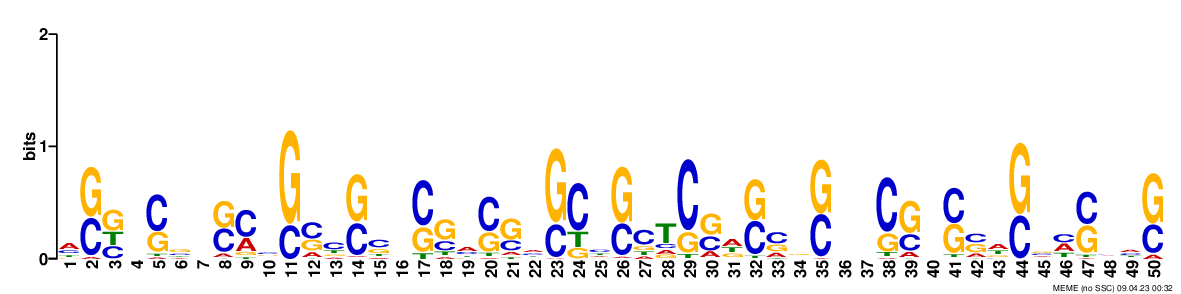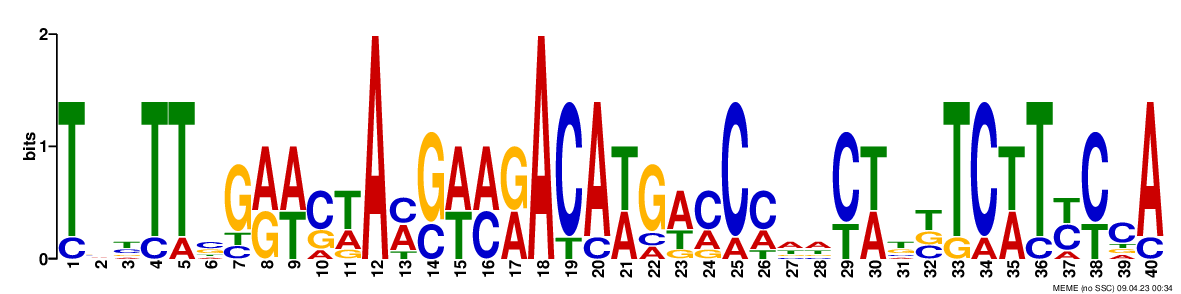Поиск сигнала посадки σ-субъединицы РНК-полимеразы
Подготовка данных
В рамках данного практикума проводился поиск сигналов в промоторной области генов бактерии Streptomyces lincolnensis. Для этого была использована референсная сборка генома GCF_003344445.1. На основе файлов с последовательностью (*.fasta) и аннотацией (*.gff) с использованием сервиса Operon-mapper был получен список оперонов. Промотором было решено считать 100 нуклеотидов перед опероном. В качестве материала для обучения было выбрано 50 случайных генов домашнего хозяйства, при этом поиск генов домашнего хозяйства проводился по ключевым словам в описании функции оперона. Для негативного контроля использованы случайные последовательности генома длиной 100 нуклеотидов. Все материалы получены в результате работы скрипта, который принимает на вход fasta-файл с последовательностью и список оперонов. В результате работы скрипта получаются следующие файлы: housekeeping.fasta, promotors.fasta и negative.fasta – файлы с последовательностями для обучения, тестирования и негативного контроля соответственно.
Запуск MEME
Для поиска сигналов в последовательностях был использован локальный MEME:
meme housekeeping.fasta -dna -mod zoops -nmotifs 3 -minw 6 -maxw 50 -maxsites 50
В результате работы MEME было найдено три мотива. Все три мотива длинные и странные, не вижу ничего похожего на SD. Зато у них низкие e-value (2.2·10-020, 6.9·10-006 и 4.1·10-004 соответственно). Один из них просто GC-богатый участок (сомневаюсь, что эта находка несёт какой-либо смысл). В дальнейшем решено работать с мотивом 2. Выдача программы в текстовом формате: meme.txt
Поиск сигнала в материале для тестирования с помощью FIMO
Для поиска второго найденного мотива в положительном и орицательном контроле были запущены следующие команды:
fimo --norc -motif TNYTTSGRWSWAMGWMRACAWGWMCMWWCWKBTCWTYCCA -thresh 0.001 meme.txt promotors.fasta
fimo --norc -motif TNYTTSGRWSWAMGWMRACAWGWMCMWWCWKBTCWTYCCA -thresh 0.001 meme.txt negative.fasta
В результате чего получены следующие результаты выдачи: promotors_fimo.tsv и negative_fimo.tsv. Было найдено 182 находки среди всех промоторов и 13 находок среди негативного контроля.