Домены и профили
Выбор домена и архитектуры для работы
В рамках данного практикума для поиска домена, удовлетворющего требованиям использованы Google Таблицы. В результате был выбран домен CD4-extracel (PF09191) со следующими характеристиками:
AC домена
Название домена
Seed
Full
UniProt
Средняя длина
Среднее сходство (%)
Средний процент покрытия белка доменом
Длина профиля HMM
Получение HMM-профиля
Для дальнейшей работы были скачаны fasta-файлы со всеми последовательностями, содержащими выбранный домен (Muravyov-full-171.fasta) и с последовательностями с выбранной доменной архитектурой (architectures.fasta). С использованием Jalview на основе второго файла получено выравнивание, из которого были удалены последовательности совпадающие более чем на 90%, а также участки до начала домена CD4-extracel и после конца домена Tcell_CD4_C. Полученное выравнивание содержит 19 последовательностей. Для получения HMM-профил был использован пакет HMMER, а именно последовательно были выполнены следующие команды:
hmm2build HMM aligment.fasta hmm2calibrate HMM hmm2search --cpu=1 HMM.xxx Muravyov-full-171.fasta > search.txt
В результате был получен HMM-профиль двухдоменной архитектуры, именющий длину 264 а.о. и файл, содержащий 140 находок, на основе которых с помощью скрипта получена таблица, содержащая сведения о всех последовательностях содержащих домен CD4-extracel.
| Название | Входит в семейство |
Входит в выборку для построения профиля |
Входит в список находок |
Вес находки | E‑value |
|---|---|---|---|---|---|
| A0A212CD61_CEREH | 0 | 0 | 1 | \(\begin{equation}-116,9\end{equation}\) | \(\begin{equation}0,15\end{equation}\) |
| A0A7E6D1T7_9CHIR | 0 | 0 | 1 | \(\begin{equation}-109,2\end{equation}\) | \(\begin{equation}0,054\end{equation}\) |
| A0A091UPT0_NIPNI | 1 | 1 | 1 | \(\begin{equation}600,2\end{equation}\) | \(\begin{equation}3,6\cdot10^{-179}\end{equation}\) |
| A0A1U8DZG6_ALLSI | 0 | 0 | 1 | \(\begin{equation}104,6\end{equation}\) | \(\begin{equation}5,7\cdot10^{-30}\end{equation}\) |
| F1SLT4_PIG | 0 | 0 | 0 | None | None |
Анализ HMM-профиля
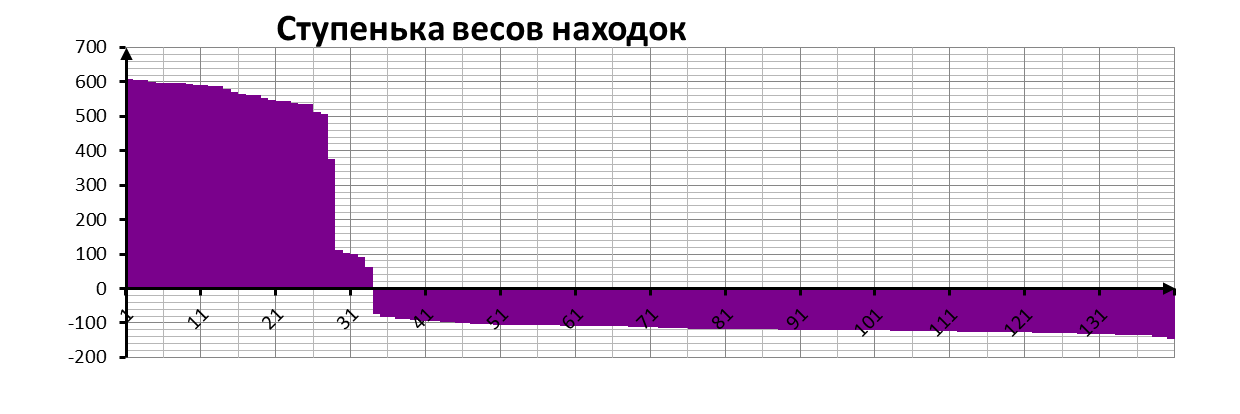
В результате работы всё того же скрипта получена таблица, содержащая сведения о находках. С использованием Excel получены графики ROC, F1, а также распределение весов. Скачать таблицу.
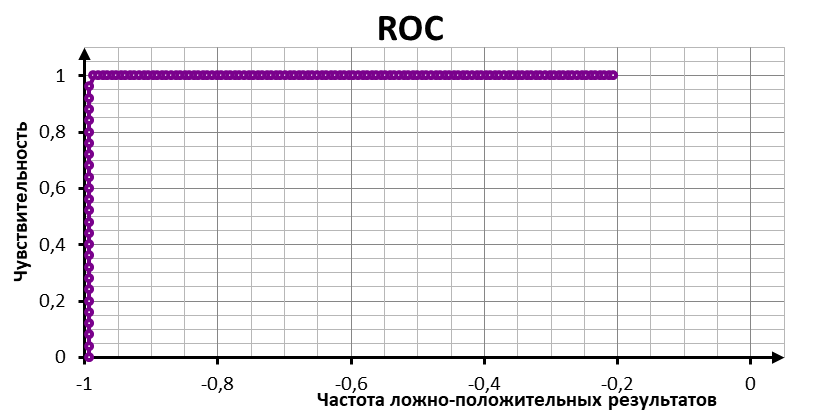
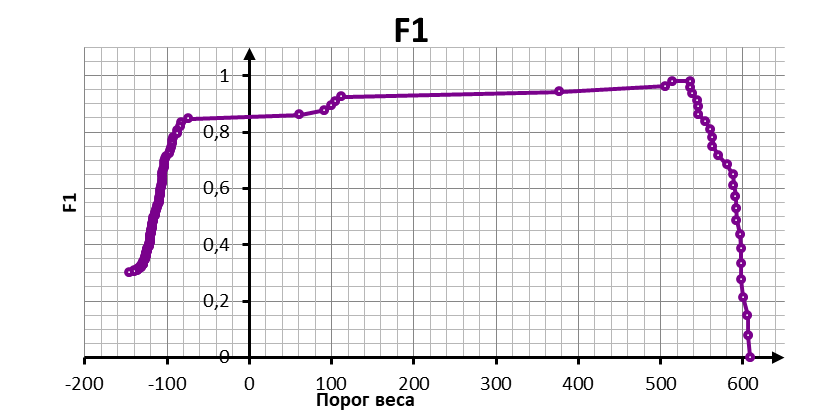
Полученная ROC имеет резкий скачок, что связано с резким скачком весов. Показатель AUC=0,78, равный площади, ограниченной ROC-кривой и осью частоты ложно-положительных результатов, свидетельствует о высоком качестве классификатора. [1]. Исходя из графика зависимости F1 от веса, можно сказать, что порог веса обеспечивающий наибольшие точность и полноту заключён между 514,00 и 536,20. (Действительно это веса лучшей неправильной находки и худшей правильной находки соответственно.)
Построение филогенетического дерева
На основе выравнивания всех последовательностей, содержащих домен CD4-extracel было построено филогенетическое дерево. Цветом выделены последовательности, содержащие двухдоменную архитектуру CD4-extracel, Tcell_CD4_C. Все клады, несодержащие эту архитектуру были схопнуты, чтобы не загромождать изображение. Можно замиетить, что двухдоменная архитектура встречается только в одной кладе, при чём в рамках этой клады только две последовательности обладают другой архитектурой. Последовательность A0A3L8S9T8_CHLGU обладает трёхдоменной архитектурой V-set, CD4-extracel, Tcell_CD4_C, то есть отличается от остальных последовательностей клады добавление в начало нового домена. Последовательность A0A0Q3URE4_AMAAE является однодоменной. (Можно предположить случайную утрату второго домена в ходе эволюции.)