Ортологи и паралоги
Составление списка гомологичных белков, включающих паралоги
Поиск с помощью blastp
- Перенесла последовательность CLPX_ECOLI в файл clpx_ecoli.fasta
- Скопировала файлы с протеомами нужных бактерий в дирректорию ~/term4
Объединила протеомы выбранных бактерий в один fasta-файл
cat AGRFC.fasta BRUSU.fasta ECOLI.fasta NEIMA.fasta PSEMY.fasta RHIME.fasta THIDA.fasta > proteomes.fasta
Создала БД из протеомов выбранных бактерий для поиска blastp
makeblastdb -in proteomes.fasta -dbtype prot -out db
Произвела поиск с помощью blastp по запросу clpx_ecoli.fasta в полученной БД с порогом на e-value 0.001, результаты записаны в файл clpx_out.txt
blastp -query clpx_ecoli.fasta -db db -evalue 0.001 -out clpx_out.txt
Список находок из выдачи BLAST
sp|P0A6H1|CLPX_ECOLI ATP-dependent Clp protease ATP-binding subun... 860 0.0
sp|A4XTZ6|CLPX_PSEMY ATP-dependent Clp protease ATP-binding subun... 658 0.0 sp|Q3SI99|CLPX_THIDA ATP-dependent Clp protease ATP-binding subun... 642 0.0 sp|Q92QQ2|CLPX_RHIME ATP-dependent Clp protease ATP-binding subun... 596 0.0 sp|Q8UFY5|CLPX_AGRFC ATP-dependent Clp protease ATP-binding subun... 596 0.0 sp|Q8G0I5|CLPX_BRUSU ATP-dependent Clp protease ATP-binding subun... 586 0.0 sp|Q9JTX8|CLPX_NEIMA ATP-dependent Clp protease ATP-binding subun... 557 0.0 sp|P0A6H5|HSLU_ECOLI ATP-dependent protease ATPase subunit HslU O... 93.6 7e-21 sp|A4XPN6|HSLU_PSEMY ATP-dependent protease ATPase subunit HslU O... 93.2 9e-21 sp|Q92TA7|HSLU_RHIME ATP-dependent protease ATPase subunit HslU O... 92.8 1e-20 sp|Q8UJ87|HSLU_AGRFC ATP-dependent protease ATPase subunit HslU O... 92.0 2e-20 sp|Q8FY12|HSLU_BRUSU ATP-dependent protease ATPase subunit HslU O... 90.9 5e-20 tr|Q3SFW1|Q3SFW1_THIDA ATP-dependent protease ATPase subunit HslU... 86.7 2e-18 sp|P0AAI3|FTSH_ECOLI ATP-dependent zinc metalloprotease FtsH OS=E... 46.2 2e-05 tr|A0A0H3GCZ6|A0A0H3GCZ6_BRUSU ATP-dependent zinc metalloprotease... 45.4 3e-05 tr|Q92M98|Q92M98_RHIME ATP-dependent zinc metalloprotease FtsH OS... 45.4 3e-05 tr|Q7CT50|Q7CT50_AGRFC ATP-dependent zinc metalloprotease FtsH OS... 45.4 4e-05 tr|Q3SJR4|Q3SJR4_THIDA ATP-dependent zinc metalloprotease FtsH OS... 43.5 1e-04 sp|Q9JUB0|RUVB_NEIMA Holliday junction branch migration complex s... 42.7 2e-04 sp|P0ABH9|CLPA_ECOLI ATP-dependent Clp protease ATP-binding subun... 43.1 2e-04 tr|Q3SJH1|Q3SJH1_THIDA ATP-dependent Clp protease, ATP-binding su... 42.7 3e-04 tr|A0A0U1RJ22|A0A0U1RJ22_NEIMA Replication-associated recombinati... 40.8 9e-04Реконструкция и визуализация
Поместила последовательности находок в один fasta-файл и отредактировала их названия, оставив только ID. Полученнный файл загрузила в NGPhylogeny.fr с параметрами:
Multiple Alignment: MAFFT
Tree Inference: FastME
Gamma distributed rates across sites: No
Starting tree: BIONJ
No refinement
Bootstrap branch supports: Yes
Number of replicates: 100
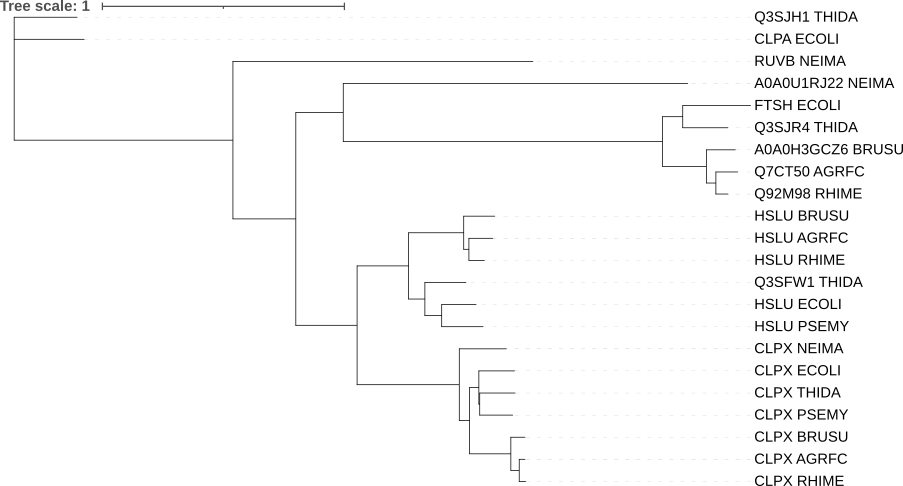
Укоренила дерево в среднюю точку и отобразила bootstrap анализ в двух вариантах: числами и символами.

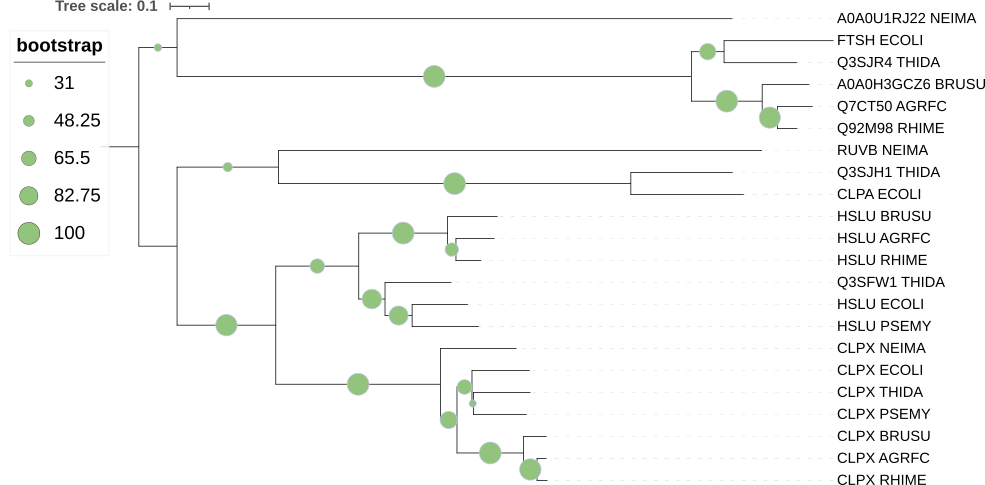
Пары:
Ортологи: CLPX_THIDA и CLPX_PSEMY; CLPX_AGRFC и CLPX_RHIME; HSLU_AGRFC; HSLU_RHIME
Паралоги: CLPX_RHIME и HSLU_RHIME; FTSH_ECOLI и CLPA_ECOLI; CLPX_AGRF и Q7CT50_AGRFC
Визуализация
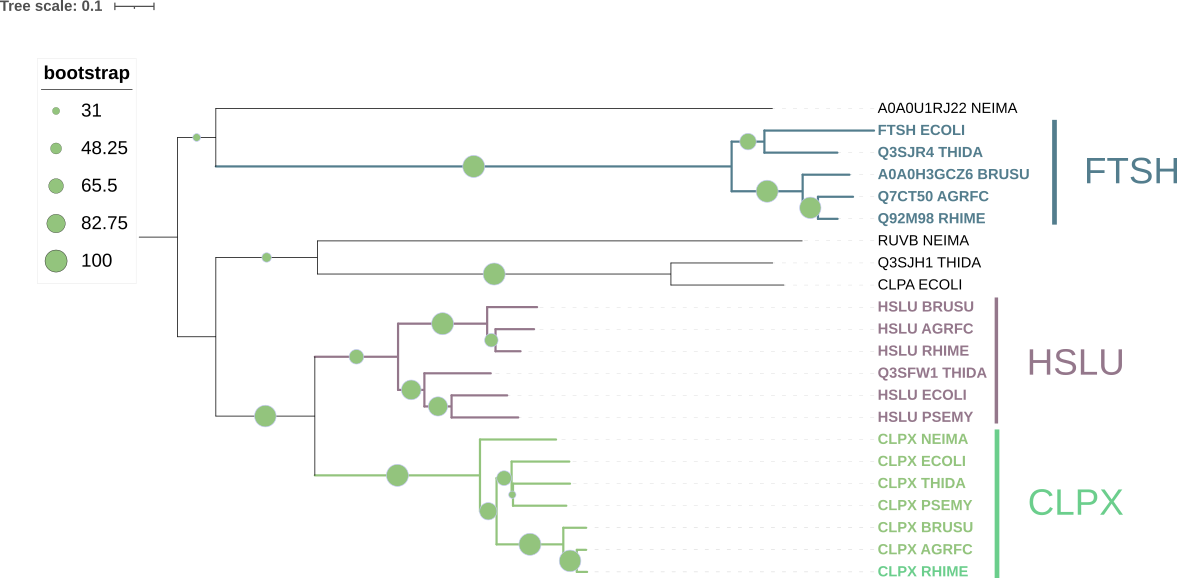

В дереве выделяются 3 ортологические группы:
CLPX – АТФ-связывающая субъединица АТФ-зависимой протеазы Clp (ATP-dependent Clp protease ATP-binding subunit ClpX). В эту группу входят белки всех 7 выбранных бактерий (включая ECOLI). Топология отличается от исходного дерева. Совпадает только топология в кладе {BRUSU, AGRFC, RHIME}, в остальном всё отличается. У несовпадающих ветвей поддержка bootstrap ниже.
HSLU – АТФ-связывающая субъединица HslU АТФ-зависимой протеазы (ATP-dependent protease ATPase subunit HslU). В эту группу входят белки 6 выбранных бактерий (включая ECOLI), то есть все кроме NEIMA. Топология совпадает с исходной
FTSH – АТФ-зависимая цинковая металлопротеаза FtsH (ATP-dependent zinc metalloprotease FtsH). В эту группу входят белки 5 выбранных бактерий (включая ECOLI), то есть всех кроме NEIMA и PSEMY. Топология совпадает с исходной.