Базы данных
Входные данные
Для этого задания я получила набор из 35 генов. Если поверхностно посмотреть на список, вот что можно заметить:
- NADSYN1 – НАД-синтетаза. Соответственно, фермент, катализирующий реакцию образования НАД.
- NADK, NADK2 – тоже что-то, связанное с НАД.
- интересный набор из 7 генов: SIRT1, SIRT2, SIRT3, SIRT4, SIRT5, SIRT6, SIRT7. Я прочитала, что это семейтсво НАД-зависимых белков.
В общем, здесь явно замешан НАД. Так что предполагаю, что гены из данного набора кодируют ферменты, участвующие в метаболизме НАД. В таком случае какая-то особая тканеспецифичность наблюдаться не должна
Gene Ontology, PANTHER
Первым делом я решила проанализировать набор генов в Gene Ontology. Ввела список из символов генов, для сравнения выбрала все гены человека. Я хоть и догадываюсь, в каком биологическом процессе участвуют белки, кодируемые данными генами, но на самом деле пока мне достоверно ничего про мой набор неизвестно. В качестве статистического теста поставила тест Фишера. Выдача в PANTHER.
Далее я заметила, что одного из 35 генов, как я думала, в выдаче нет.
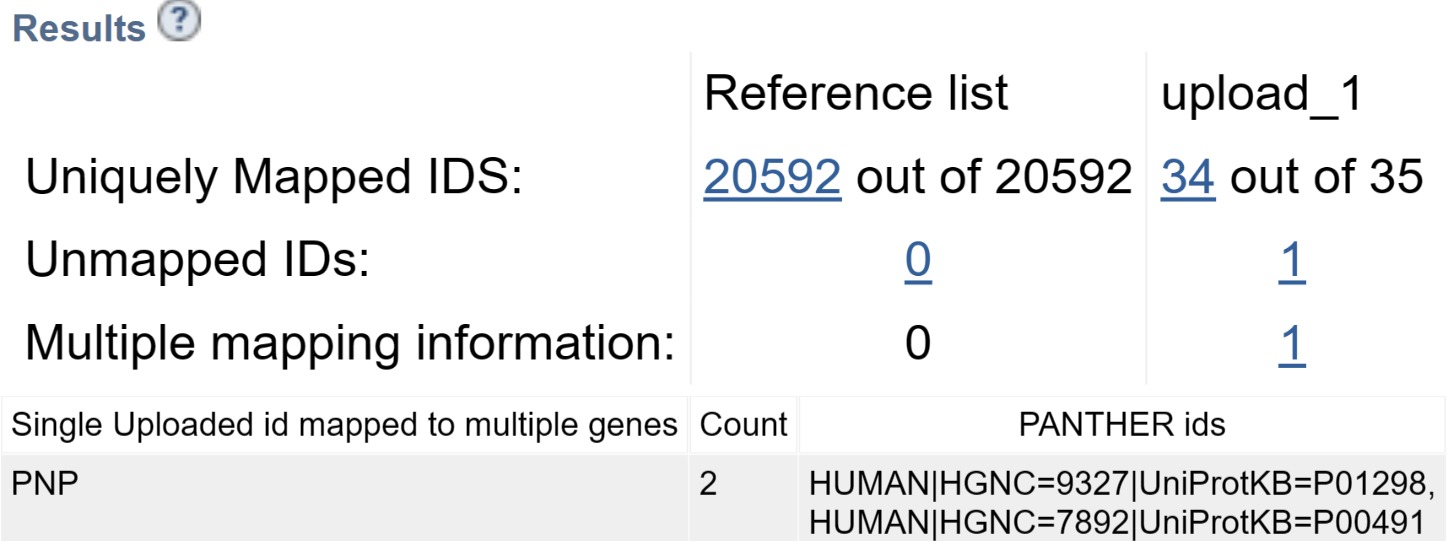
Оказалось, я не заметила, что один из генов на самом деле не ген, а локус на 2 хромосоме: NT5C1B-RDH14. В Genome Browser посмотрела, что это за локус. В этом локусе происходит естественная read-through транскрипция между соседними генами NT5C1B (5'-нуклеотидаза) и RDH14 (ретинолдегидрогеназа 14). Альтернативный сплайсинг приводит к образованию разных вариантов транскрипта, один из которых кодирует слитый белок. Так что локус в списке я заменила на названия двух этих белков: NT5C1B и RDH14
Но на этом неожиданности не закончились. Для одного ID нашлось два варианта. Это PNP. Для этого ID приведено два AC UniProt:
Пожалуй, первый белок нас не особо интересует, но, я думаю, с этим ничего не поделать, так что придётся просто смириться и помнить об этом.
Итоговая выдача в виде таблицы.
Анализ
Для начала убедилась в том, что для всех GO терминов гены в выборке перепредставлены, и p-value везде хорошее. Значит, можно спокойно анализировать.
В целом, GO термины относятся к процессам биосинтеза и катаболизма. Также некоторые термины относятся к переносу различных групп с белков: депропионилирование, десукцинилирование, демалонилирование, деглутарилирование, деацетилирование. Ещё меня удивило наличие терминов, относящихся к формированию гетерохроматина.
- Биосинтез: НАДФ
- Катаболизм: ИМФ, дАМФ, дГМФ, АМФ, дУМФ
- Метаболические процессы в целом: аллантоин, аденозин
Аллантоин является продуктом катаболизма пуринов, так что эти процессы должны быть связаны.
Подтвердилась моя изначальная догадка о том, что гены из моего набора участвуют в синтезе НАДФ. Дальше посмотрим в других базах.
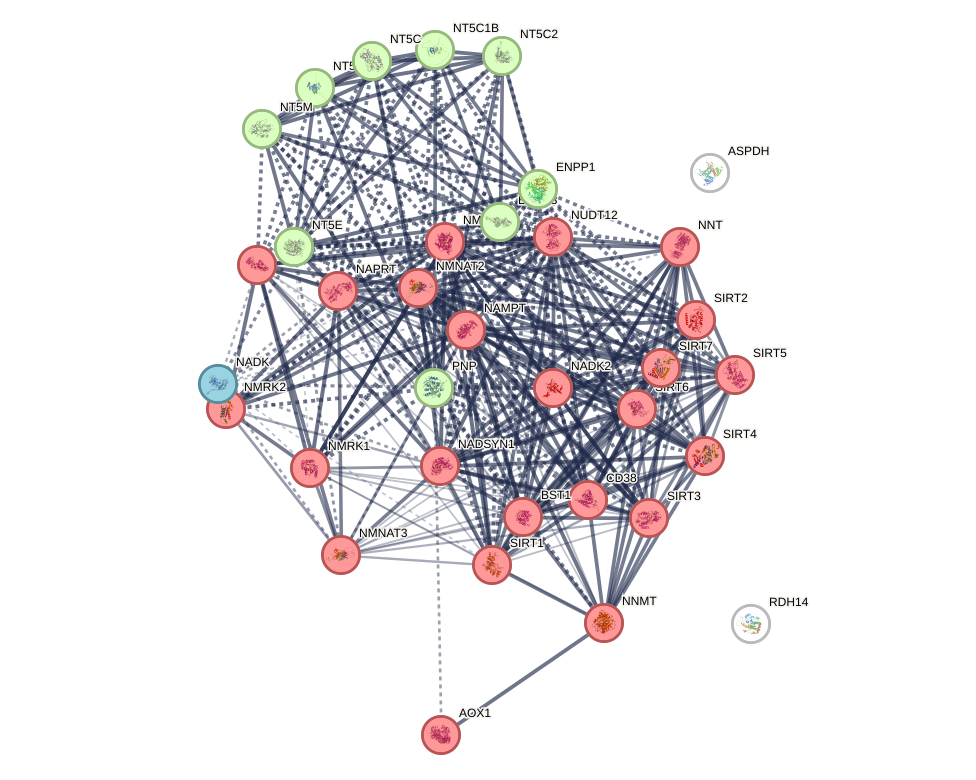
STRING
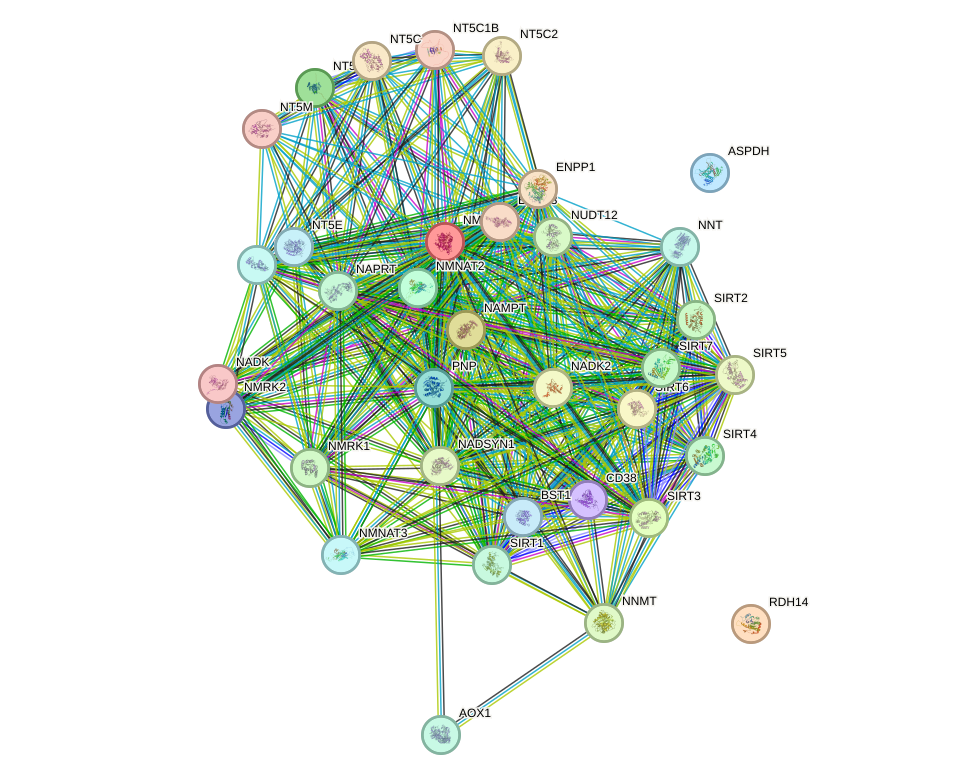
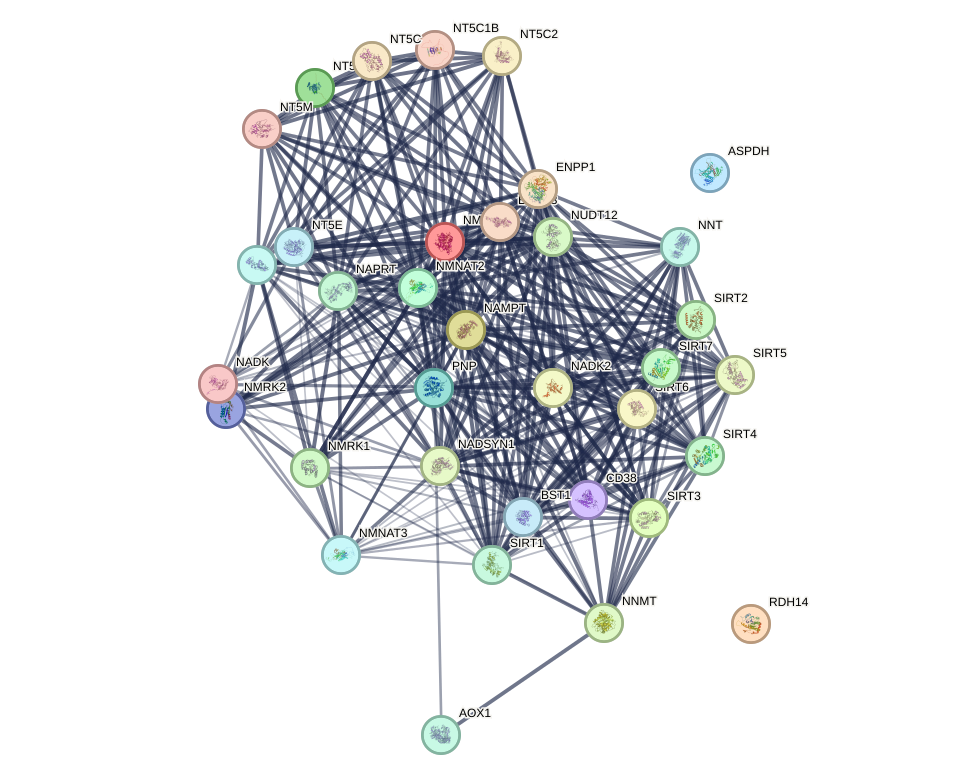
Анализ
Сразу бросается в глаза, что есть две изолированные вершины. Одна из них – тот самый ген, который был в локусе, так что это неудивительно. А вот вторая вершина меня заинтересовала. Посмотрим в NCBI Genes, что это за ген:
ASPDH. Из описания следует, что предполагается, что это ген кодирует белок, который принимает участие в биосинтезе НАДФ. Казалось бы, почему тогда он не связан с другими генами? Честно говоря, я так и не поняла, как ответить на этот вопрос. Вообще, вроде, точная функция этого белка не известна, и это только предполгаемая функция. Может, причина в этом... Но это, конечно, какое-то очень странное объяснение, так что с уверенностью назвать причину не могу.
Также из графа видно, что для всех генов известна 3D структура продукта.
Далее посмотрела, какие белки образуют комплексы друг с другом. Для этого в настройках для Network type установила 'physical subnetwork':
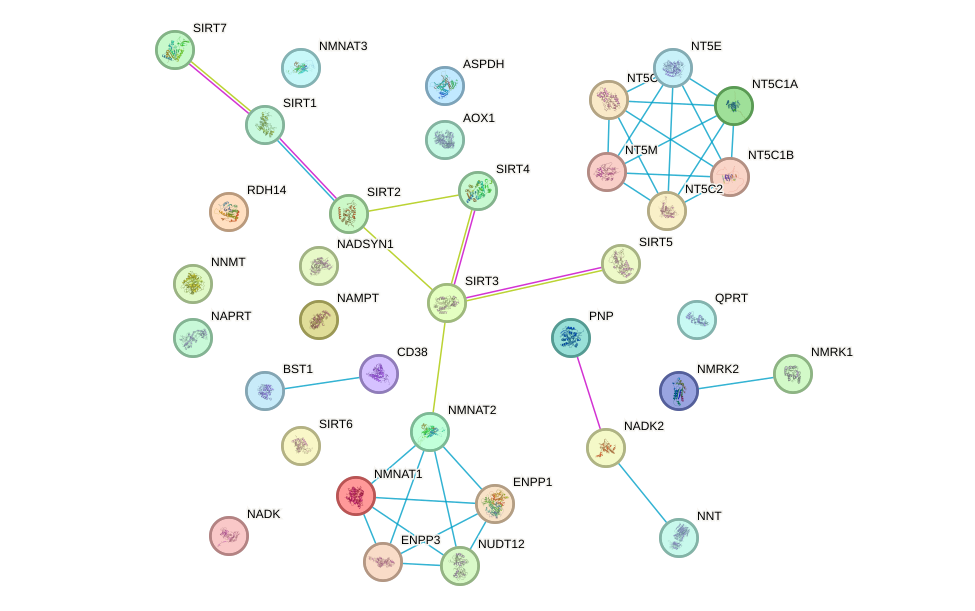
Итак, получается, образуется 6 комплексов. Вообще, ожидаемо, что белки SIRT, NT5 и NMRK образуют комплексы.
Можно теперь попробовать кластеризовать гены:
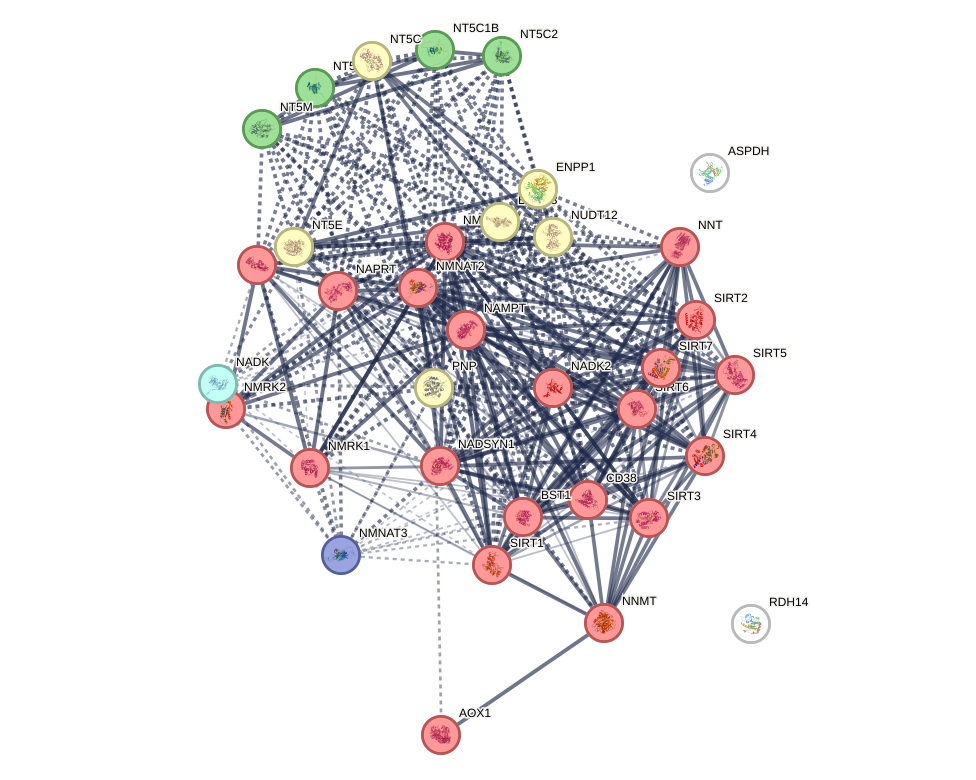
При выборе количества кластеров больше двух NADK всегда выделяется один в отдельный кластер. А при количестве болше 4 высегда выделяются 4 белка из комлекса NT5.
Далее в разделе Analysis посмотрела на биологические процессы GO:
- Процесс биосинтеза НАД (FDR 3.25e-22)
- Процессы метаболизма нуклеотидов (FDR 3.43e-25)
Некоторые из локальных кластеров STRING:
- Метаболизм никотината и никотинамида и НАД-киназа (18/23)
- Биосинтез пиридиннуклеотидов: NAPRT, NMNAT1, NMNAT2, NMRK1, NMRK2, QPRT, NAMPT (4 из них образуют 2 комплекса) (7/7)
- Метаболизм пиримидинов и биосинтез пуриновых нуклеозидмонофосфатов
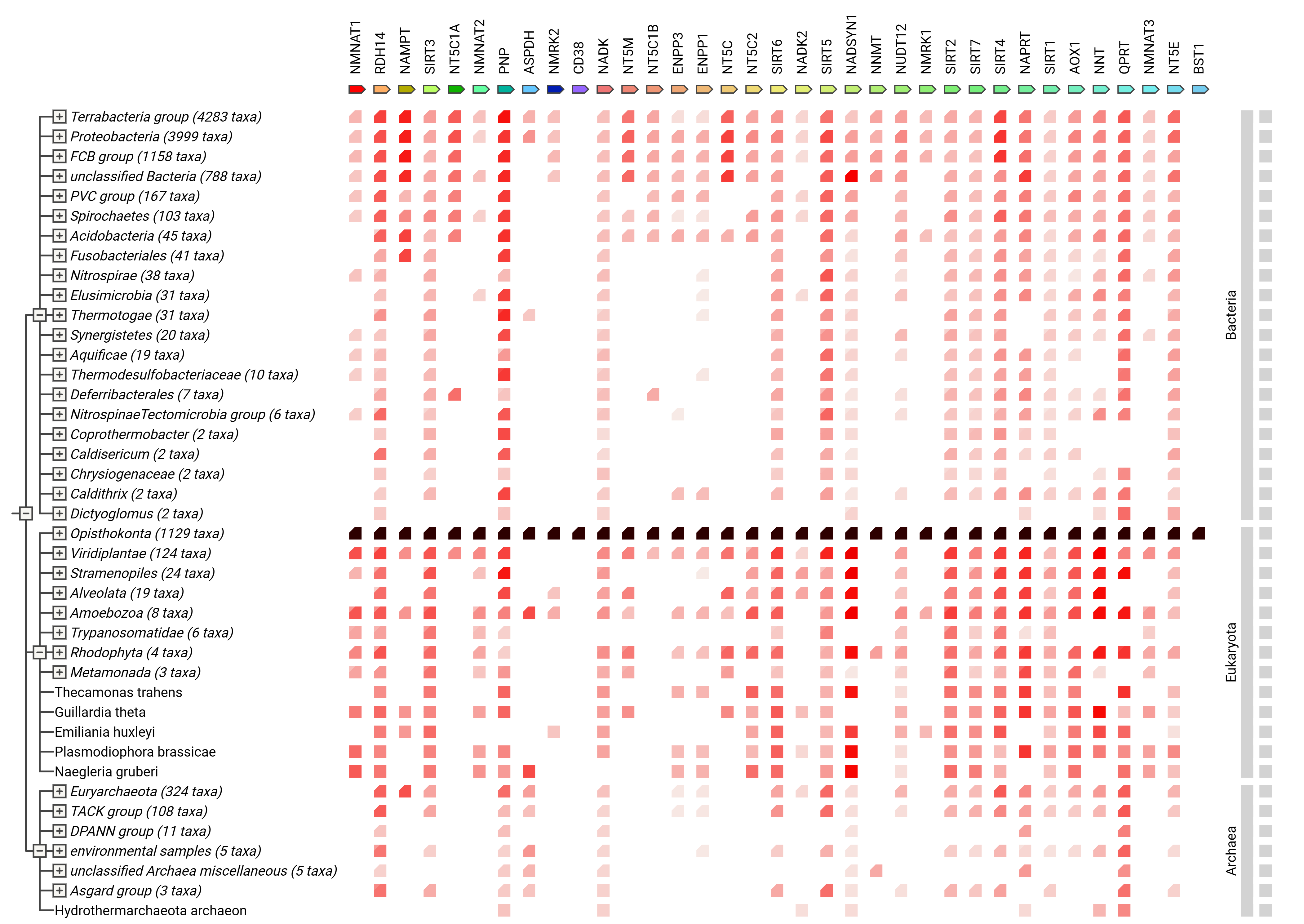
Human Protein Atlas
Так как в Protein Atlas можно смотреть только на экспрессию отдельных белков, посмотрю на экспрессию NADK, который выделялся в отельный кластер, и сравню с экспрессией NADK2.
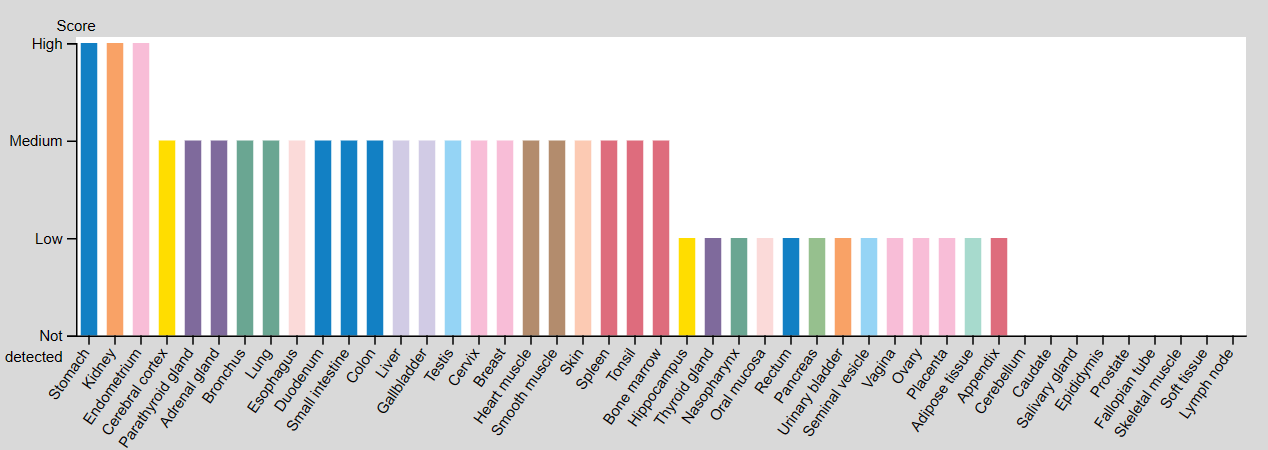
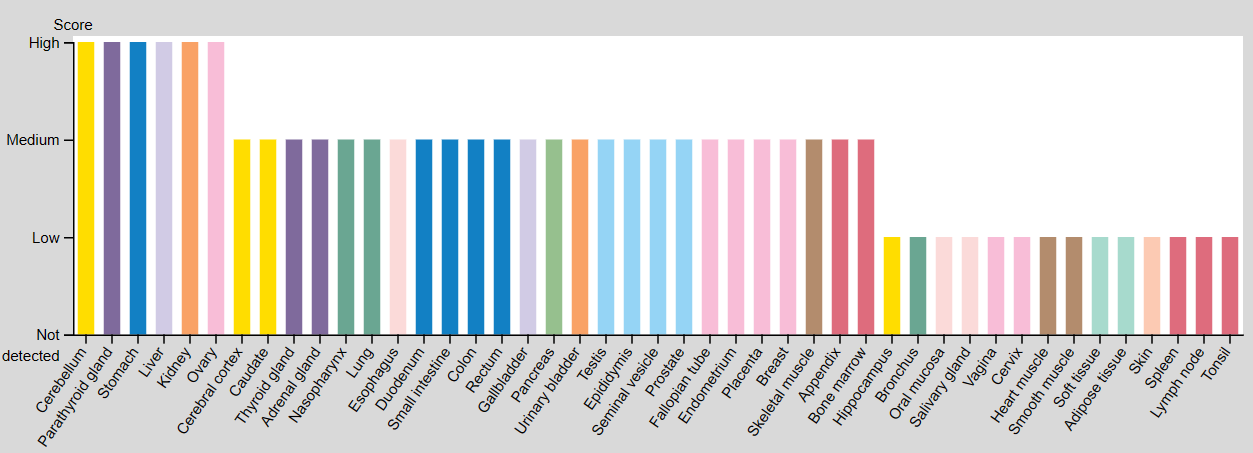
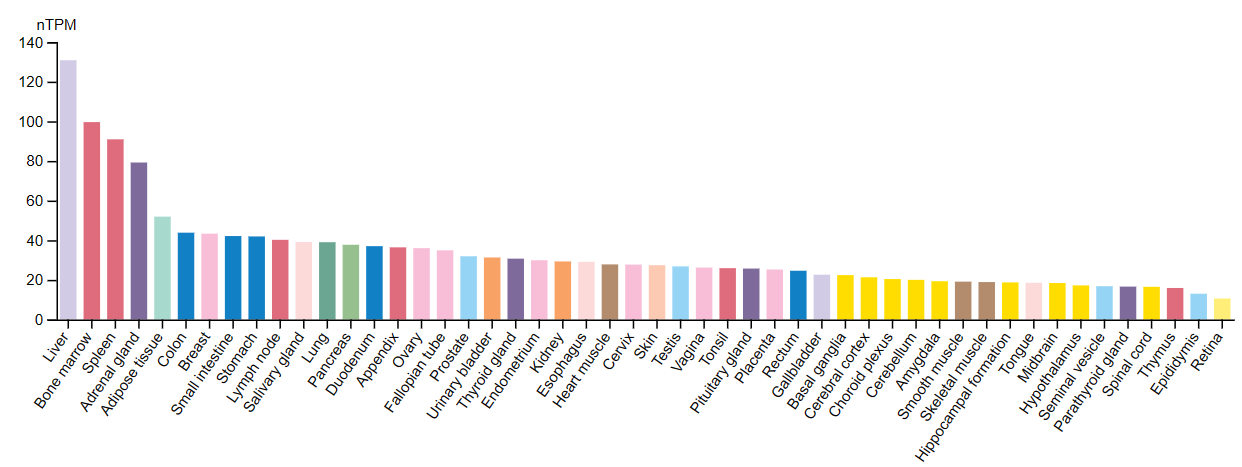
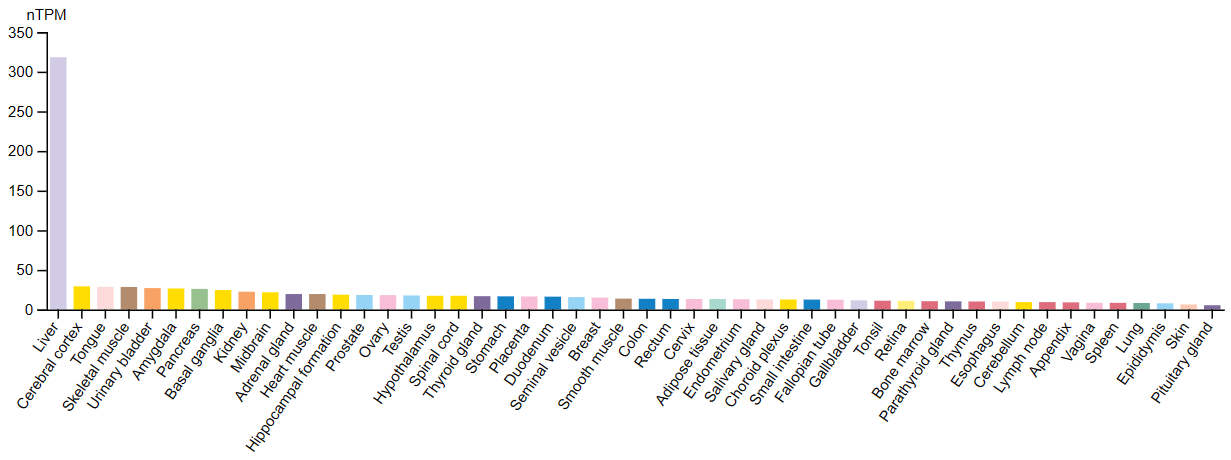
Экспрессия белка для обоих генов повышена в почках и желудке. Для NADK2 также повышена в мозжечке, паращитовидной железе, печени и яичниках. По экспрессии РНК для обоих генов на первом месте печень, но для NADK2 тканечпецифичность видна ну очень явно.
Просматривая различную информацию об этих генах, вот что ещё интересное нашла:
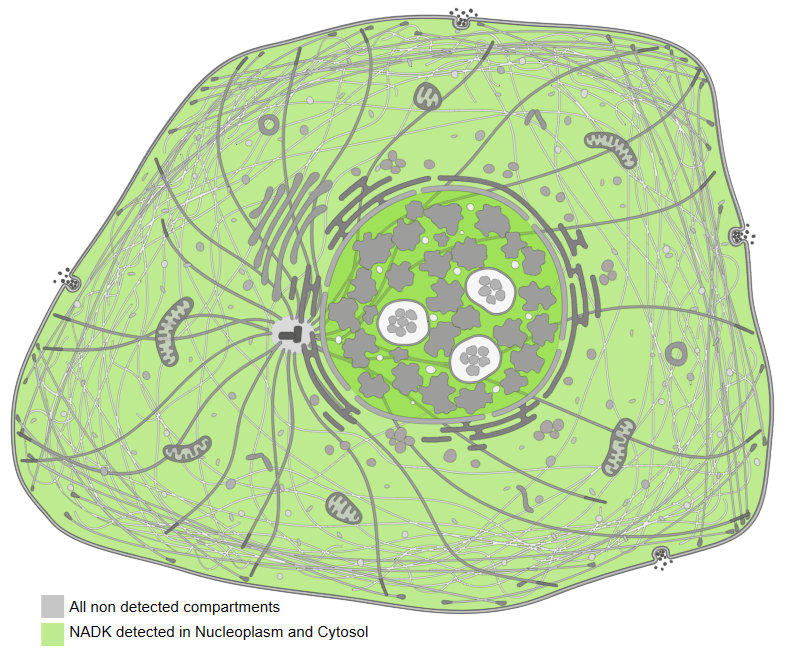
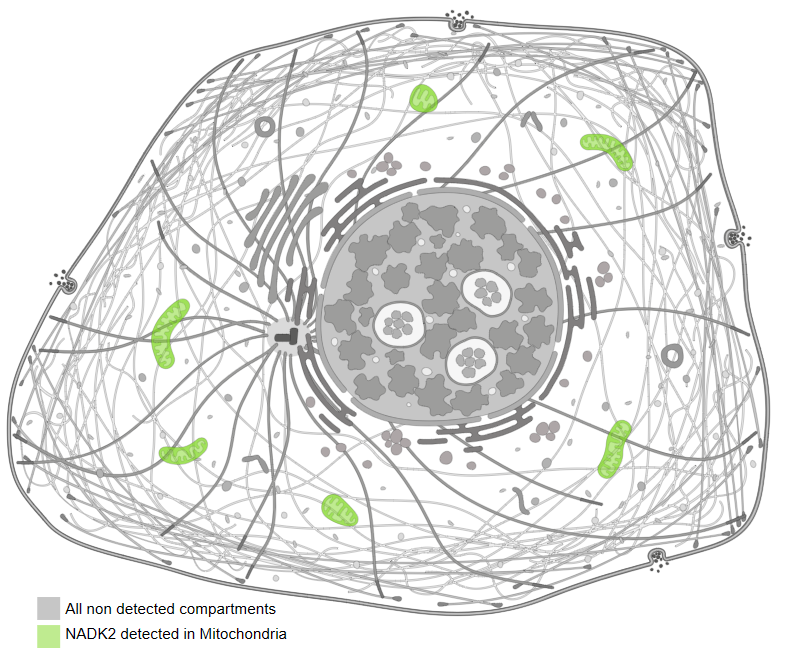
NADK обнаружен в нуклеоплазме и цитозоле, а NADK2 – в митохондриях.
Reactome
Reactome предлагает следующие процессы:
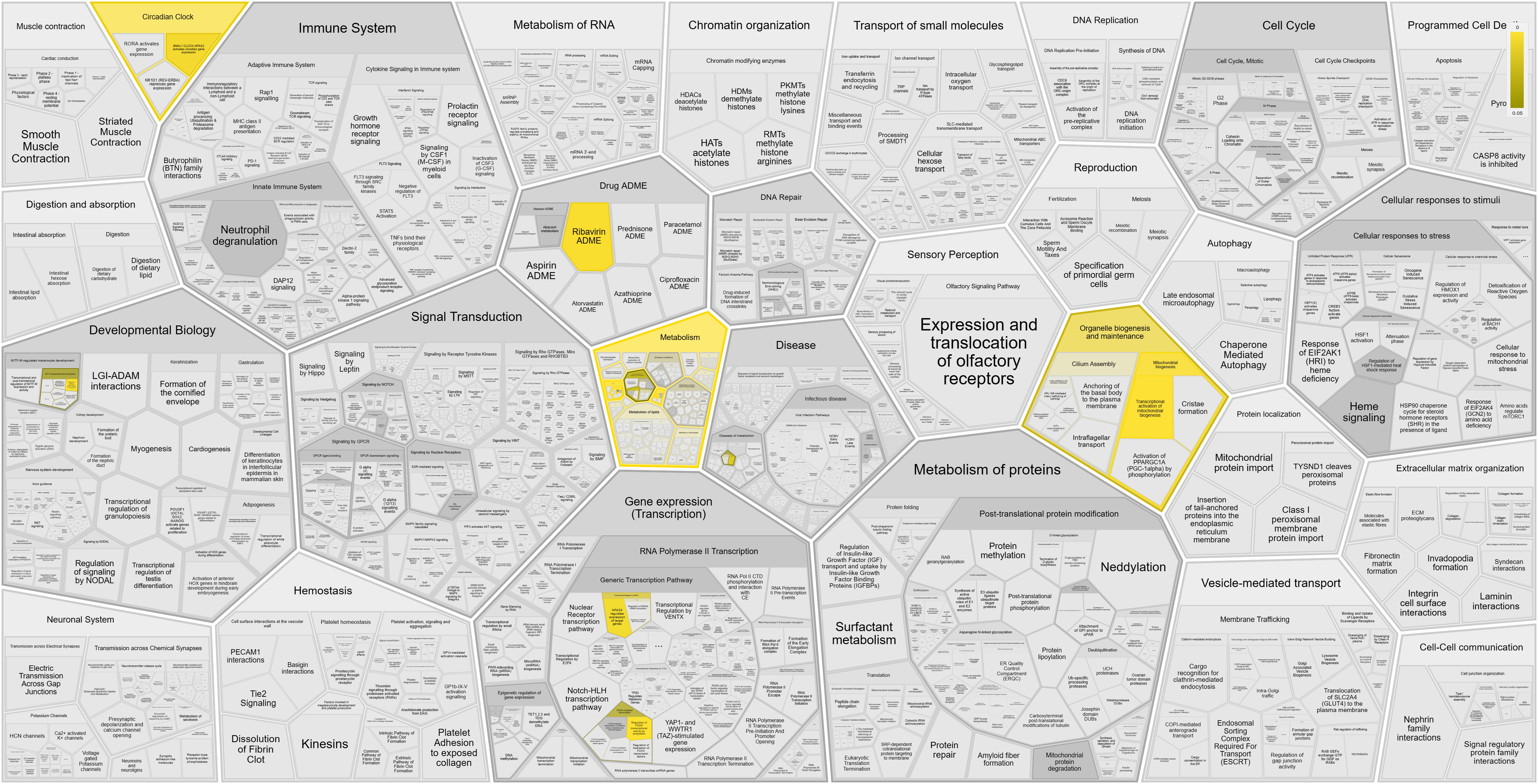
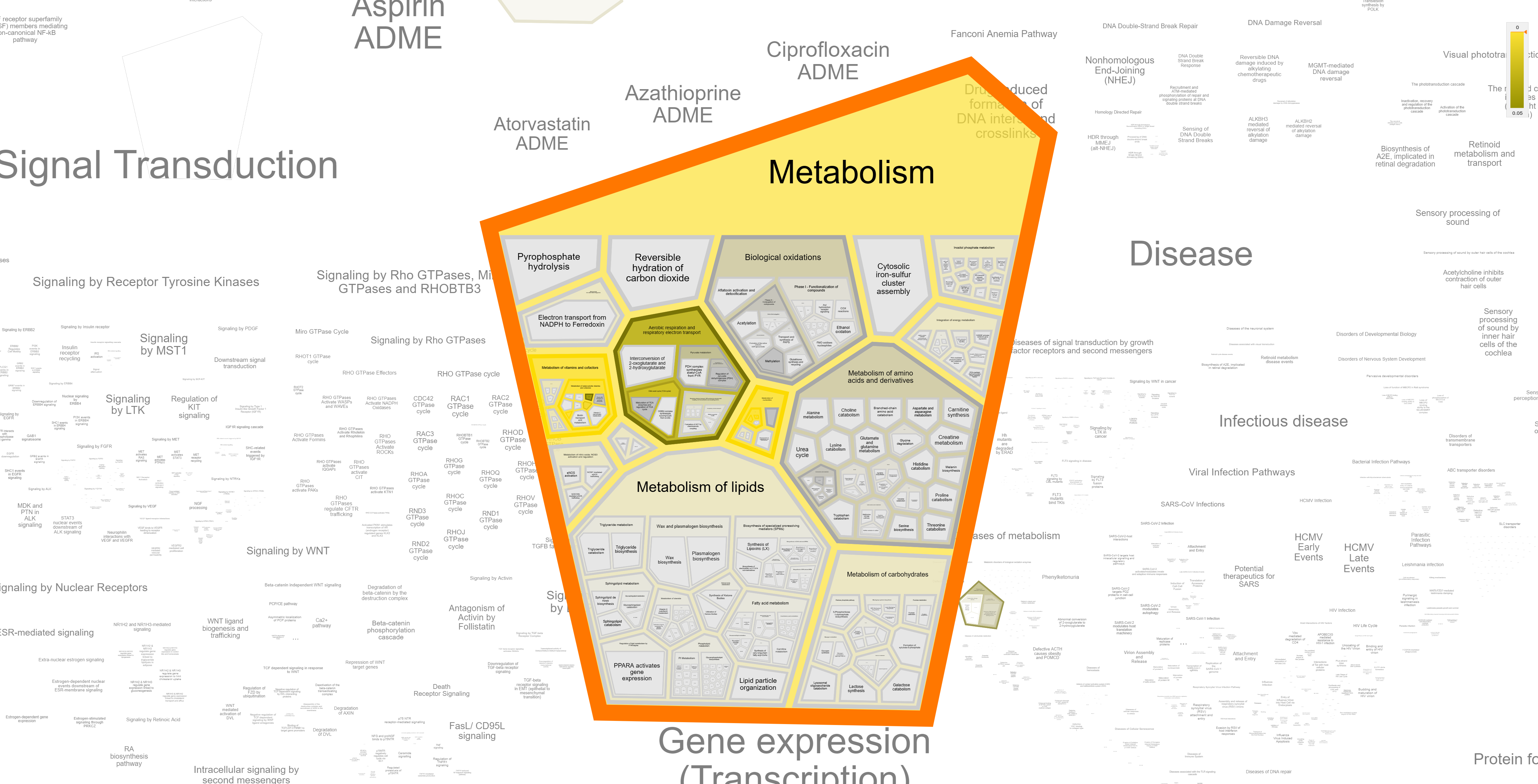

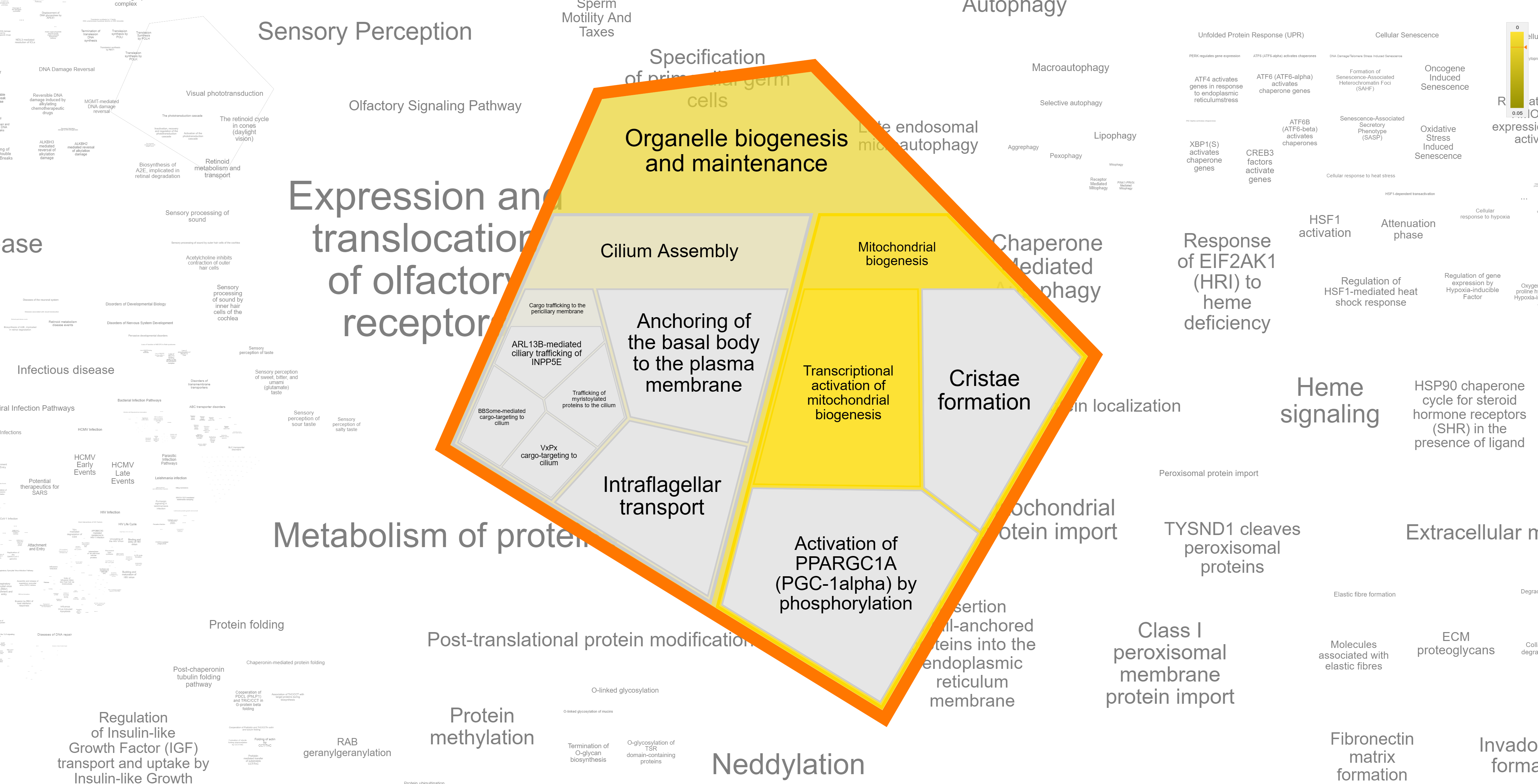
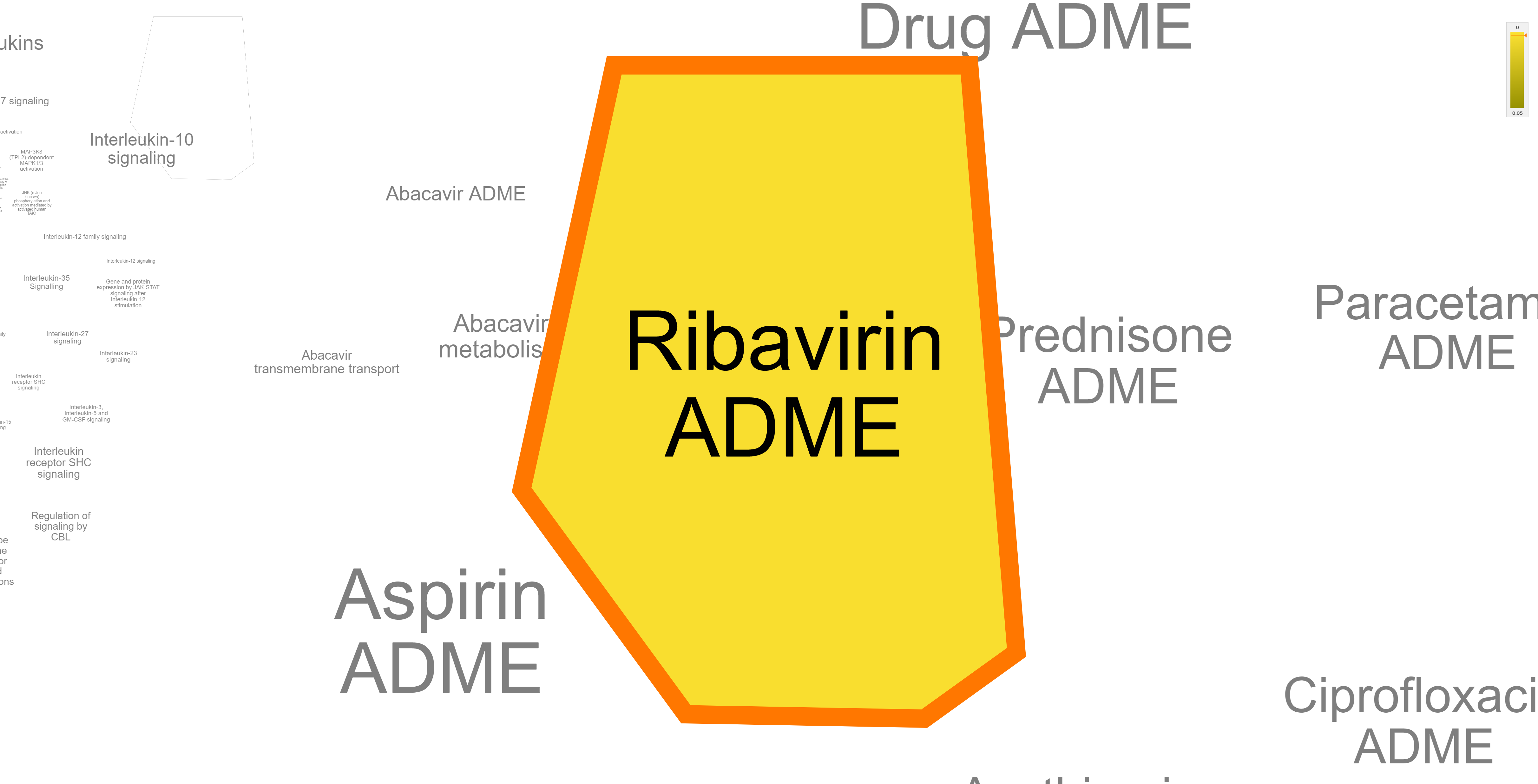
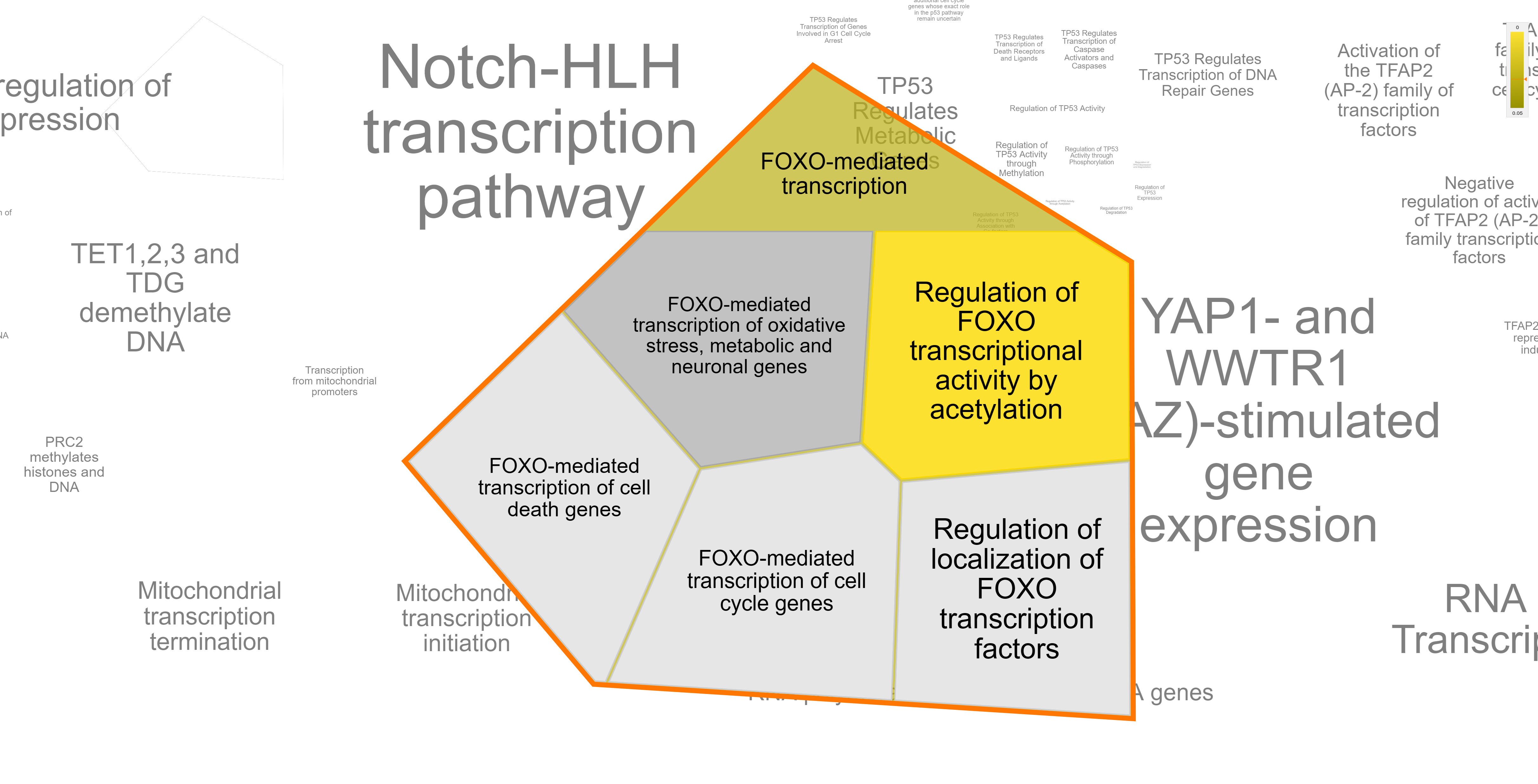
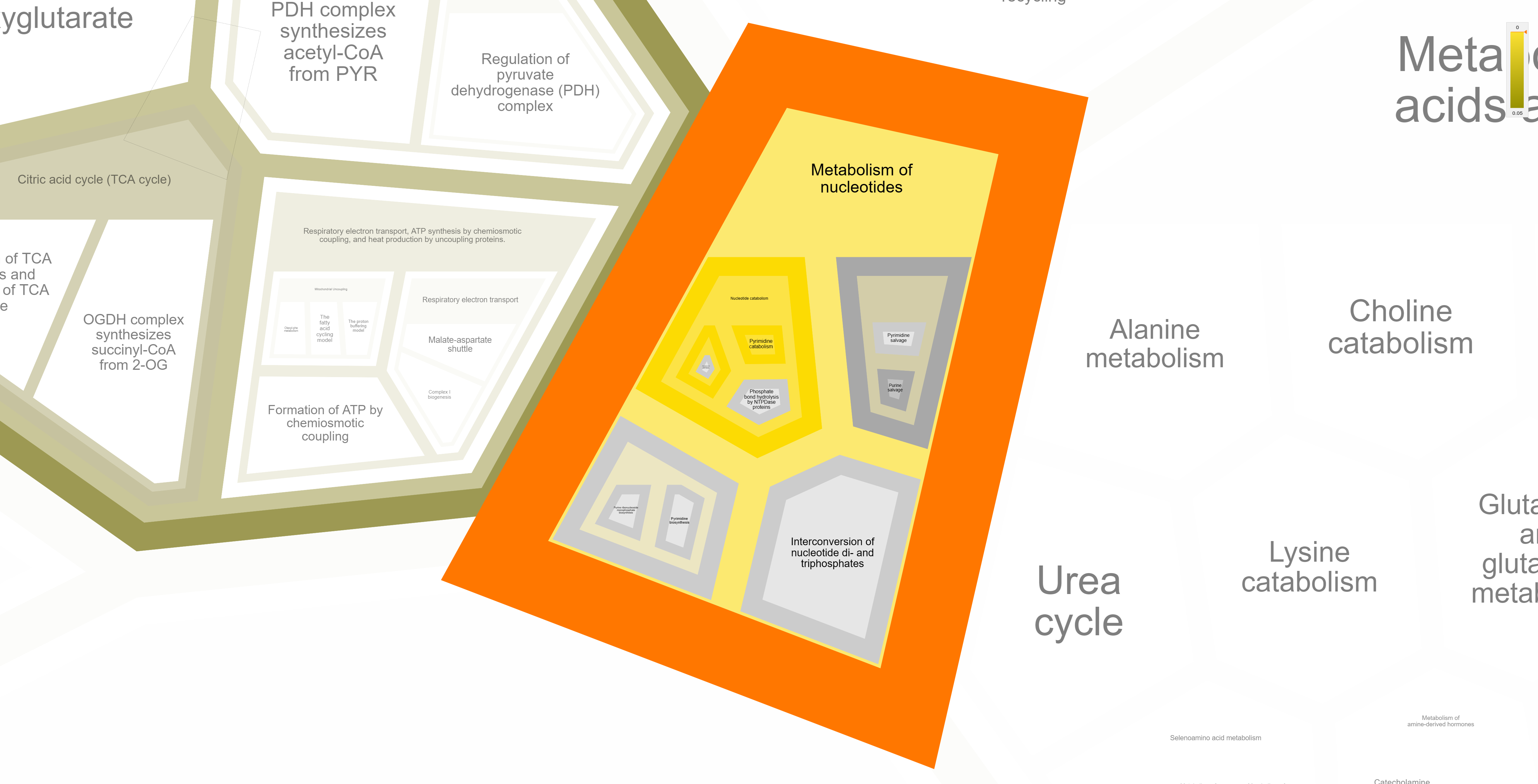
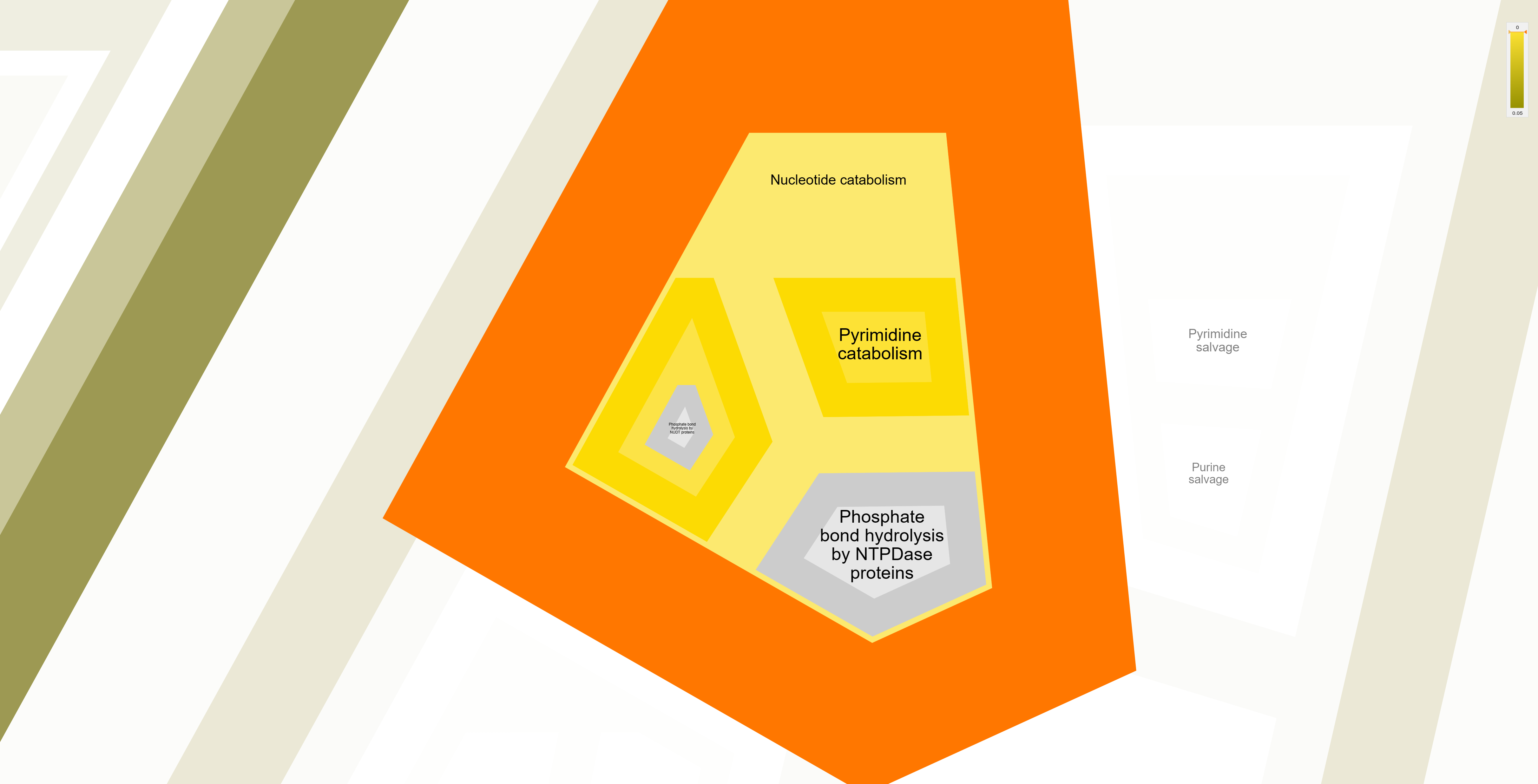
KEGG
Я посмотрела в Enrichr обогащение моего набора по данным о метаболических путях из KEGG. Наибольшее число генов принимает участие в метаболическом пути Nicotinate and nicotinamide metabolism. Нашла его в KEGG и выделила коды, которые соответствую генам из списка:
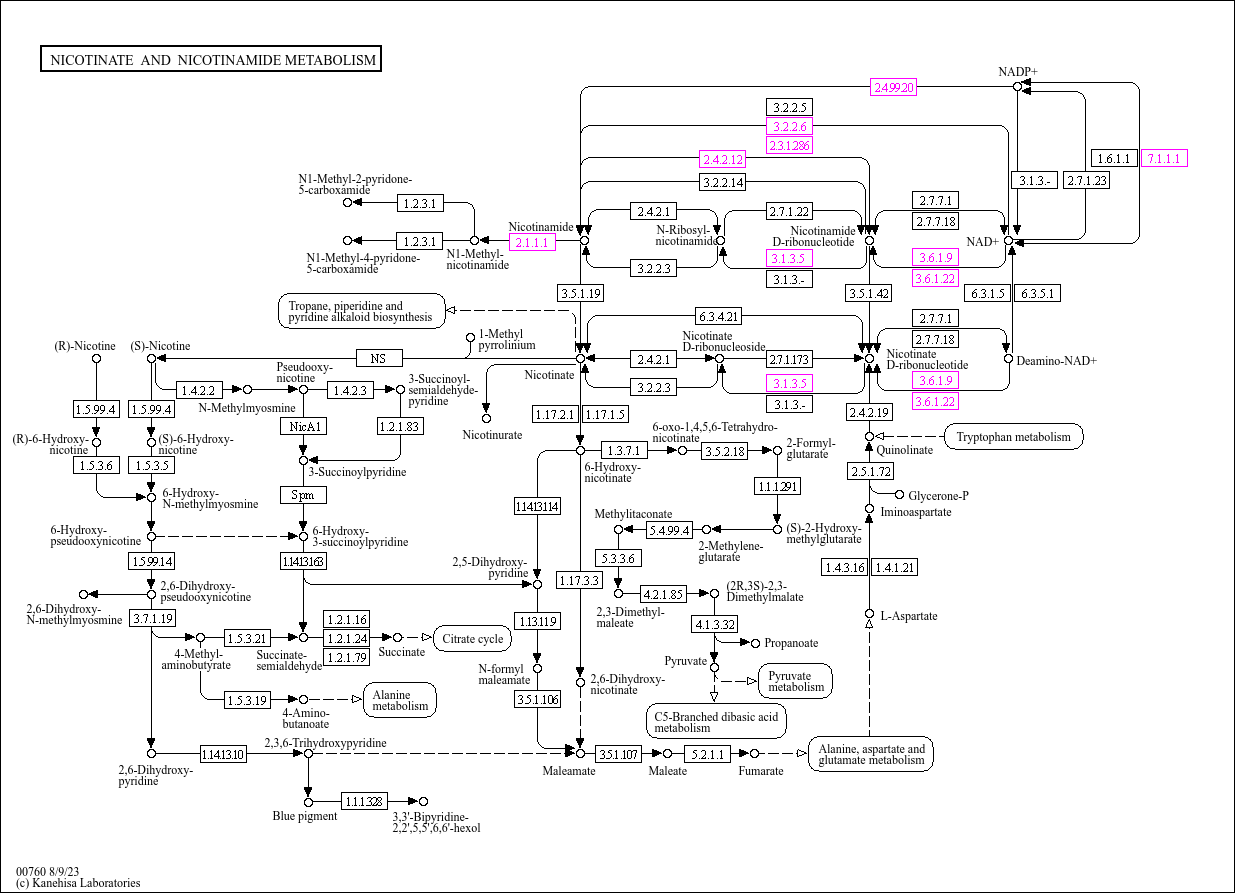
Выводы
GO мне показалась очень удобной и понятной для стоявшей перевдо мной задачей базой данных. В STRING больше всего понравилось, что можно посмотреть, какие белки собираются в комплекс, и сразу наглядно видно, какие белки не имеют никаких взаимодействий с другими. Также в разделе Analysis много полезной информации. Human Protein Atlas мне доводилось использовать ранее, это удобная база данных для изучения информации о конкретном белке, особенно когда нужно посмотреть тканеспецифичность. Для набора генов она, конечно, мало чем может быть полезна. Reactome было сложновато пользоваться, но в целом она хороша. В Enrichr тоже всё довольно понятно и информативно.
В итоге моя догадка про биосинтез НАД подтвердилась. Если всё обобщить, получается что мой набор генов в основном участвует в следующих процессах:
- Биосинтез никотинамида/НАД
- Метаболизм пиримидинов
