Реконструкция филогении по нуклеотидным последовательностям. Паралоги.
1. Построение дерева по нуклеотидным последовательностям.
Построение филогенетического дерева для восьми выбранных бактерий проводилось на основе нуклеотидных последовательностей РНК малой субъединицы рибосомы (16S rRNA).
Последовательности 16S рибосомальной РНК (рРНК) для каждой бактерии были получены из базы полных геномов NCBI (для каждого вида выбиралась одна из последовательностей 16S РНК одного штамма). Последовательности 16S рРНК содержатся в файле с расширением .frn. Все полученные последовательности были записаны в fasta-файл, в качестве названий последовательностей были оставлены только соответствующие мнемоники бактерий. Далее последовательности были импортированы в MEGA и выравнены с помощью ClustalW. Полученное выравнивание было открыто для анализа (Analyze). Дерево было реконструировано методом Neighbor-Joining (рисунок 1).
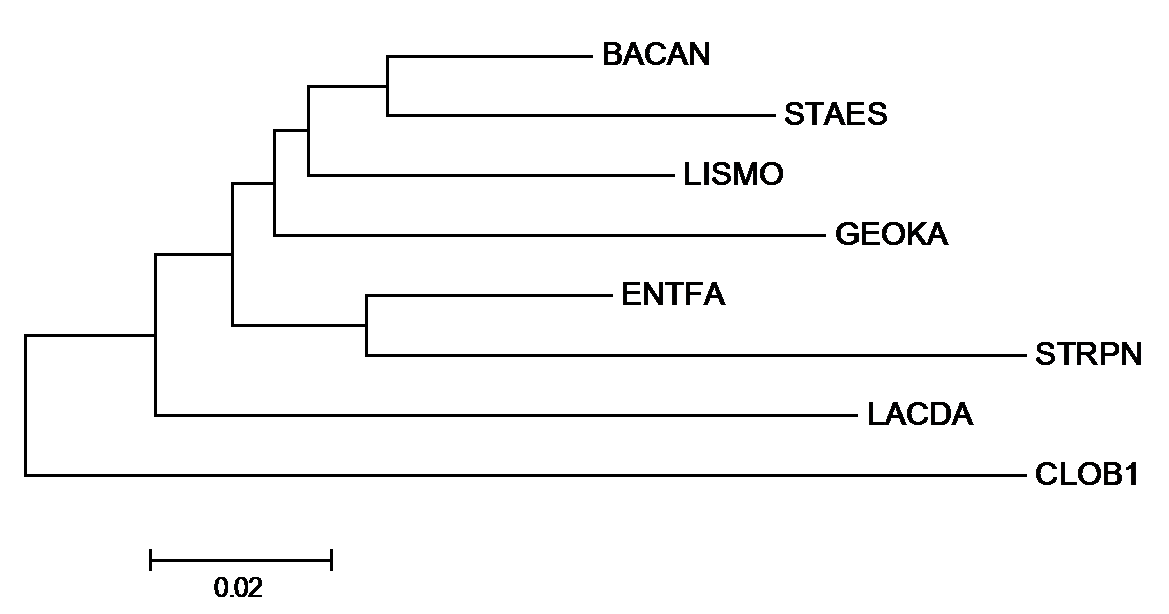
Рисунок 1. Филогенетическое дерево для восьми выбранных видов бактерий, построенное методом Neighbor-Joining на основе выравнивания нуклеотидных последовательностей 16S рРНК.
{BACAN, STAES} против {GEOKA, CLOB1, LACDA, ENTFA, STRPN, LISMO},
{BACAN, STAES, LISMO} против {GEOKA, CLOB1, LACDA, ENTFA, STRPN},
{LACDA, CLOB1} против {BACAN, GEOKA, ENTFA, STRPN, LISMO, STAES}.
Они заменили ветви следующие ветви правильного дерева:
{LACDA, ENTFA, STRPN} против {CLOB1, BACAN, GEOKA, LISMO, STAES},
{BACAN, GEOKA, LISMO} против {CLOB1, LACDA, ENTFA, STRPN, STAES},
{BACAN, GEOKA} против {CLOB1, LACDA, ENTFA, STRPN, LISMO, STAES}.
2. Построение и анализ дерева, содержащего паралоги.
Среди белков выбранных бактерий были найдены гомологи белка CLPX_BACSU (последовательность этого белка была получена на сайте Uniprot). Для этого из директории P:\y13\term4\Proteomes были получены полные протеомы выбранных бактерий и объединены в один файл. Поиск гомологов проводился с помощью blastp:
makeblastdb -in allproteomes.fasta -dbtype prot -out allproteomes1.fasta blastp -query CLPX_BACSU.fasta -db allproteomes1.fasta -evalue 0.001 -outfmt 7 -word_size 3 -out homologusБыло найдено 30 гомологичных последовательностей, которые были получены на сайте Uniprot. В программе MEGA эти последовательности были выравнены с помощью ClustalW. На основании полученного выравнивания было построено дерево с использованием метода Maximum Likelihood, рисунок 2. На построенном дереве показаны примеры ортологов (разделение путей эволюции белков в результате видообразования) и парологов (дупликация гена с последующей эволюцией).
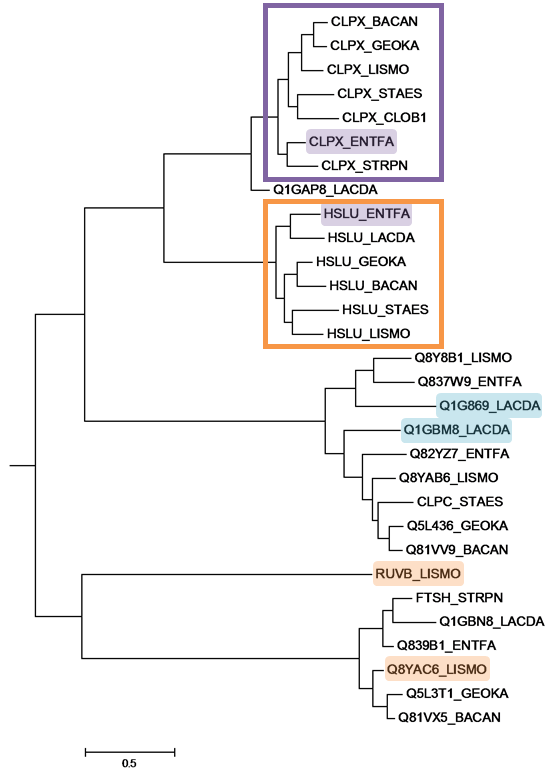
Рисунок 2. Филогенетическое дерево гомологов белка CLPX_BACSU, построенное методом Maximum Likelihood. Разным фоном подсвечены некоторые паралоги, в рамки обведены группы ортологов.
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 18.1.16