Электронная плотность. Рассеяние на электроне.
1. Выбор структуры белка для дальнейшей работы.
Я выбрала белок 4IP7 - печеночную пируваткиназу человека. Это довольно хорошо изученный и описанный белок, состоящий из 543 аминокислот.
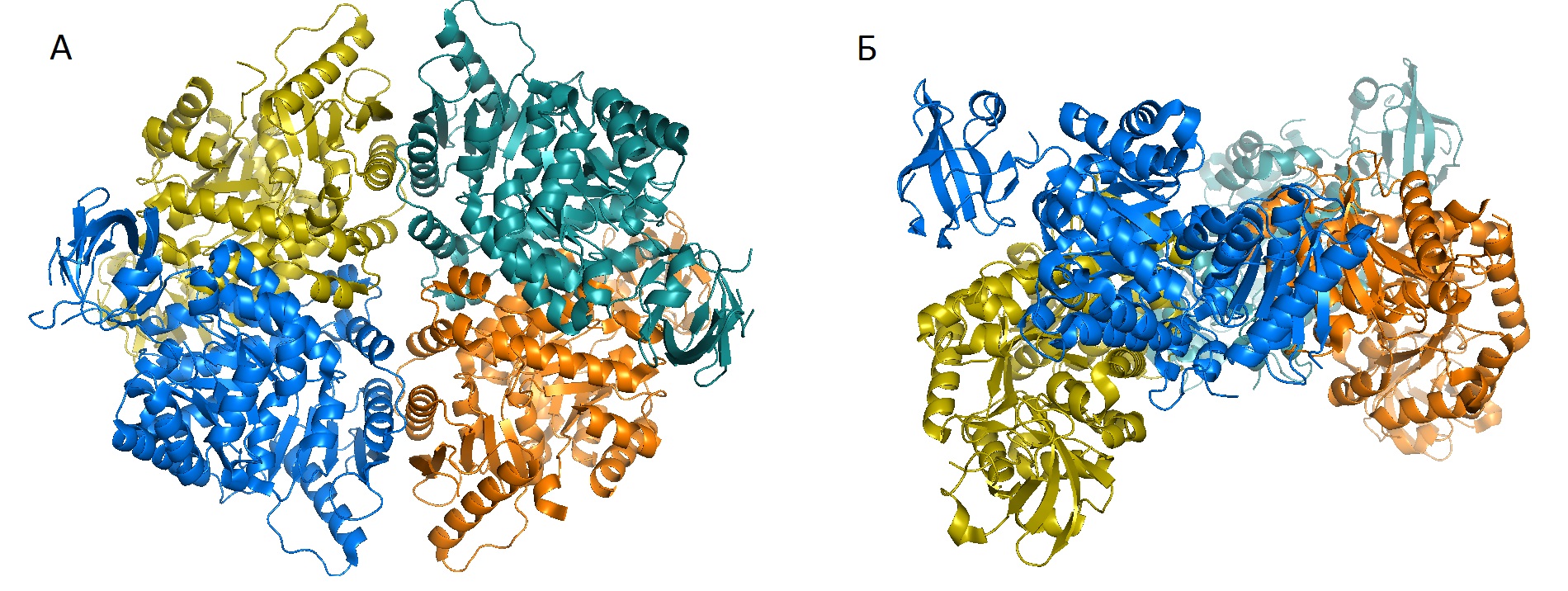
Рисунок 1. Изображение тетрамера выбранного белка в двух различных ракурсах.
- Сервис EDS знает PDB-код, то есть для данной структуры в PDB доступна не только ее модель, но и экспериментальные данные.
- Для выбранного белка имеются подходящие структурные гомологи. Проверку осуществляем следующим образом: PDBeFold => launch => Query => вводим PDB код. Убеждаемся, что в таблице с находками не меньше 5 разных белков с RMSD от 0.8 до 2.5 ангстрем и N_align от 50 до 90% аминокислотных остатков входного белка. Всего было выявлено 289 гомологов. Информация о пяти выбранных подходящих гомологах представлена в таблице 1:
| Название | RMSD | N_align | PDB ID |
| HUMAN PYRUVATE KINASE M2 | 0.83 | 504 | 1t5a |
| HUMAN M1 PYRUVATE KINASE | 0.84 | 498 | 3srf |
| HUMAN ERYTHROCYTE PYRUVATE KINASE | 0.97 | 500 | 2vgb |
| PYRUVATE KINASE FROM RABBIT MUSCLE WITH MG, K, AND L- PHOSPHOLACTATE | 1.87 | 465 | 1afq |
| CRYSTAL STRUCTURE OF LEISHMANIA MEXICANA PYRUVATE KINASE (LMPYK) IN COMPLEX WITH FRUCTOSE 2,6 BISPHOSPHATE | 2.19 | 467 | 3hqq |
2. Получение необходимых файлов.
На сайте PDB были получены PDB модель выбранной структуры, файл mmCIP и файл структурных факторов. На сайте EDS в разделе Download => Maps была получена карта электронной плотности для выбранной структуры (использовались параметры по умолчанию).
Разрешение для полученной структуры 1.8 Å:
REMARK 2 RESOLUTION. 1.80 ANGSTROMS.В .pdb-файле находится информация об атомах белка, каждому атому соотвествует строчка ATOM. В файле со структурными данными находится информация о всех структурных факторах, полученных в результате эксперимента. Ниже представлен фрагмент файла структурных факторов:
_refln.crystal_id _refln.wavelength_id _refln.scale_group_code _refln.index_h _refln.index_k _refln.index_l _refln.status _refln.F_meas_au _refln.F_meas_sigma_au _refln.F_calc _refln.phase_calc _refln.fom 1 1 1 -43 0 1 o 100.8 10.2 116.5 180.0 1.00 ...Строчки _refln соответствуют столбцам данных, которые далее приведены в файле. Информация о каждом структурном факторе указана в отдельной строчке.
В .pdb-файле находится 17146 строчек, содержащих информацию об атомах белка. В файле структурных данных находится 221991 строчек, содержащих информацию об отдельных структурных факторах. Таким образом, количество структурных факторов не соответствует количеству атомов в белке. Собственно, в этом нет ничего удивительного, так как структурные факторы описывают весь кристалл белка, а не отдельные атомы. Но используя структурные данные можно получить модель молекулы, содержащую информацию об отдельных атомах.
3. Изображение электронной плотности структуры.
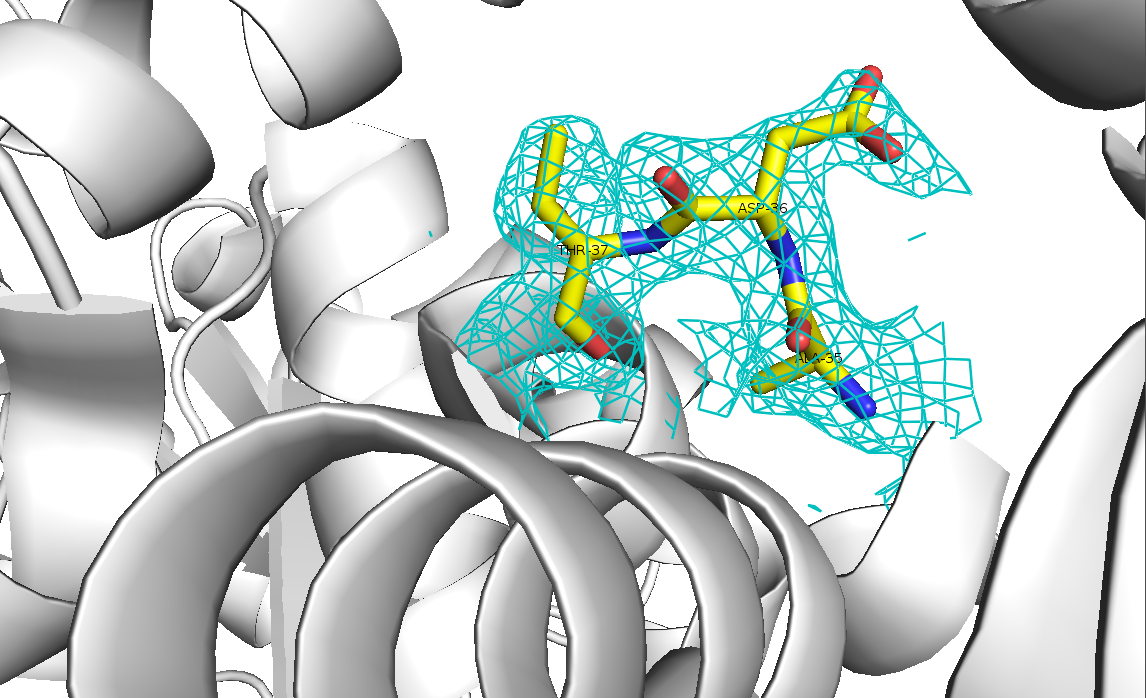
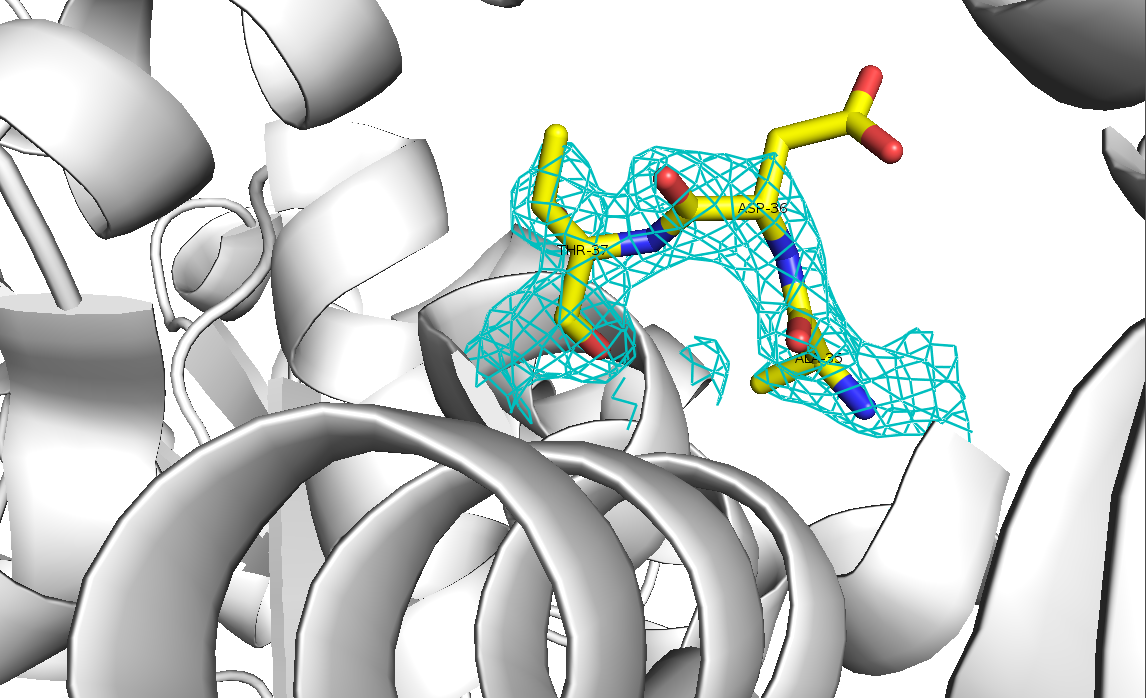
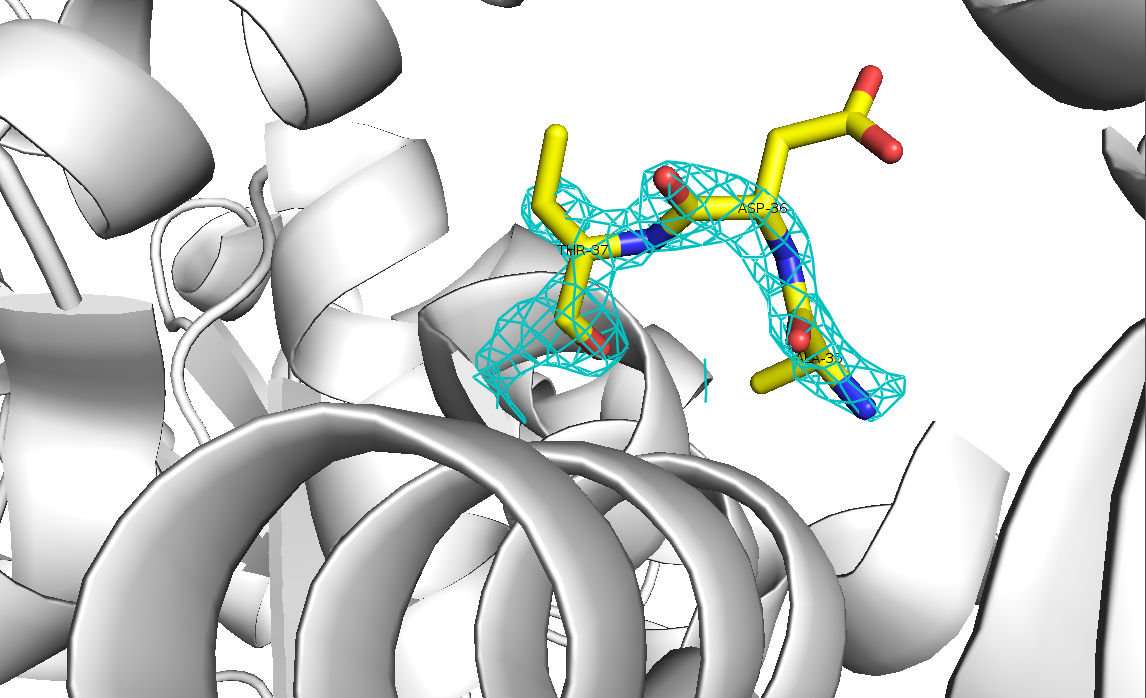
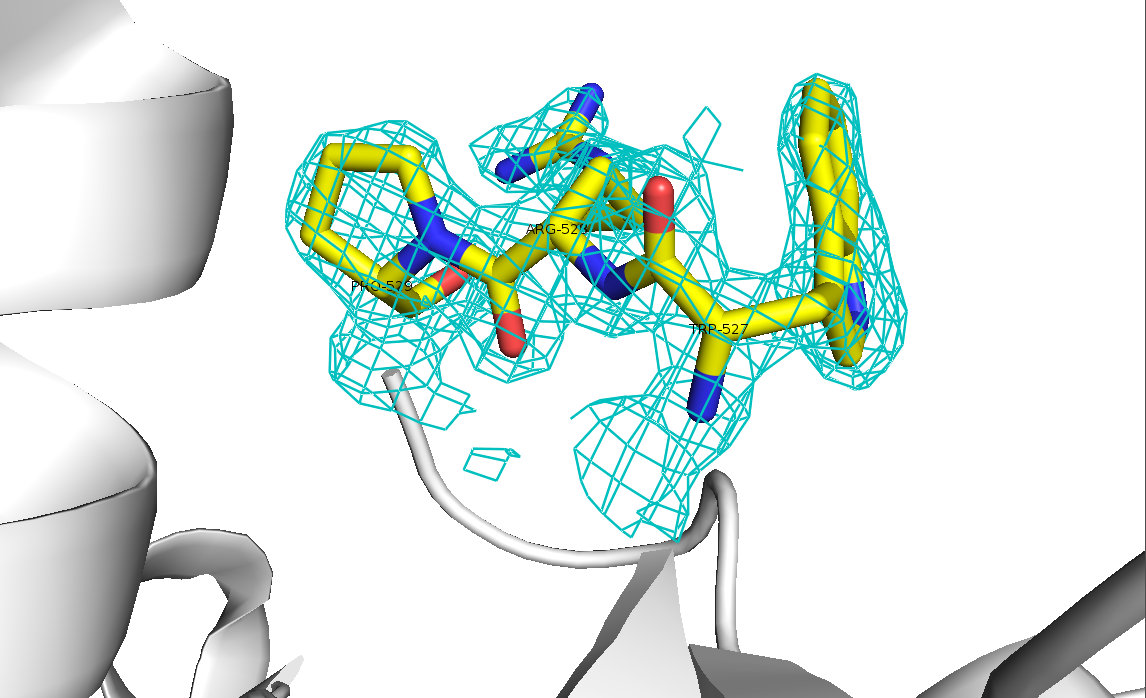
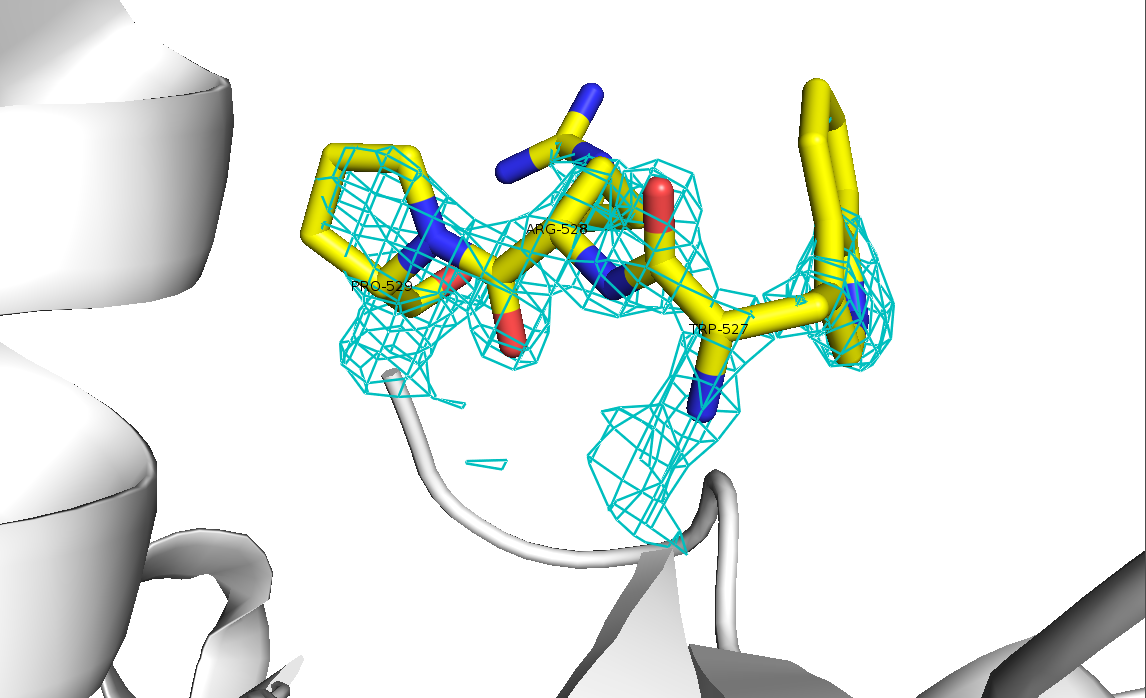
Для того, чтобы оценить качество модели выбранной структуры, на нее в программе PyMOL была наложена карта электронной плотности. На рисунке 2 изображены различные уровни подрезки электронной плотности (1σ, 2σ и 3σ) для всей полипептидной цепи. Для того, чтобы отсечь сигнал от соседних молекул, учитывалась только электронная плотность на расстоянии не больше 2.5 Å от молекулы.

Рисунок 2. Различные уровни подрезки электронной плотности для всей полипептидной цепи.
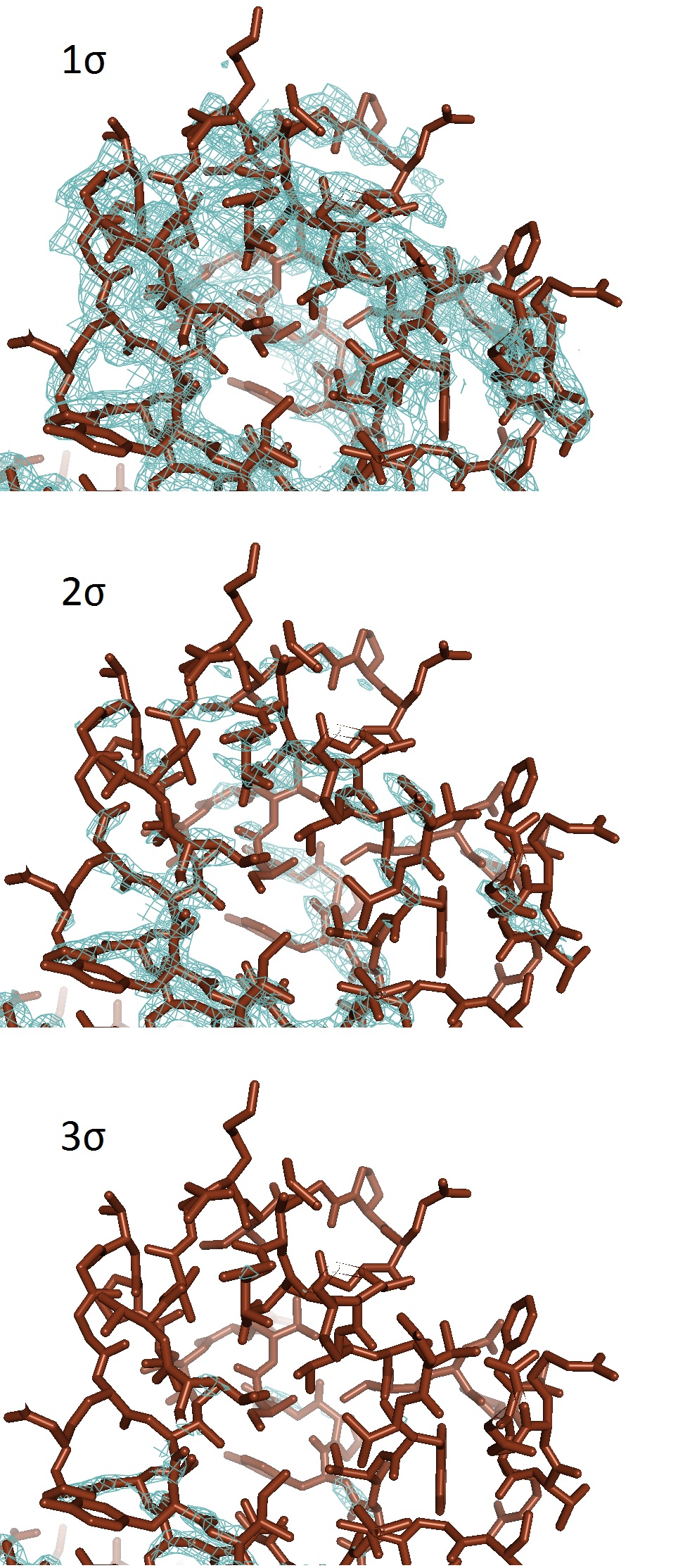
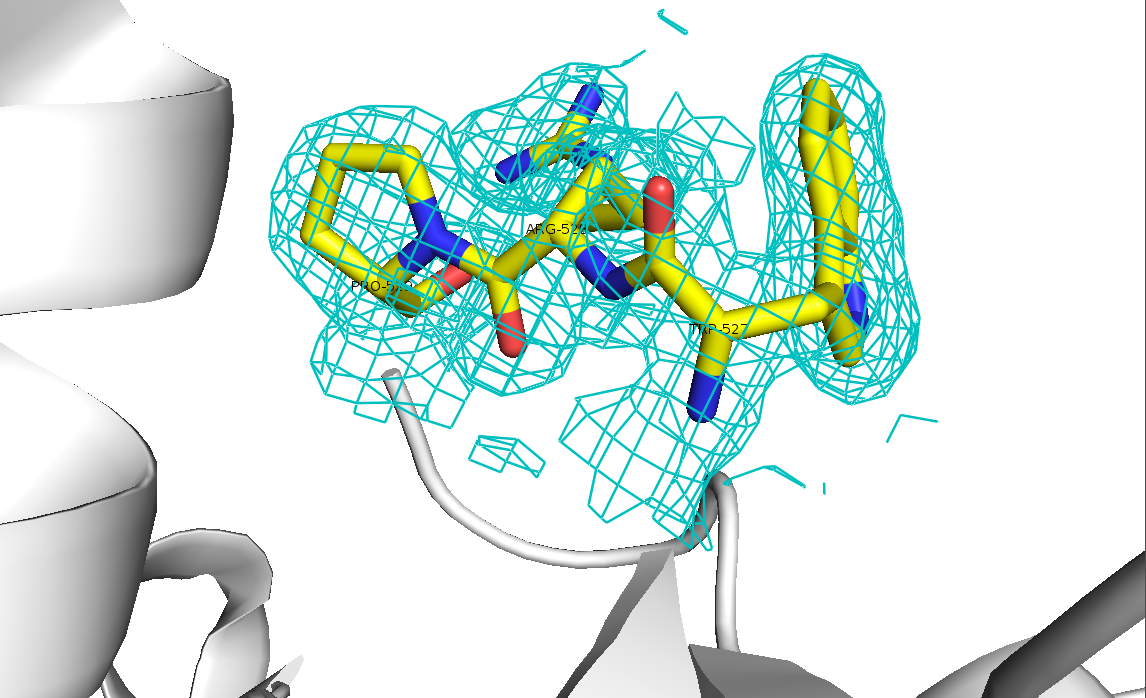
Рисунок 3. Различные уровни подрезки электронной плотности для β-бочонка. При уровне подрезки 3σ электронная плотность практически полностью исчезает на данном участке.
На рисунке 7 видны шумы, которые практически исчезают на рисунке 8 при увеличении уровне подрезки электронной плотности. На рисунке 9 уже опять пропадают участки, соответсвующие боковым радикалам аминокислот.
Таким образом, можно точно сказать, что подрезка 3σ - слишком сильная. При уровне 1σ могут встречаться не очень сильные шумы, которые практически полностью исчезают при 2σ. Для данной структуры разрешение 1.8 Å, это очень хорошее разрешение, и похоже, что модель достаточно хорошо соответствует структуре.
© Наталья Ланина
e-mail: n.lanina@fbb.msu.ru
последний раз обновлялось: 4.1.15