Задание 1
Что сделали? Построили модели дуплексов A–, B– и Z–формы ДНК, соответствующих последовательности "GATCGATCGATCGATCGATC" в случае A– и B–формы ДНК, и "GCGCGCGCGCGCGCGCGCGC" в случае Z–формы ДНК. Пользовались инструментами пакета 3DNA, а именно программой fiber. Полученные файлы можно скачать: A–форма ДНК | B–форма ДНК | Z–форма ДНК
Задание 2
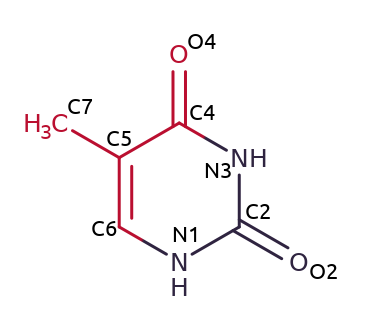
Что сделали? Из экспериментальной структуры B–формы ДНК 1BNA выбрали одно основание тимин 19 (цепь B), на основе этого азотистого основания определили атомы, обращенные в сторону большой и малой бороздок.
Как оказалось, в сторону большой бороздки были обращены атомы C4, C5, C6, C7 и O4. В сторону же малой бороздки были обращены следующие атомы: N1, C2, O2 и N3. Это распределение изображено на рисунке 1.
Дополнительно, были проанализированы экспериментальные структуры A–, B– и Z–формы ДНК ( 3V9D, 1BNA и 1TNE соответственно). С результатами изучения структур можно ознакомиться в таблице 1.
Задание 3
Для выполнения дальнеших упражнений была выбрана экспериментальная структура глутаминовой тРНК 1O0B. Для работы с пакетом 3DNA потребовалось перевести pdb файл в старый формат командой remediator. Далее воспользовались конвеером:
user@pc:~$ find_pair -t 1o0b_old.pdb stdout | analyze
Упражнение 1
Что сделали? Определили торсионные углы структуры 1O0B (таблицы 2 и 3) и схожесть структуры с различными формами ДНК. Как оказалось, структура 1O0B схожа с A-ДНК.
Упражнение 2
Что сделали? Нашли стебли в структуре тРНК. Номера нуклеотидов, образующих стебли, можно найти ниже. Красным и синим цветом отображены нуклеотиды в составе стеблей. Координаты стеблей: 902...907 – 966...971, 949...953 – 961...965, 937...944 – 926...933 и 910...912 – 923...925. В этой структуре было обнаружено 9 неканонических пар оснований (помечены хотя бы одной *) и 2 пары оснований, стабилизирующих третичную структуру тРНК (помечены x)
Strand I Strand II Helix 1 (0.014) ....>B:.902_:[..G]G-----C[..C]:.971_:B<.... (0.007) | 2 (0.007) ....>B:.903_:[..G]G-----C[..C]:.970_:B<.... (0.007) | 3 (0.005) ....>B:.904_:[..G]G-----C[..C]:.969_:B<.... (0.005) | 4 (0.009) ....>B:.905_:[..G]G-----C[..C]:.968_:B<.... (0.007) | 5 (0.012) ....>B:.906_:[..U]U-----A[..A]:.967_:B<.... (0.010) | 6 (0.012) ....>B:.907_:[..A]A-----U[..U]:.966_:B<.... (0.009) | 7 (0.005) ....>B:.949_:[..C]C-----G[..G]:.965_:B<.... (0.009) | 8 (0.004) ....>B:.950_:[..G]G-----C[..C]:.964_:B<.... (0.003) | 9 (0.003) ....>B:.951_:[..A]A-----U[..U]:.963_:B<.... (0.002) | 10 (0.007) ....>B:.952_:[..G]G-----C[..C]:.962_:B<.... (0.005) | 11 (0.005) ....>B:.953_:[..G]G-----C[..C]:.961_:B<.... (0.006) | 12 (0.002) ....>B:.954_:[..U]U-**--A[..A]:.958_:B<.... (0.010) | 13 (0.003) ....>B:.955_:[..U]U-**+-G[..G]:.918_:B<.... (0.005) x 14 (0.009) ....>B:.937_:[..A]A-**--U[..U]:.933_:B<.... (0.006) | 15 (0.004) ....>B:.938_:[..U]U-**--U[..U]:.932_:B<.... (0.005) | 16 (0.004) ....>B:.939_:[..U]U-----A[..A]:.931_:B<.... (0.003) | 17 (0.003) ....>B:.940_:[..C]C-*---G[..G]:.930_:B<.... (0.009) | 18 (0.005) ....>B:.941_:[..C]C-----G[..G]:.929_:B<.... (0.010) | 19 (0.006) ....>B:.942_:[..G]G-----C[..C]:.928_:B<.... (0.005) | 20 (0.009) ....>B:.943_:[..G]G-----C[..C]:.927_:B<.... (0.008) | 21 (0.005) ....>B:.944_:[..C]C-**--A[..A]:.926_:B<.... (0.009) | 22 (0.011) ....>B:.910_:[..G]G-----C[..C]:.925_:B<.... (0.005) | 23 (0.002) ....>B:.911_:[..C]C-----G[..G]:.924_:B<.... (0.009) | 24 (0.002) ....>B:.912_:[..C]C-----G[..G]:.923_:B<.... (0.011) | 25 (0.004) ....>B:.913_:[..A]A-**+-A[..A]:.945_:B<.... (0.005) | 26 (0.005) ....>B:.914_:[..A]A-**--U[..U]:.908_:B<.... (0.009) | 27 (0.007) ....>B:.915_:[..G]G-**+-C[..C]:.948_:B<.... (0.019) x 28 (0.026) ....>B:.919_:[..G]G-----C[..C]:.956_:B<.... (0.003) +
Упражнение 3
Что сделали? Нашли динуклеотидные пары с наибольшим (рисунки 2 и 3) и наименьшим (рисунки 4 и 5) стекинг–взаимодействием (перекрытием). Чтобы вырезать пару (number) из файла stacking.pdb, сделали следующее:
user@pc:~$ ex_str -(number) stacking.pdb step(number).pdb
Для того, чтобы создать изображение стекинга, воспользовались следующим:
user@pc:~$ stack2img -cdolt step(number).pdb step(number).ps
Дополнительные материалы
| A–форма | B–форма | Z–форма | |
|---|---|---|---|
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 28,6 | 35,7 | 45,6 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 12,8 (/3v9d//A/DT`7/P – /3v9d//B/DC`2/P) |
17,3 (/1bna//A/DG`2/P – /1bna//B/DT`20/P) |
18,1 (/1tne//A/DC`3/P – /1tne//B/DG`8/P) |
| Ширина малой бороздки (Å) | 15,8 (/3v9d//A/DT`7/P – /3v9d//B/DG`11/P) |
9,8 (/1bna//A/DC`9/P – /1bna//B/DT`20/P) |
8,2 (/1tne//A/DG`6/P – /1tne//B/8MG`10/P) |
| # | base | alpha | beta | gamma | delta | epsilon | zeta | chi |
|---|---|---|---|---|---|---|---|---|
| 1 | G | — | — | 163,4 | 88,6 | -140,9 | -65,6 | 178,3 |
| 2 | G | -70,4 | 177,6 | 54,4 | 77,2 | -156,2 | -75,1 | -168,7 |
| 3 | G | -37,3 | 164,5 | 45,3 | 80,1 | -159,0 | -60,1 | -160,4 |
| 4 | G | 153,6 | -176,7 | -179,2 | 82,4 | -144,1 | -73,9 | -171,0 |
| 5 | U | -61,1 | 172,2 | 52,3 | 81,6 | 176,5 | -79,2 | -157,4 |
| 6 | A | 179,3 | -134,9 | 170,0 | 104,3 | -81,4 | -49,9 | -167,2 |
| 7 | C | 28,8 | -148,1 | 46,8 | 87,0 | -154,4 | -66,4 | -166,7 |
| 8 | G | -58,4 | 179,5 | 50,5 | 82,8 | -155,1 | -76,1 | -159,9 |
| 9 | A | -57,0 | 173,0 | 38,9 | 80,3 | -159,8 | -85,3 | -152,4 |
| 10 | G | 146,7 | -156,2 | 178,3 | 82,1 | -132,3 | -76,8 | -178,0 |
| 11 | G | -35,8 | 159,8 | 46,6 | 80,9 | -159,3 | -64,4 | -172,0 |
| 12 | U | -85,5 | -157,9 | 50,5 | 86,4 | -160,0 | -59,6 | -160,7 |
| 13 | U | -77,6 | -177,2 | 54,6 | 88,1 | -122,8 | -101,3 | -162,5 |
| 14 | A | -117,4 | -166,8 | 59,6 | 80,4 | -144,4 | -62,1 | -175,6 |
| 15 | U | -59,7 | 164,3 | 58,1 | 83,8 | -137,9 | -63,5 | -167,6 |
| 16 | U | -62,0 | 175,8 | 49,7 | 85,3 | -158,1 | -59,0 | -156,6 |
| 17 | C | -61,8 | 179,1 | 45,0 | 85,4 | -124,9 | -134,8 | -156,3 |
| 18 | C | 106,4 | -110,9 | -178,8 | 83,9 | -105,5 | -94,8 | 180,0 |
| 19 | G | -49,4 | 145,5 | 52,4 | 76,0 | -151,2 | -69,1 | -170,1 |
| 20 | G | -60,9 | -179,4 | 49,5 | 77,4 | -148,6 | -71,7 | -159,9 |
| 21 | C | -53,1 | 170,9 | 47,6 | 79,4 | -104,1 | -128,2 | -141,4 |
| 22 | G | -148,7 | 158,2 | 41,7 | 87,6 | -150,6 | -67,6 | -176,9 |
| 23 | C | -75,6 | -167,1 | 52,2 | 83,9 | -162,1 | -74,2 | -159,8 |
| 24 | C | 136,3 | -165,8 | -171,6 | 83,3 | -126,8 | -63,1 | -172,3 |
| 25 | A | -45,3 | 165,1 | 50,8 | 79,0 | -177,7 | -73,6 | -156,7 |
| 26 | A | 174,8 | 170,5 | 172,2 | 90,5 | -119,7 | -62,8 | -170,0 |
| 27 | G | -47,6 | 152,6 | 55,4 | 83,3 | 173,8 | 24,9 | -144,4 |
| 28 | G | -105,5 | 124,5 | 77,6 | 89,5 | -170,2 | -156,1 | -133,5 |
| # | base | alpha | beta | gamma | delta | epsilon | zeta | chi |
|---|---|---|---|---|---|---|---|---|
| 1 | C | -61.2 | 178.2 | 50.2 | 84.3 | -137.0 | -88.2 | -148.2 |
| 2 | C | -66.4 | 176.0 | 52.5 | 80.6 | -150.8 | -76.3 | -160.3 |
| 3 | C | -67.7 | 179.0 | 55.6 | 82.2 | -148.1 | -72.9 | -172.4 |
| 4 | C | -63.5 | 166.5 | 54.5 | 80.8 | -161.1 | -71.0 | -152.9 |
| 5 | A | -53.4 | 171.5 | 46.8 | 83.0 | -149.4 | -78.3 | -154.4 |
| 6 | U | -56.8 | 171.9 | 44.3 | 80.7 | -153.5 | -75.6 | -161.9 |
| 7 | G | 144.8 | -145.8 | -178.3 | 84.9 | -134.7 | -78.7 | 177.9 |
| 8 | C | -47.8 | 154.9 | 46.4 | 83.8 | -152.3 | -87.4 | -152.8 |
| 9 | U | -73.3 | -165.3 | 46.2 | 80.2 | -152.4 | -87.6 | -152.5 |
| 10 | C | -67.2 | -178.7 | 53.4 | 85.1 | -165.8 | -56.5 | -170.5 |
| 11 | C | -90.2 | -161.0 | 50.9 | 85.4 | -152.9 | -76.6 | -169.0 |
| 12 | A | -73.2 | -118.1 | 67.2 | 103.4 | -176.6 | -101.5 | -91.5 |
| 13 | G | 40.5 | -173.5 | 37.3 | 92.6 | -27.5 | 147.2 | -118.0 |
| 14 | U | -59.0 | 162.2 | 50.8 | 83.9 | -109.1 | -119.7 | -154.0 |
| 15 | U | 132.7 | -136.4 | -177.3 | 84.7 | -108.5 | -76.7 | 179.0 |
| 16 | A | -56.9 | -166.3 | 45.1 | 83.6 | -154.6 | -94.0 | -153.3 |
| 17 | G | -63.0 | 163.8 | 49.3 | 78.2 | -166.2 | -67.0 | -170.8 |
| 18 | G | 151.0 | 176.2 | -179.5 | 87.7 | -126.6 | -73.9 | -175.4 |
| 19 | C | -68.4 | -165.7 | 45.4 | 85.8 | -176.8 | -61.9 | -139.6 |
| 20 | C | -57.1 | 174.4 | 49.4 | 79.2 | -152.1 | -69.4 | -154.3 |
| 21 | A | -72.1 | -178.9 | 53.7 | 80.6 | -155.0 | -59.4 | -159.2 |
| 22 | C | -58.7 | 170.1 | 50.7 | 77.1 | -147.5 | -53.9 | -166.5 |
| 23 | G | -62.2 | 173.9 | 56.0 | 80.6 | -149.9 | -69.2 | -167.2 |
| 24 | G | -54.5 | 166.2 | 48.5 | 80.9 | -154.3 | -75.9 | -170.9 |
| 25 | A | -36.2 | -175.4 | 57.8 | 85.6 | 169.2 | 20.2 | -113.3 |
| 26 | U | -66.7 | -138.4 | 57.6 | 82.0 | -152.1 | -128.2 | -161.4 |
| 27 | C | -5.9 | 130.7 | 49.6 | 97.5 | 177.0 | 152.8 | -153.0 |
| 28 | C | 151.5 | -152.4 | 49.3 | 84.6 | -139.7 | -75.4 | -165.0 |