Предсказание вторичной структуры тРНК (ID 1O0B)
Мы продолжаем изучение объекта предыдущего практикума — глутаминовой тРНК 1O0B. В этом задании мы намерены предсказать инвентированные повторы во вторичной структуре тРНК. Для этого мы воспользовались программой einverted из пакета EMBOSS
Что сделали? Запустили программу einverted с измененными параметрами: Gap penalty – 4, Minimum match score – 15, так как тРНК, по–видимому, небольшая. Но и меньше параметры брать не стоит, так как гэпов будет много.
В итоге, получили один инвертированный повтор:
SEQUENCE: Score 23: 9/9 (100%) matches, 1 gaps 1 tggggtatcg 10 ||||||| || 71 accccat-gc 63
Предсказание вторичной структуры тРНК по алгоритму Зукера
Также предсказание вторичной структуры получили с помощью применения алгоритма Зукера, воспользовавшись командой RNAfold из пакета Viena Rna Package. Корректную структуру тРНК получили при первом же запуске (рисунок 1)
Дополнительно, была получена настоящая экспериментальная вторичная структура тРНК. Ее получили с сервера RNApdbee в виде изображения (рисунок 2). Затем всю полученную информацию сравнили и результат записали в таблицу 1. Как оказалось, RNAfold и find_pair оказались довольно точными, а einverted дал плохой результат.
Поиск ДНК-белковых контактов в заданной структуре (ID 1P47)
Что сделано? Для выполнения данного задания была выбрана структура комплекса ДНК с белком Zif268 (ID 1P47).
Упражнение 1
Следующие действия были проделаны с помощью JMOL:
- Определили множество атомов кислорода 2'-дезоксирибозы (set1),
- Определили множество атомов кислорода в остатке фосфорной кислоты (set2),
- Определили множество атомов азота в азотистых основаниях (set3).
Также сделали следующее:
- Создали скрипт-файл с определениями этих множеств (скрипт №1),
- Создали скрипт-файл, вызов которого в JMol дает последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3 (скрипт №2).
Скрипты можно скачать здесь: Первый скрипт | Второй скрипт.
Упражнение 2
Что сделано? Были предсказаны многочисленные контакты между белком и нуклеиновой кислотой на основании расстояния между атомами. Результат можно найти в таблице 2.
В целом, можно сказать, что основной вклад в полярные взаимодействия дают остатки фосфорной кислоты, что довольно логично. Еще можно сказать, что неполярные взаимодействия дают только нуклеозиды. Интересно, что все неполярные взаимодействия с азотистыми основаниями находятся в области большой бороздки.
Nucplot
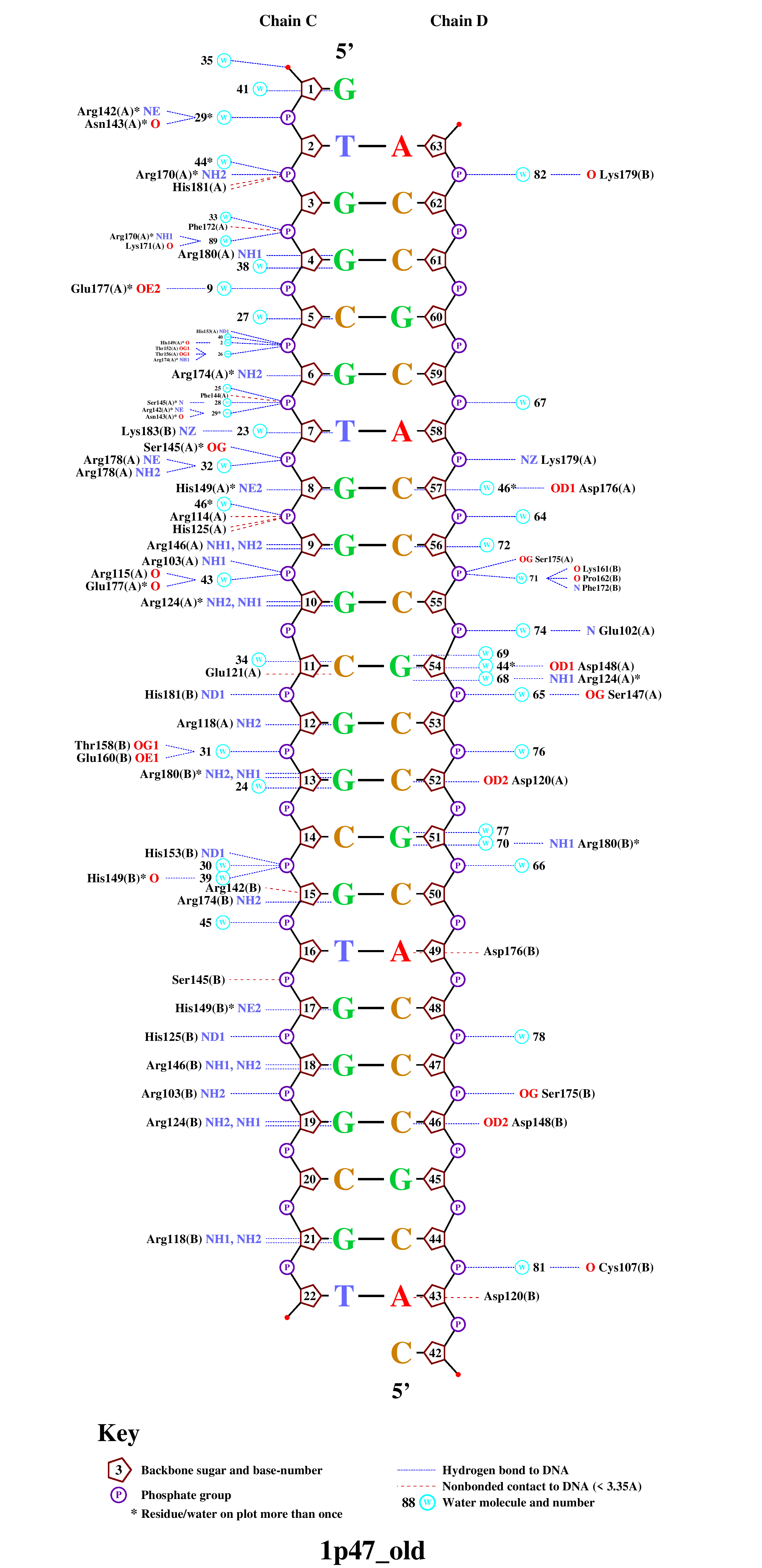
Для получения схемы ДНК–белковых контактов (ID 1P47) была использована консольная программа nuclplot. Изображение этой схемы на рисунке 3.
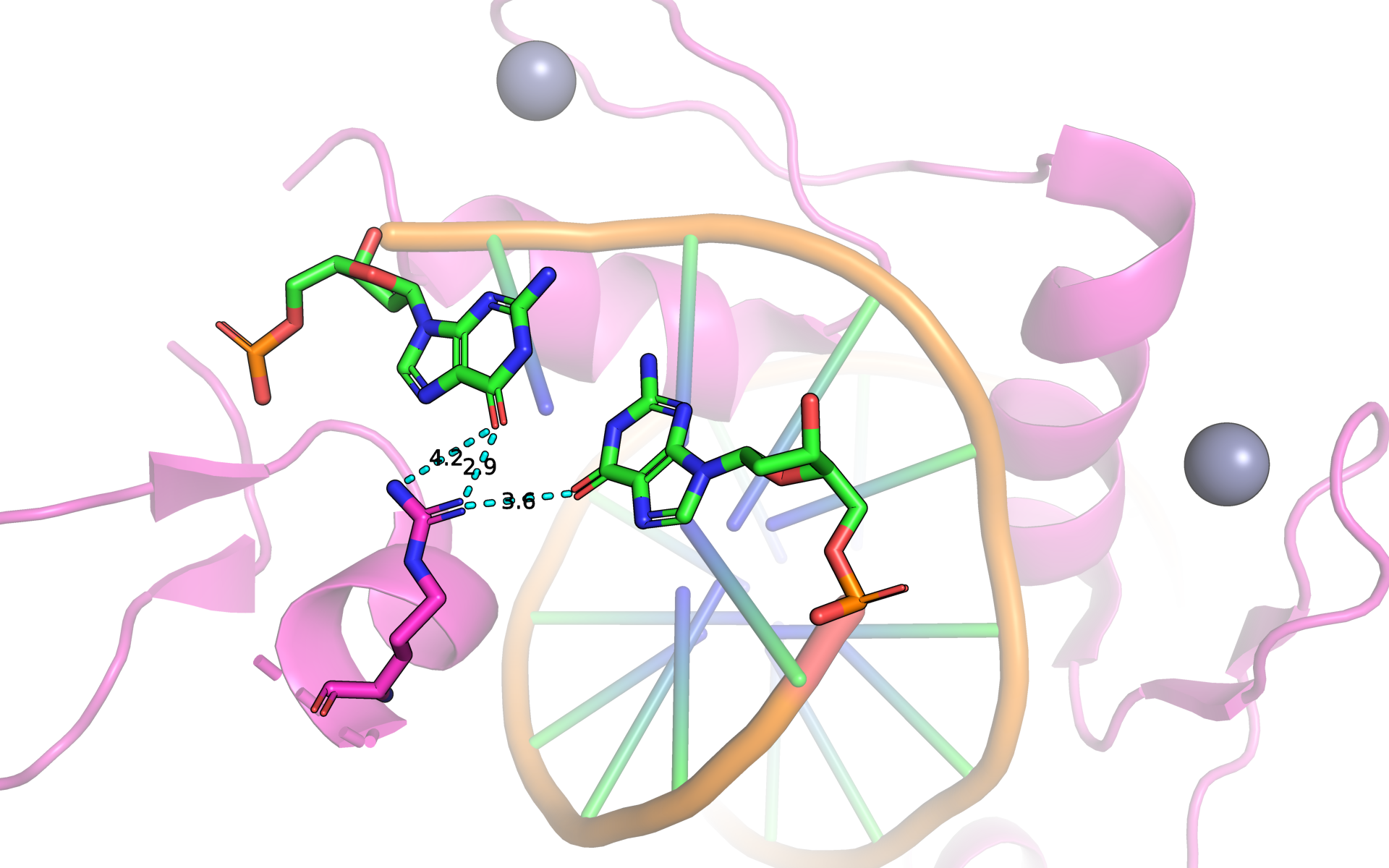
На схеме видно, что наибольшее количество контактов ДНК с белком у аминокислоты Arg124(A). Поэтому я считаю данную аминокислоту самой важной для распознавания. Дополнительно, данная аминокислота взаимодействует сразу с двумя азотистыми основаниями. Взаимодействие Arg124(A) с G10 и G54 можно увидеть на рисунке 4.
Дополнительные материалы
| Участок структуры | find-pair | einverted | RNAfold |
|---|---|---|---|
| Акцепторный стебель |
5'-902-907-3' 5'-966-972-3' 6 пар |
9 пар | 7 верных пар, 1 лишняя |
| D-стебель | 5'-910-912-3' 5'-923-925-3' 3 пары |
— | 3 верных пары 2 не хватает |
| T-стебель | 5'-961-965-3' 5'-949-953-3' 5 пар |
— | 4 верных пары, 1 лишняя и одной не хватает |
| Антикодоновый стебель |
5'-926-932-3' 5'-937-944-3' 7 пар |
— | 5 верных пар (канонических) |
| Общее число канонических пар нуклеотидов |
11 | 9 | 19 |
| Контакты атомов белка с... | Полярные | Неполярные | Всего |
|---|---|---|---|
| ...остатками 2'–дезоксирибозы |
3 | 13 | 16 |
| ...остатками фосфорной кислоты |
28 | 0 | 28 |
| ...остатками азотистых оснований со стороны большой бороздки |
16 | 7 | 0 |
| ...остатками азотистых оснований со стороны малой бороздки |
0 | 1 | 1 |