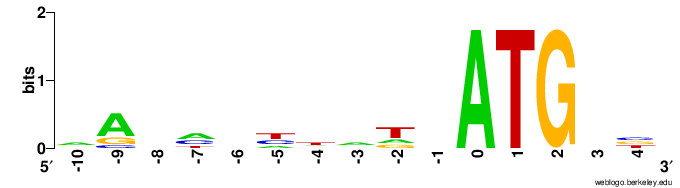
Нужно вычислить IC (информационное содержание, information content) последовательностей* Козак в геноме Danio rerio и создать его LOGO.
*Выравнивание (вариант 7)
cagcatgac
aaacatggg
caaaatggt
caacatgtc
aaccatgga
gacaatggc
aattatggc
gaatatggc
gaagatgga
caagatgtc
cagcatgtc
aatcatgga
IC вычисляется по следующим формулам:
(1) IC(b, j) = f(b, j) * log2(f(b, j) / p(b))
(дополнительное условие: если f(b, j) = 0, то IC(b, j) = 0)
(2) IC(j) = ΣbasesIC(b, j)
(3) IC = ΣpositionsIC(j)
| IC(b, j) | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 |
|---|---|---|---|---|---|---|---|---|---|
| A | 0.039576 | 1.703689 | 0.351845 | -0.146879 | 1.703689 | 0.000000 | 0.000000 | -0.156773 | -0.074078 |
| C | 0.462622 | 0.000000 | -0.035273 | 0.686664 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.930836 |
| G | 0.093332 | 0.000000 | -0.035273 | -0.035273 | 0.000000 | 0.000000 | 2.373327 | 1.192243 | -0.100970 |
| T | 0.000000 | 0.000000 | -0.146879 | -0.146879 | 0.000000 | 1.703689 | 0.000000 | -0.074078 | -0.156773 |
| IC(j) | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 |
|---|---|---|---|---|---|---|---|---|---|
| A+C+G+T | 0.595529 | 1.703689 | 0.134421 | 0.357633 | 1.703689 | 1.703689 | 2.373327 | 0.961393 | 0.599016 |
TOTAL IC: 10.132

Для обнаружения мотивов в геноме Magpie-robin coronavirus HKU18 был использован результат комманды из пакета MEME (а не с сайта, как в практикуме 6)
meme upstream_sequences.fasta -dna -nmotifs 3 -minw 6 -maxw -50
Отличие от предыдущего практикума состоит в том, что был захвачен более длинный upstream регион (для поздних белков: -200, в стандартном случае получить нормальный результат FIMO трудно)
Ссылка на результат работы FIMO fimo.txt
Для создания LOGO последовательности Козак были использованы последовательности с координатами от -10 до 4 для всех генов.

Как можно заключить из следующего изображения, последовательность Козак вируса почти не совпадает с человеческой
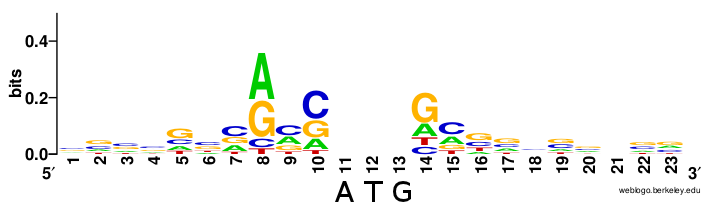
К сожалению, для доказательства специфичности не нашлось секвенированных геномов вирусов такого же вида, но отличных по штамму. Зато было проведено сравнение мотивов с мотивами Bulbul coronavirus HKU11-934. Можно сказать, что схожие (возможно CS) последовательности есть (ACACCA), хотя они в достаточной мере вариабельны как в геноме изучаемого мною вируса, так и в геноме его родственника (хотя большое сходство есть, даже upstream координаты близки и примерно равны -170 нуклеотидов)
Ссылка на результат работы FIMO relative_fimo.txt
| Номер | Наименование гена | Белковый продукт | Координаты |
|---|---|---|---|
| 1 | orf1ab | replicase polyprotein | join(596..11434,11434..19356) |
| 2 | S | spike glycoprotein | 19338..22991 |
| 3 | E | envelope protein | 22985..23233 |
| 4 | M | membrane protein | 23226..23882 |
| 5 | NS6 | NS6 protein | 23882..24172 |
| 6 | N | nucleocapsid protein | 24355..25395 |
| 7 | NS7a | NS7a protein | 25407..25580 |
| 8 | NS7b | NS7b protein | 25561..25932 |
| 9 | NS7c | NS7c protein | 25941..26195 |