Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков создали таблицу с интересующими нас параметрами. Медианы посчитали с помощью Excel. Cсылка на файл с расчетами.
Таблица 1. Описание трансмембранных белков с известной 3D структурой
| PDB код | Тип | Какая мембрана (внутренняя или внешняя, организм, органелла) | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 3ohn | баррель | Внешняя мембрана грам-отрицательной бактерии (Escherichia coli) | 21.2 ± 1.1 A* | 8 |
| 2jk4 | баррель | Внешняя митохондриальная мембрана (Homo sapiens) | 23.4 ± 2.3 A* | 8 |
| 1uun | баррель | Внешняя мембрана грам-положительной бактерии (Mycobacterium smegmatis) | 40.7 ± 2.1 | 9,5 |
| 2m3g | спираль | Внутренняя мембрана грам-отрицательной бактерии (Nostoc sp.) | 31.8 ± 0.8 A* | 18 |
| 1zza | спираль | Внешняя митохондриальная мембрана (Homo sapiens) | 30.0 ± 2.0 A* | 19 |
| 4h1w | спираль | Мембрана эндоплазматического ретикулума (Oryctolagus cuniculus) | 27.6 ± 1.0 A* | 19 |
Отбор гомологов
Составили небольшую репрезентативную выборку из гомологов с помощью blastp с порогом E-value = 0.00001. Провели несколько поисков: 1)исключили Enterobacteriales из результатов, 2) искали по эукариотам (выбрано 4 предсказанных белка), 3) искали по грам-положительным бактериям, 4) исключили протеобактерии. Ссылка на файл с отредактированными названиями (название организма и идентификатор белка).Анализ структуры выданного белка
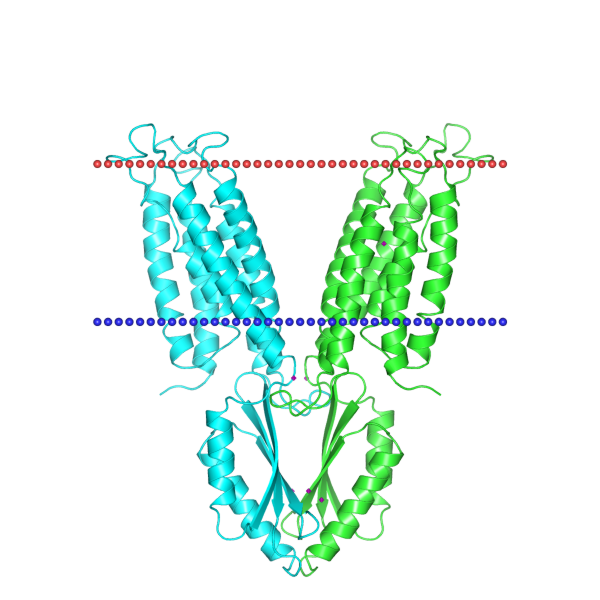
Рисунок 1. Ориентация белка в мембране по OPM (изображение взято с сайта OPM)
В кристаллической структуре белка 4 одинаковых цепи, цепи А и С являются трансмембранными, В и D - артефактные структуры. Рассматриваем цепь А.
Таблица 2.Описание структуры трансмембранного белка Ferrous-iron efflux pump FieF (идентификатор PDB 3H90, цепь A).
PDB ID |
Организм |
Тип мембраны |
TC-код |
Угол наклона спиралей (бета-тяжей) к нормали |
Количество трансмембранных спиралей (бета-тяжей в бочонке) |
3H90 |
Escherichia coli |
Бактериальная (Грам-отрицательная) внутренняя мембрана |
2.A.4.7.1 |
18° |
6 (2 трансмембранные субъединицы - цепи A и C, по 6 в каждой) |
TC система является аналогом ферментной системы EC.
- Значения полей TC-кода (2.A.4.7.1):
- 2: Транспортеры, управляемые электрохимическим потенциалом;
- 2.A: Различные портеры (uniporters, symporters, antiporters);
- 2.A.4: Семейство белков, обеспечивающих диффузию катионов(CDF);
- 2.A.4.7.1: Zn2+/Cd2+/Hg2+/Fe2+:H+ антипортеры.
Обобщенная реакция переноса для членов семьи CDF: Me2+ (in) H+ (out) ± K+ (out) = Me2+ (out) H+ (in) ± K+ (in).
Структура, полученная с разрешением 3.8 ангстрема, показывает гомодимер, взаимосвязанный в цитоплазматическом домене с помощью 4 атомов цинка.
Анализ множественного выравнивания трансмембранных белков
Последовательности были выравнены с помощью программы Muscle со стандартными настройками.
К выравниванию добавили дополнительные аннотации положения трансмембранных спиралей:
1)TM_REAL - по исходному белку с известной структурой (привязали структуру 3H90 к последовательности) отметили трансмембранные участки цепи А;
2)TM_PREDICTED - по белку Klebsiella_sp предсказали трансмембранные спиралис помощью программы TMHMM.
Окрасили по цветовой схеме Hydrophobicity (by conversation = 10%, интенсивность цвета зависит от того, насколько позиция консервативна).
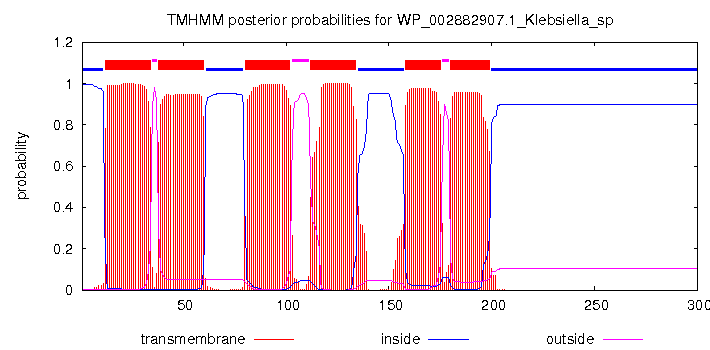
Рисунок 2. Предсказанная TMHMM структура белка из Klebsiella.
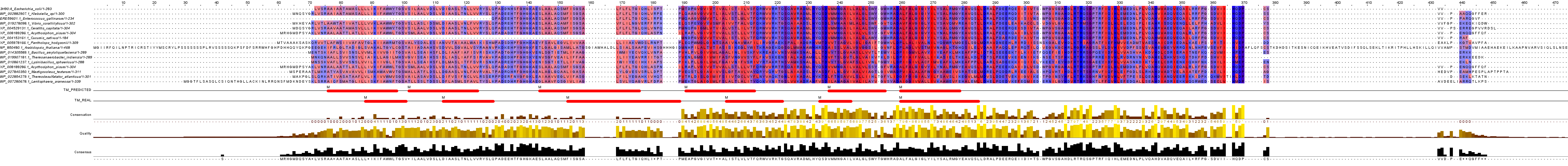
Рисунок 3. Выравнивание гомологов 3H90, окраска по гидрофобности, интенсивность окраски зависит от консервативности позиции (10%). Трансмембранные участки отмечены буквой "M" в дополнительных аннотациях TM_REAL и TM_PREDICTED.
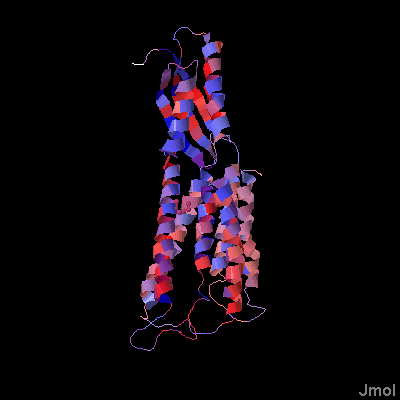
Рисунок 4.Трехмерная структура цепи А белка 3H90, часть белка ориентированая в n-сторону мембраны (в сторону цитоплазмы) - сверху, а ориентированная в p-сторону - снизу.
Результаты:
1) Участки, относящиеся к трансмембранным спиралям, относительно консервативны. В них часто встречаются гидрофобные аминокислотные остатки (лейцин, изолейцин, валин, реже - аланин).
2) Участки между спиралями тоже довольно консервативны.
3) В трансмембранных спиралях иногда встречаются достаточно консервативные заряженные остатки (аргинин) и полярные (глутамин), но большая часть действительно консервативных позиций (гистидин и аспаргиновая кислота) приходятся на участки вне мембраны.
4) Программа предсказала такое же количество трансмембранных участков. Реальные и предсказанные структуры приходятся на почти одинаковые позиции с небольшим сдвигом. Полагаю, что такие различия незначительны для разных таксонов и предсказание можно считать точным.
