Я выбрала бактерию Cyanothece sp. PCC 8801, которая содержит одну кольцевую хромосому (идентификатор NCBI: NC_011726) и три плазмиды. Из базы данных NCBI я скачала аннотацию в формате genbank (sequence.gb) и последовательность в формате fasta (sequence.fasta). Taxonomy ID: 41431. Полная таксономия: cellular organisms; Bacteria; Cyanobacteria; Oscillatoriophycideae; Chroococcales; Cyanothece; Cyanothece sp. PCC 8801.
Для аннотирования последовательности хромосомы бактерии Cyanothece sp. PCC 8801 я использовала сервер RAST. После регистрации и получения письма с подтверждением и паролем на почту я создала новое задание (Your Jobs => Upload New Job). Затем я ввела необходимые данные по таксономии о нужной мне последовательности, прикрепила файл с последовательностью в формате fasta и через некоторое время получила результат. Выдача программы дает большое количество информации о последовательности хромосомы Cyanothece sp. PCC 8801. Например, на рис.1 представлена графическая информация о функциональном распределении генов, закодированных в хромосоме. Также в выдаче есть файл с аннотацией хромосомы в формате GenBank и таблица Excel с информацией о локализации генов.
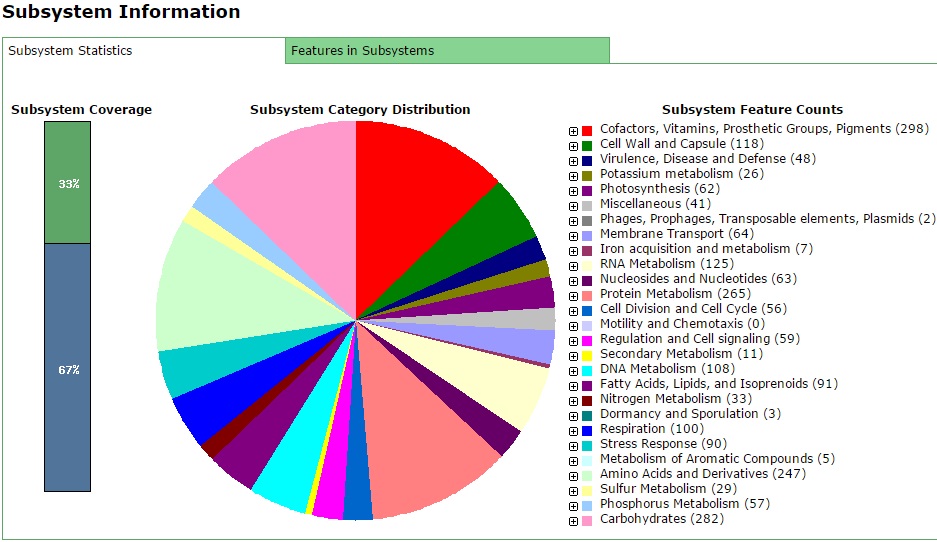
Рис.1 Функциональное распределение генов, закодированных в хромосоме бактерии Cyanothece sp. PCC 8801.
Из практикума "EMBOSS" я взяла таблицу аннотированных генов белков в формате Excel из записи GenBank: GenBank.xlsx. Затем я обработала таблицу Excel с информацией о локализации генов, полученную с помощью RAST. Объединив обе таблицы я получила файл G&R.xlsx.
Используя функцию ЕСЛИ, я получила следующие данные:
- число одинаково аннотированных генов - совпадают и старт, и стоп-кодоны: 3626
- число генов с одинаковым стоп-кодоном, но разными старт-кодонами: 294
- число генов с одинаковым старт-кодоном, но разными стоп-кодонами: 281
- число генов, аннотированных RAST, и не аннотированных в записи GenBank: 617
- число генов, аннотированных в записи GenBank, и не аннотированных в записи RAST: 221
Для трех генов с несовпадающими аннотациями я проверила аннотацию с помощью blastp.
У первого гена не совпадает старт-кодон, однако стоп-кодон совпадает. Так как белок длиннее в аннотации GenBank на две аминокислоты, я проверила его последовательность с помощью blastp. Первые две аминокислоты (MV) не встречаются ни в одной из находок, но вместо них часто стоит LL, что говорит о том, что, возможно, можно считать аннотацию GenBank более правильной.
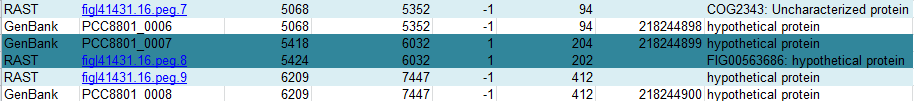
Рис.2 Ген с несовпадающими старт-кодонами.
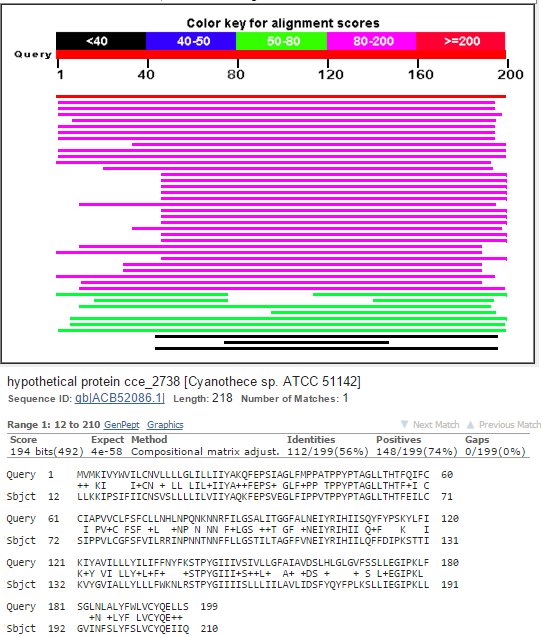
Рис.3 Фрагмент выдачи blastp.
У второго гена не совпадает стоп-кодон, но старт-кодон совпадает. Я проверила последовательности бека из аннотации RAST и GenBank с помощью blastp. Результат представлен на рис.5. К последовательности из RAST на найдено ни одного гомолога, что странно. Но тем не менее, скорее всего, аннотация GenBank более корректная, так как хотя бы 2 гомологичные поседовательности были найдены.
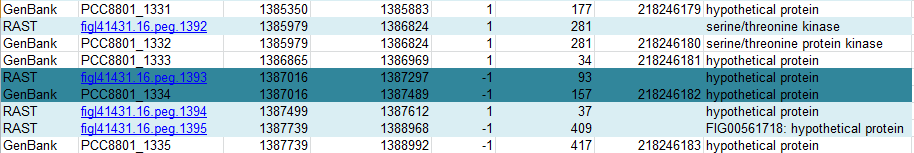
Рис.4 Ген с несовпадающими стоп-кодонами.
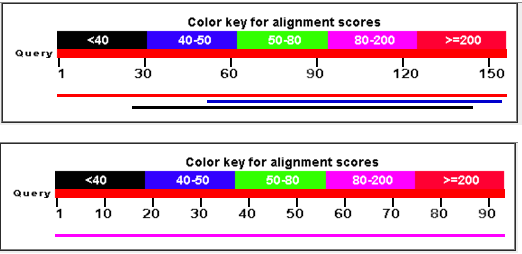
Рис.5 Фрагмент выдачи blastp. Сверху последовательность GenBank, снизу RAST.
У третьего гена не совпадает стоп-кодон, но старт-кодон совпадает. Я проверила последовательности бека из аннотации RAST и GenBank с помощью blastp. Результат представлен на рис.7. К последовательности из RAST не найдено ни одного гомолога, который захватывал бы последовательность перед старт-кодоном GenBank, что говорит о том, что аннотация GenBank более верная.
Рис.6 Ген с несовпадающими стоп-кодонами.
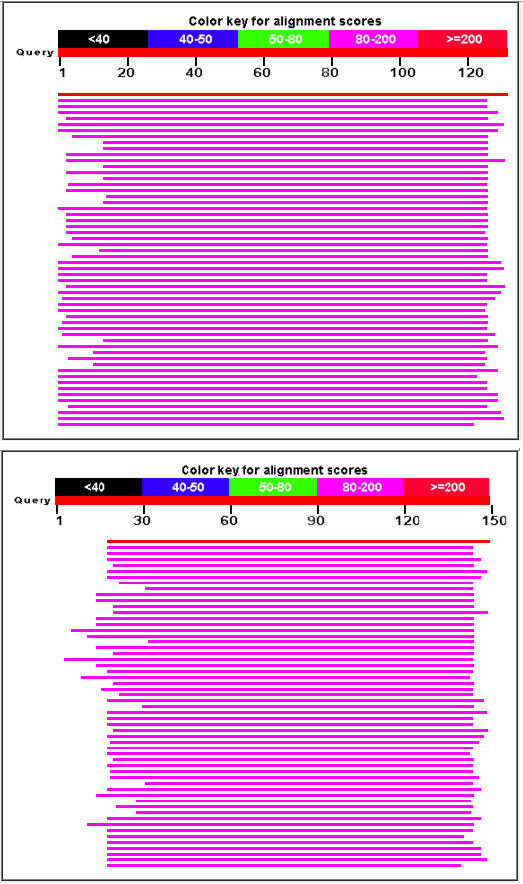
Рис.7 Фрагмент выдачи blastp. Сверху последовательность GenBank, снизу RAST.
.jpg)