Для выполнения данного задания был выбран домен RMMBL (AC в Pfam: PF07521), Zn-зависимый металло - гидролазный РНК специфичный домен из белка beta-lactamase domain-containing protein. Данный домен имеется в 3260 известных белках и встречается в составе 46 архитектур. Для дальнейшей работы было выбрано 2 архитектуры, представленные на рисунках 1 и 2. Все виды архитектур представлены по ссылке.
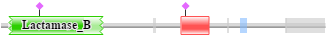
Рис. 1 Одна из доменных архитектур белков (W5USJ3_9MOLU), содержащих домен RMMBL. Изображение получено из базы данных Pfam.
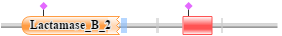
Рис. 2 Одна из доменных архитектур белков (K9RFM7_9CYAN), содержащих домен RMMBL. Изображение получено из базы данных Pfam.
Архитектура W5USJ3_9MOLU встречается в 1098 последовательностях. В ее состав входят 2 домена: Lactamase_B (AC в Pfam: PF00753), RMMBL. Домен Lactamase_B содержится в белках класса бета-лактамаз и в ряде других белков. Металло-бета-лактамазы являются важными ферментами, поскольку они участвуют в разрушении антибиотиков у устойчивых к антибиотикам бактерий.
Архитектура K9RFM7_9CYAN встречается в 274 последовательностях. В ее состав входят 2 домена: Lactamase_B_2 (AC в Pfam: PF12706), RMMBL. Домен Lactamase_B_2 является частью бета-лактамазного надсемейства.
Было скачано выравнивание из Pfam (Full) в JalView для данного домена и раскрашено по консервативности с порогом 5% ClustalX. Проект в JalView: PF07521_full.jar, PF07521_full.fasta.
Затем была получена таблица с информацией об архитектуре всех последовательностей, содержащих выбранный домен, из файла swisspfam, содержащий эту информацию для всех последовательностей Uniprot при помощи скрипта swisspfam_to_xls.py, который отбирает последовательности с указанным доменом и составляет таблицу для Excel. Полученная таблица: DOMAINS_INFO.xlsx. Из этой таблицы была построена сводная таблица в Excel: строки – AC последовательностей, столбцы – домены Pfam. В список последовательностей были добавлены колонки с информацией о таксономической принадлежности и о длине выбранного домена из каждой последовательности. Сводная таблица приведена на Листе2 в DOMAINS_INFO.xls.
Был выбран таксон Bacteria и два подтаксона Acidobacteria и Cyanobacteria. Выбранный однобуквенный код для Acidobacteria - А, для Cyanobacteria - С. Из доменных архитектур K9RFM7_9CYAN (Лист3) и W5USJ3_9MOLU (Лист4) было выбрано по 20 последовательностей, отмеченных в таблице буквами А и С соотвественно подтаксонам.
Выравнивание отобранных последовательностей домена, разбитое на группы по доменным архитектурам и раскрашенное по консервативности внутри групп: AC_alig.jar. Группа K9RFM7_9CYAN расскрашена ClustalX с порогом консервативности 10%, а группа W5USJ3_9MOLU - 15%.
В целях улучшения выравнивания были удалены N- и C-концевые участки и следующие плохо выровненные на общем фоне последовательности: A0A073CJ81, A0A089Y308, D5E7B0.
Полученное выравнивание правильное, так как оно состоит из консервативных блоков, в которых позиции довольно консервативны.
Построение филогенетического дерева последовательностей доменаИз полученного в предыдущем задании выравнивания был выделен блок, содаржащий общий домен RMMBL (AC в Pfam: PF07521). К именам всех последовательностей спереди были добавлены коды архитектуры и таксона. Код архитектуры: 1 - (K9RFM7_9CYAN), 2 - (W5USJ3_9MOLU). Код таксона: A - Acidobacteria, C - Cyanobacteria.
На основании полученного выравнивания было построено филогенетическое дерево последовательностей домена. Для построения использовался метод "Neighbor Joining Using % Identity" в программе MEGA. Изображение дерева представлено на рисунке 3. Дерево в Newick-формате (скобочная формула): tree.nwk.
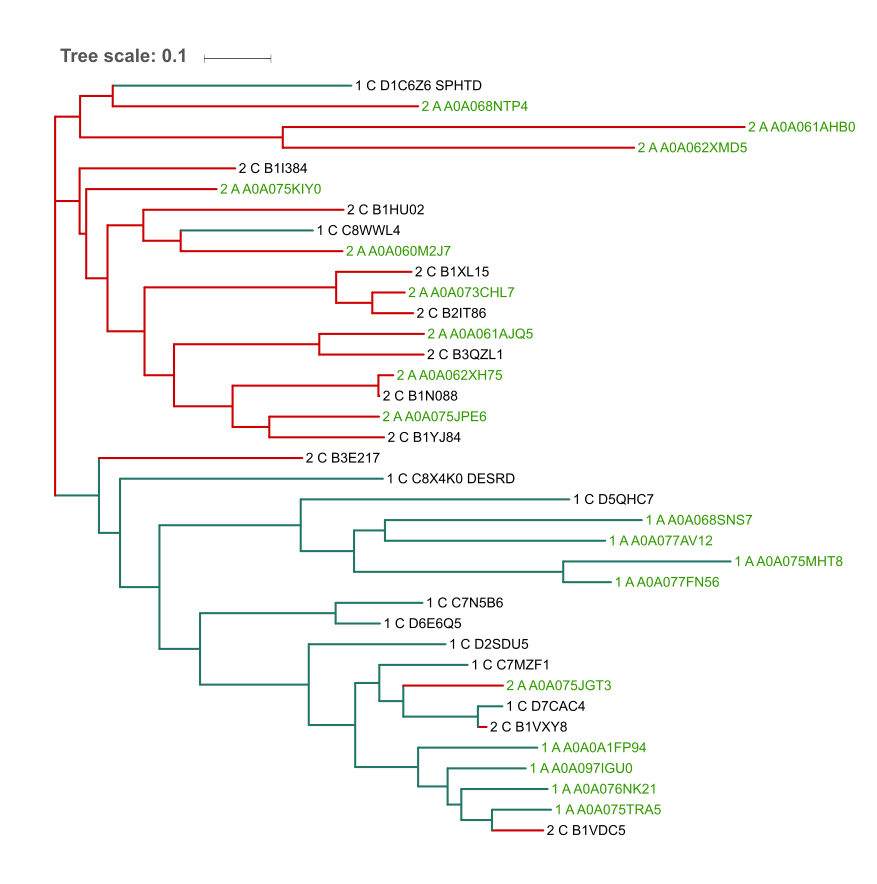
Рис. 3 Изображение дерева, полученное из выравнивания доменов RMMBL (AC в Pfam: PF07521) из белков с разной доменной архитектурой: K9RFM7_9CYAN (зеленые ветви на дереве), W5USJ3_9MOLU (красные ветви). Зеленым цетом отмечены названияя последовательностей из таксона Acidobacteria, черным - из таксона Cyanobacteria. Изображение получено с помощью Itol.
Из рисунка 3 видно, что белки, имеющие одинаковую доменную архитектуру, за редким исключением образуют отдельные друг от друга клады. Такое разделение не может быть связано с ошибками построения выравнивания, так как вырванивание и внутри архитектур, и всех последовательностей в целом достаточно хорошее, последовательности гомологичны, а плохо выровненные последовательности были удалены. Такое разделение можно объяснить тем, что, возможно, был общий предок для этих двух доменных архитектур, который имел один общий домен RMMBL, а затем часть белков приобрела домен Lactamase_B_2, а другая часть Lactamase_B, вследствие чего белки стали отдаляться друг от друга в эволюционном плане. Существование небольшого количества белков, которые встретились в массе белков с другой доменной архитектурой, можно объяснить тем, что эти белки либо имеют низкий уровень мутаций в гене данного белка, либо отделились на более позднем этапе эволюции, и их домены еще не успели достаточно разойтись при независимой эволюции.
Однако на рисунке нельзя выделить отдельных клад, образованных белками из одного подтаксона. Распределение подтаксонов Acidobacteria и Cyanobacteria кажется случайным. Скорее всего, белки являются консервативными для всех групп бактерий.
Построение профиля подсемейства и характеристика качества его работыДля построения профиля были выделены последовательности, представленные на рисунке 4. Дерево неукорено, поэтому нижняя ветка дерева может относиться в выбранной кладе. Выравнивание выбранных последовательностей: prof.fasta.
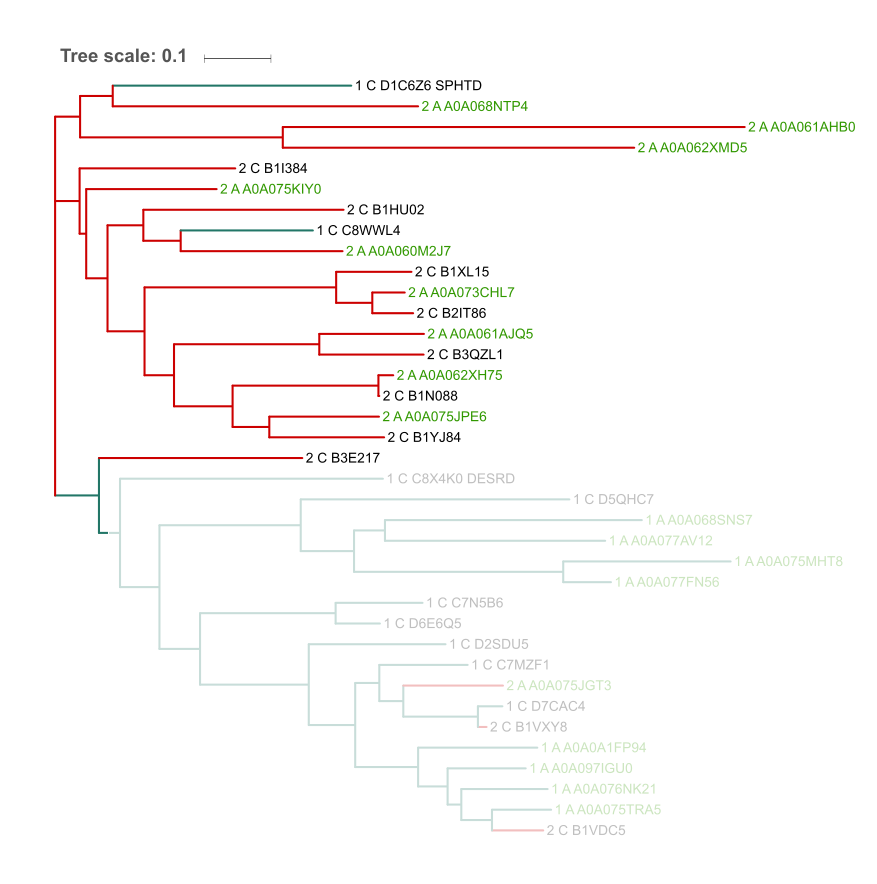
Рис. 4 Подсемейство, выбранное для построения профиля. Изображение дерева, полученное из выравнивания доменов RMMBL (AC в Pfam: PF07521).
С помощью пакета HMMER (hmm2build - hmm2build hmm.out prof.fasta и hmm2calibrate - hmm2calibrate hmm.out) была получен профиль для выбранных последовательностей: hmm.out. С помощью программы hmm2search (hmm2search hmm.out PF07521_full.fasta > hmm2search.out) с использованием полученного профиля был проведен поиск по всем белкам UniProt, содержащим домен RMMBL. В результате был получен следующий файл: hmm2search.out.
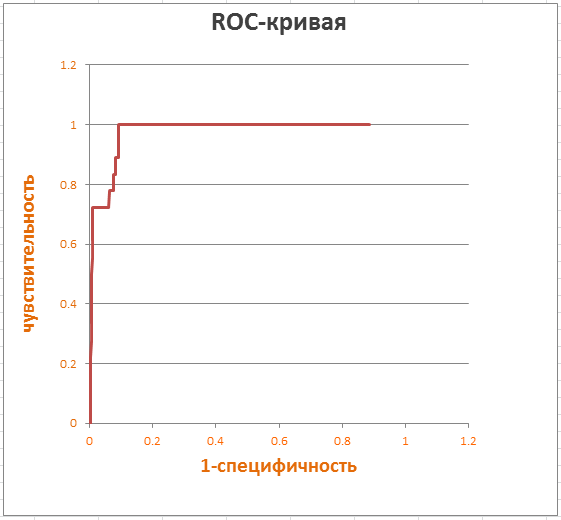
Рис. 5 ROC-кривая.
Таблица 1. Значения при выбранном пороге
| На самом деле | принадлежит подсемейству | не принадлежит | сумма |
| Выше порога по профилю | 14 | 45 | 59 |
| Ниже порога по профилю | 5 | 2907 | 2912 |
| Сумма | 19 | 2952 | 2971 |
.jpg)