Учебный Сайт Николая Николаева
Назад на страницу семестра
Выравнивание геномов.
1. Выбор 3-5 штаммов одного вида бактерий или архей с известными полностью собранными геномами.
Файл genomes.tsv с информацией о выбранных геномах E. coli.2. Построение нуклеотидного пангенома.
После выполнения npge Examine параметр MIN_IDENTITY был установлен на 0.899.log-файлы, содержащие информацию о работе программ: prepare_log; makepangenome_log.
3. Описание стабильного ядра пангенома.
- Число блоков (s-blocks): 354;
- Размер ядра в процентах от совокупности всех геномов: 82.75%;
- Процент консервативных колонок в объединённом выравнивании s-блоков: 99.96%.
4. Описание делеций.
Файл pangenome.bi был импортирован в Excel. Данные были отсортированы по возрастантю количества фрагментов и затем по убыванию длины.
В Табл. 1 приведена информация о некоторых блоках, отсутствующих только у одного штамма. За полную длину делеции считается длина подтверждающего блока и всех соседних с ним блоков, отсутствующих тоько у данного штамма.
| Штамм | 97-3250 | FORC-028 | 2015C-4944 | E2865 | MBT-5 |
|---|---|---|---|---|---|
| Блок, подтверждающий делецию | h4x3658 | h4x1560 | h4x8715 | h4x876 | h4x1926 |
| Полная длина делеции, п.н. | 7438 | 1560 | 23467 | 1871 | 37332 |
| Гены | Фаговый большой белок капсида | тРНК-циклоцитидин синтетаза TtcA (фрамент) | Дельта-подобный белок из семейства белков с цинковым пальцем | Белок группы Rhs RhsC | Фаговый белок каркаса капсида; Белок хвоста фага |
5. Перестановка синтений.
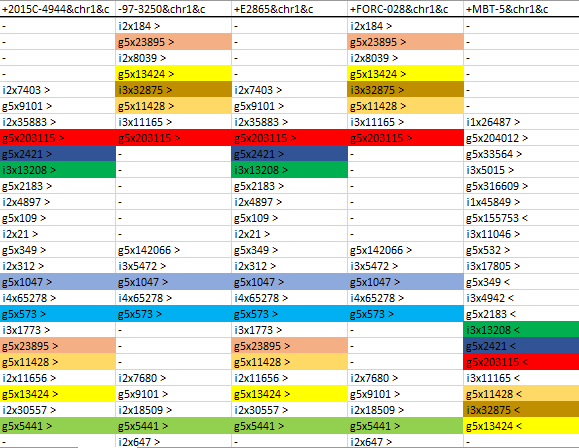
Данные о положении блоков (blocks.blocks) были импортиованы в Excel и транспонированы.
Интересно положение блоков в позициях 32-56 (Рис. 1). Во-первых, видно, что у штаммов 97-3250 и FORC-028 отсутствуют блоки, имеющиеся у остальных трёх штаммов, например, i3x13208. Во-вторых, последовательности блоков g5x13424-i3x32875-g5x11428 и g5x203115-g5x2421-i3x13208 в MBT-5 инвертированы по сравнению с остальными штаммами и находятся рядом.
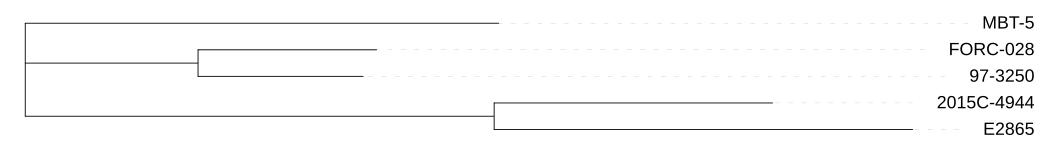
Скорее всего, предковое состояние не имело этой инверсии. Тогда, с учётом филогенетического дерева (Рис. 2), у ветви 2015C-4944 - E2865 должно было произойти перемещение блоков g5x23895, g5x13424 и g5x11428, а ветвь 97-3250 - FORC-028 потеряла многие блоки, такие, как i3x13208.
6. Ошибки аннотации генов.
Для поиска ошибок в аннотации генов был взят s-блок с наибольшим их количеством- s5x117812. Некоторые примеры неаннотированных белков и их положение в блоке (названия для MBT-5):
- adenine-specific DNA-methyltransferase (7988-8872)
- DNA-binding transcriptional regulator Fis (8958-9254)
- tRNA dihydrouridine synthase DusB (9280-10245)
Во всех трёх случаях ген аннотирован у MBT-5, FORC-028 и E2865, и отсутствует у 2015C-4944 и 97-3250, в то время как соответствующие последовательности идентичны на 100.0% или около 100% с сохранением старт- и стоп-кодонов.
В последнем случае кодирующая последовательность у FORC-028 короче, чем у MBT-5 и E2865 за счёт другого старт-кодона (Рис.3). Ни перед одним из двух возможных старт-кодонов нет выраженной последовательности Шайна-Дальгарно. Поскольку большая рамка кодирует 321 аминокислоту, а меньшая - 281, а в UniProt среди белков, подтверждённых на уровне транскрипта или белка, присутствует запись лишь о белке длиной 321 аминокислотный остаток, достовернее всего выглядит предположение, что старт-кодон аннотирован у FORC-028 неправильно.