Учебный Сайт Николая Николаева
Назад на страницу семестра
Сигналы и мотивы-2.
1. Поиск последовательности Шайна-Дальгарно в геноме Bradyrhizobium diazoefficiens.
Для поиска сигнала был выбран геном Bradyrhizobium diazoefficiens, белки которой ранее уже становились объектом рассмотрения в одном из прошлых практикумов.
Для выбора генов, перед которыми планируется искать сигнал, была скачана таблица из UniProt, содержащая информацию о генах всех белков B. diazoefficiens из Swiss-prot. В ней были оставлены только гены белков длиной не менее 300 аминокислоных остатков, о которых имеется больше информации, чем только предсказание ("Predicted"), имеющие хорошую аннотацию по оценке UniProt (Annotation score не менее 3 из 5).
Из Feature table хромосомы B. diazoefficiens были взяты координаты CDS генов, GeneID которых нашлись в вышеупомянутой таблице. По этим координатам были вырезаны 20-нуклеотидные участки хромосомы перед старт-кодонами (как с прямой, так и с обратной цепи) - всего 182 последовательности. Они были разбиты на 100 "тренировочных" для нахождения мотива с помощью MEME и 82 "тестовые" для нахождения того же мотива с помощью FIMO. К первым была добавлена 20-нуклеотидная посдедовательность, комплементарная 3'-концу 16s рРНК.
Последовательности для MEME;
Последовательности для FIMO.
Реализация вышеописанных действий в Python-скрипте доступна по ссылке.
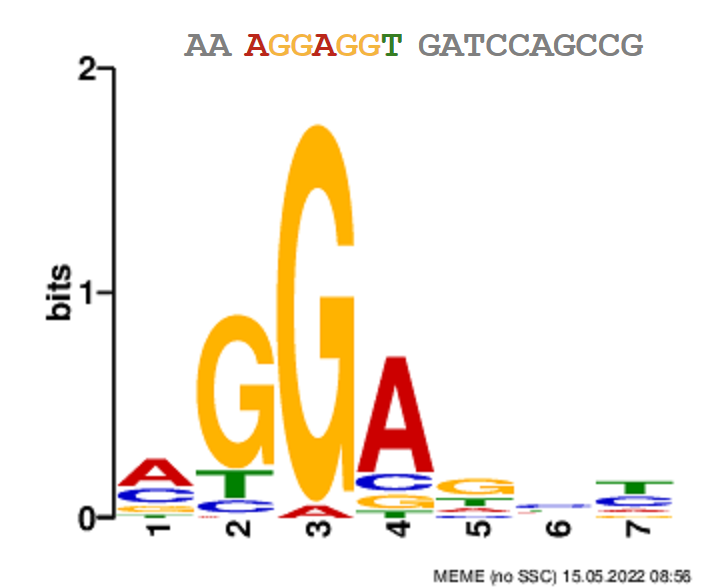
Нахождение мотива производилось в web-версии MEME Suite с поиском лишь на введёноной цепи мотивов длиной от 5 до 9 пар нуклеотидов. Поиск с разрешением отсутствия вхождения ("zoops") показал мотивы, присутствующие лишь в единичных последовательностях (не включающих рРНК) и имеющие довольно высокий E-value (не менее 2.25), поэтому результат не приводится в данном отчёте. С другой стороны, поиском с однократным вхождением мотива в каждую последовательность ("oops") был получен мотив, очень похожий на консенсус Шайна-Дальгарно и почти полностью соответствующий последовательности 16s рРНК (за исключением самой малоинформативной 6-й колонки из 7-ми; см. рис. 1). E-value при этом увеличился (3.18), что может быть ожидаемо при принудительном рассмотрении всех последовательностей.
2. Проверка PWM.
Результат работы MEME был использован для поиска мотива в "тестовых" последовательностях с помощью FIMO. Поиск производился только на введённой цепи с порогом p-value<0.01 (поиск с порогом по умолчанию p-value<10-4 не дал ни одного результата).
Выдача FIMO в формате tsv доступна по ссылке.
Было найдено лишь 39 мотивов среди 82 последовательностей, при этом q-value слишком высок, чтобы считать предсказания достоверными (не менее 0.125). Для построения более точной PWM может требоваться провести проверку генов на предмет того, транслируются ли их мРНК с помощью Шайна-Дальгарно или иным путём.