Сигналы и мотивы-3
Задание 1.
Для выполнения этого задания была выбрана Acidovorax species, так как в ней Р-М типа II с известными сайтами рестрикции.
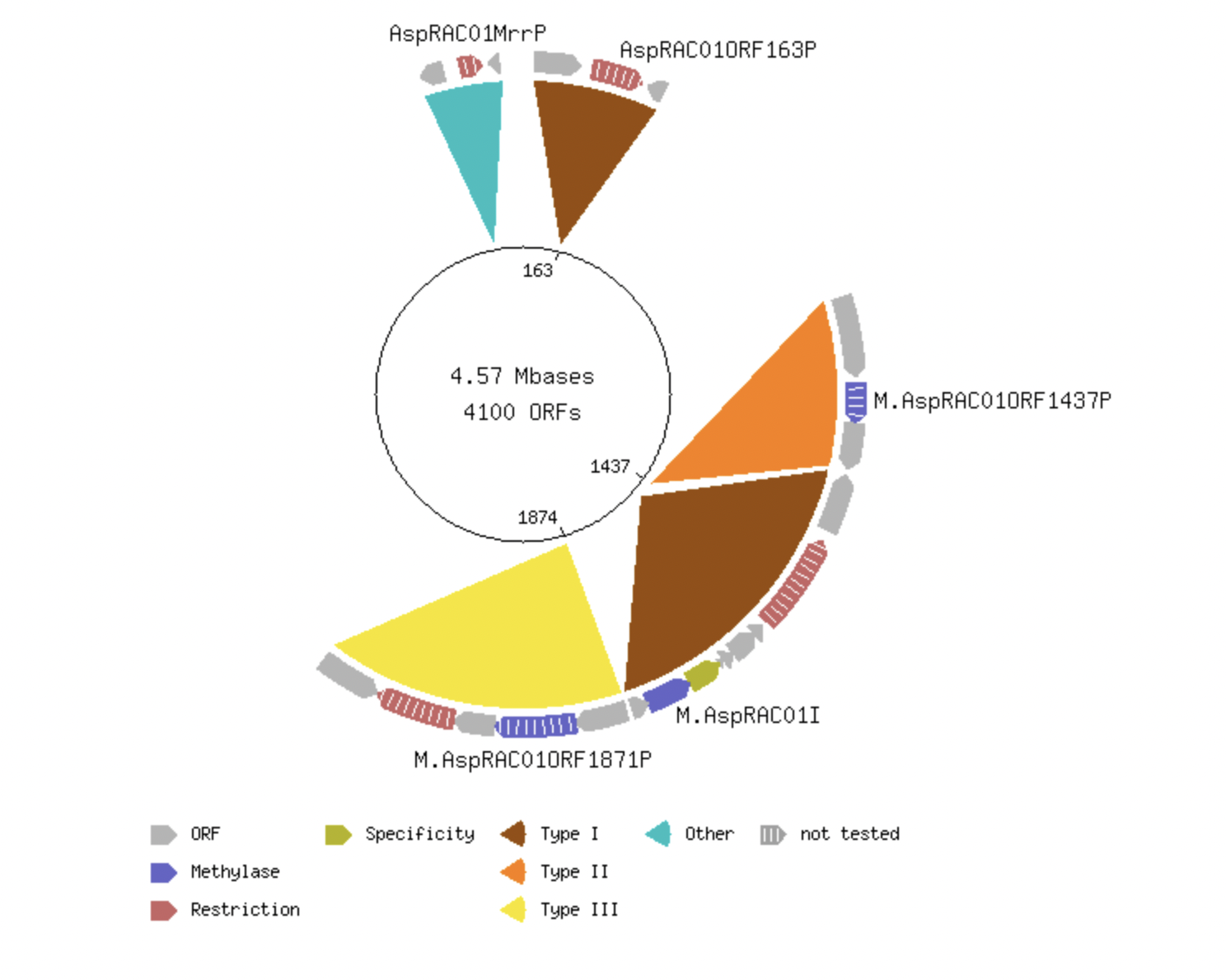
Затем геном выбранной бактерии был скачен из NCBI, он находится по ссылке.
Для того чтобы оценить представленности сайтов рестрикции в геноме выбранной бактерии была использована команда cbcalc: cbcalc -s Type2_REs.tsv -o out.tsv Acidovorax.fna. Результат
Далее полученный файл был отсортирован командой: sort -k5,5g out.tsv > out_sorted.tsv
Порог контраста был взят из условия задания, а именно 0.8: awk '$5 + 0 <= 0.8' out.tsv > result.tsv. Результат
Затем было необходимо узнать о рестриктазах, которые могут узнавать недопредставленные в геноме сайты: cut -f 5 Type2_REs.tsv |paste - Type2_REs.tsv |grep -E 'no'| cut -f 2-> final.txt.
Эта команда была необходима, потому что она выбрала из общего списка рестриктаз те, у которых в столбце Predicted записано no.
Как говорится: "Last but not least", задачей была отфильтровка списка рестриктаз на узнавание непредставленных сайтов. Это сделал скрипт Бетеньковой.Р
В итоговом файле осталось 95 записей о недопредставленных в геноме рестриктазах
Задание 2.
Был выбран идентификатор B2V8C0. Это белок MinC из бактерии рода Sulfurihydrogenibium sp., ингибитор деления клеток, блокирующий образование полярных Z-кольцевых перегородок.
| Таблица.1 | |||||
|---|---|---|---|---|---|
| Номер итерации | Число находок выше порога (0.005) | Идентификатор худшей находки выше порога | Е-value этой находки | Идентификатор лучшей находки ниже порога | Е-value этой находки |
| 1 | 163 | Q88M41.2 | 0,004 | Q4US07.1 | 0,006 |
| 2 | 188 | Q9ZM51.1 | 6E-07 | A7H8E6.1 | 0,037 |
| 3 | 188 | Q9ZM51.1 | 2E-11 | A7H8E6.1 | 0,025 |
Начиная со второй итерации, результат сошёлся. На всякий случай сделаем ещё одну итерацию, но результат был таким же. Полученные данные говорят о высокой консервативности белков, принадлежащих выбранному семейству.