Реконструкция филогенетических деревьев
Таксономия бактерий
Пользуясь таксономическим сервисом NCBI, определим, к каким таксонам
относятся отобранные в "Что такое филогенетическое дерево" бактерии.
Все они относятся к cellular organisms; Bacteria; Firmicutes, а дальнейшие различия в таксонах
представлены в табл.1
Таблица 1. Список бактерий отдела Firmicutes
Название |
Мнемоника |
Таксономия | |
Bacillus anthracis |
BACAN |
Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group | |
Bacillus subtilis |
BACSU |
Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group | |
Clostridium tetani |
CLOTE |
Clostridia; Clostridiales; Clostridiaceae; Clostridium | |
Finegoldia magna |
FINM2 |
Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia | |
Lactobacillus delbrueckii |
LACDA |
Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus | |
Listeria monocytogenes |
LISMO |
Bacilli; Bacillales; Listeriaceae; Listeria | |
Streptococcus pyogenes |
STRP1 |
Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
Теперь посмотрим есть ли на дереве отобранных нами бактерий ("Что такое филогенетическое дерево", рис.2) ветви, выделяющие какие-нибудь из таксонов. Для наглядности (рис.1), таксоны перенесем на филогенетическое дерево, полученное ранее.
(Увеличенное изображение при клике на картинке)
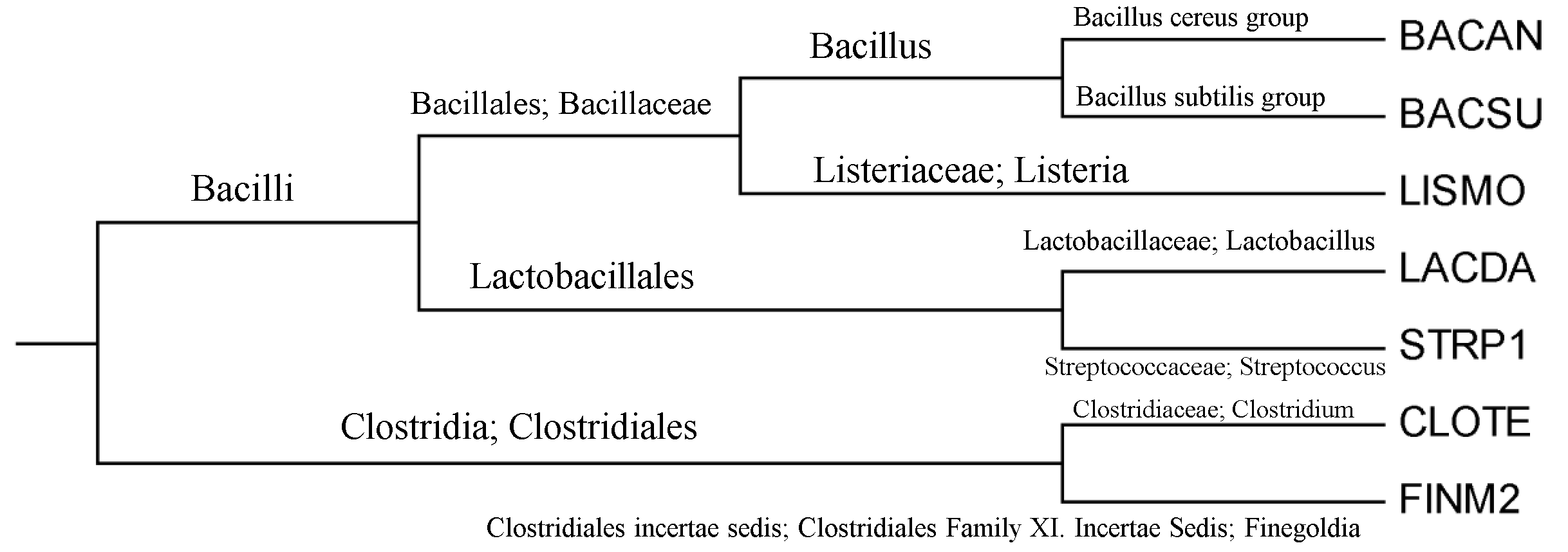
Реконструкция филогенетических деревьев
По белкам семейства HSLO будем реконструировать филогенетическое дерево.
Получим из Swiss-Prot последовательности белков шаперонинов из отобранных нами бактерий (табл.1).
Для этого зайдем в PuTTY и введем команду:
seqret sw:HSLO_*
,где * - это мнемоника из табл.1
Поместим полученные последовательности в один fasta-файл (hslo_bacteria.fasta)
и отредактируем названия последовательностей, оставив только мнемонику видов.
Создадим выравнивание отобранных белков, импортировав невыровненные последовательности в
JalView и воспользовавшись Web Service. Будем использовать сервис Muscle с дефолтными параметрами.
Теперь получим изображение выравнивания в "блочной" форме
(Format > Wrap в меню) с раскраской по проценту идентичности. Выравнивание приведено на рис.2.
Рис.2 Изображение выравнивания в "блочной" форме
(Увеличенное изображение при клике на картинке)

Реконструируем филогенетическое дерево четырьмя методами, доступными из JalView:
- Average Distance Using % Identity
- Neighbour Joining Using % Identity
- Average Distance Using BLOSUM62
- Neighbour Joining Using BLOSUM62
Каждое дерево сохраним в Newick-формате в файл с соответствующим методу выравнивания названием. Импортируем выравнивание (файл в fasta-формате) в программу Mega (при импорте выберем "Analyze"). Реконструируем дерево методом "Maximum Parsimony" (кнопка Phylogeny). Укореним дерево в ветвь (меню Subtree > Root). Полученное дерево для каждого выравнивания представлено на рис.3.
Рис.3 Реконструкция филогенетического дерева бактерий отдела Firmicutes
Ниже представлены 4 дерева, полученные 4 разными методами, доступными из JalView.
(Увеличенное изображение при клике на картинке)
Рис.3(a) Average Distance Using % Identity
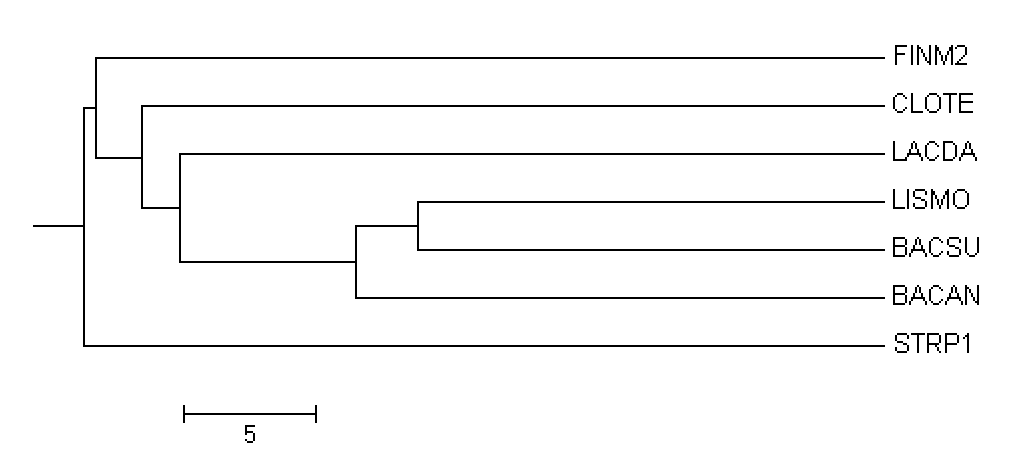
Рис.3(b) Neighbour Joining Using % Identity
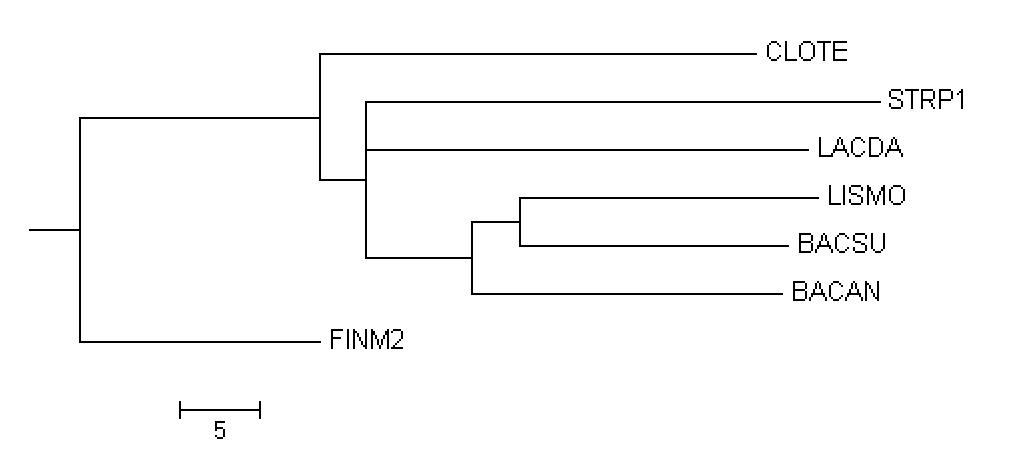
Рис.3(c) Average Distance Using BLOSUM62
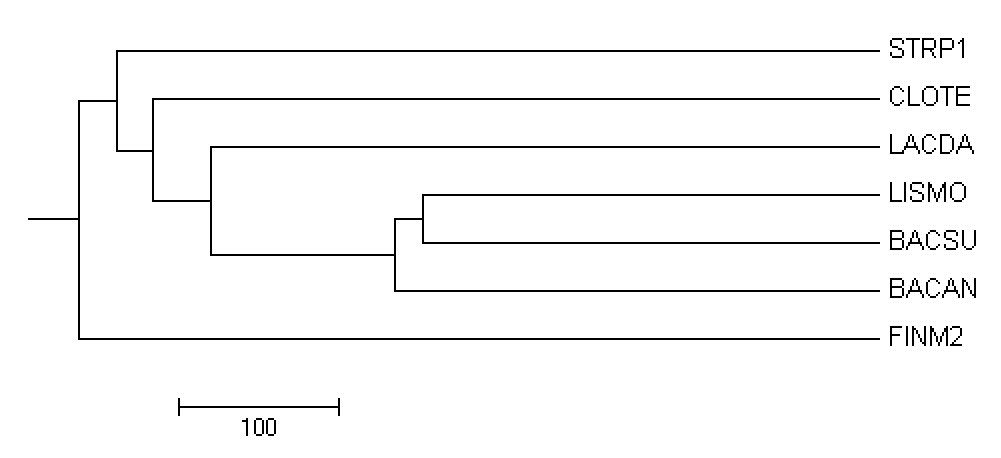
Рис.3(d) Neighbour Joining Using BLOSUM62
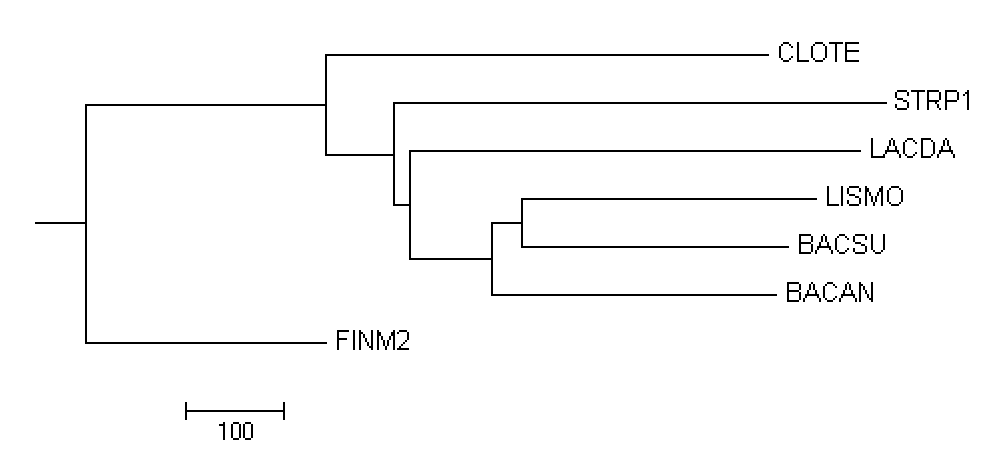
Сравним топологию полученных деревьев с топологией правильного дерева (рис.1.):
Курсивом отметим те ветви, которых нет в правильном дереве, но есть в реконструированных.
- Average Distance Using % Identity (Рис.3(a))
{BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1, BACAN}
{BACAN, BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1}
{BACAN, BACSU, LISMO, LACDA} против {CLOTE, FINM2, STRP1}
{BACAN, BACSU, LISMO, LACDA, CLOTE} против {FINM2, STRP1}
- Neighbour Joining Using % Identity (Рис.3(b))
{BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1, BACAN}
{BACAN, BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1}
{BACAN, BACSU, LISMO, LACDA} против {CLOTE, FINM2, STRP1}
{BACAN, BACSU, LISMO, STRP1} против {CLOTE, FINM2, LACDA}
{BACAN, BACSU, LISMO, STRP1, LACDA} против {CLOTE, FINM2}
- Average Distance Using BLOSUM62 (Рис.3(c))
{BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1, BACAN}
{BACAN, BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1}
{BACAN, BACSU, LISMO, LACDA} против {CLOTE, FINM2, STRP1}
{BACAN, BACSU, LISMO, LACDA, CLOTE} против {FINM2, STRP1}
- Neighbour Joining Using BLOSUM62 (Рис.3(d))
{BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1, BACAN}
{BACAN, BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1}
{BACAN, BACSU, LISMO, LACDA} против {CLOTE, FINM2, STRP1}
{BACAN, BACSU, LISMO, LACDA, STRP1} против {CLOTE, FINM2}
Реконструкция дерева из выравнивания белков в программе Mega
Импортируем выравнивание (файл в fasta-формате) в программу Mega (при импорте выберем "Analyze"). Реконструируируем дерево методом "Maximum Parsimony" (кнопка Phylogeny). Укореним дерево в наиболее правильную ветвь (меню Subtree > Root).
Рис.4 Реконструкция дерева из выравнивания белков в программе Mega
Укажем отличия от правильного дерева, представленного на рис.1.
Курсивом отметим те ветви, которых нет в правильном дереве, но есть в реконструированном.
Ветви тривиальные у реконструированного дерева (рис.4):
- {BACAN, BACSU, LISMO} против {CLOTE, FINM2, LACDA, STRP1}
- {BACSU, LISMO} против {BACAN, LACDA, STRP1, FINM2, CLOTE}
- {LACDA, STRP1} против {FINM2, BACAN, BACSU, LISMO, LACDA, CLOTE}
- {FINM2, LACDA, STRP1} против {BACAN, BACSU, LISMO,CLOTE}
Ветви тривиальные у правильного дерева (рис.1):
- {BACAN, BACSU} против {LISMO, LACDA, STRP1, CLOTE, FINM2}
- {BACAN, BACSU, LISMO} против {LACDA, STRP1, CLOTE, FINM2}
- {BACAN, BACSU, LISMO, CLOTE, FINM2} против {LACDA, STRP1}
- {BACAN, BACSU, LISMO, LACDA, STRP1} против {CLOTE, FINM2}

