Реконструкция деревьев по нуклеотидным последовательностям. Деревья, содержащие паралоги.
Построение дерева по нуклеотидным последовательностям
Для того, чтобы построить филогенетическое дерево выбранных ранее бактерий по нуклеотидным последовательностям, используем последовательности 16S рРНК. Данные по этим последовательностям приведены в таблице 1.
Таблица 1.
| Название вида | Мнемоника | АС записи EMBL | Координаты гена | Направление |
| Bacillus anthracis | BACAN | AE016879 | 9335..10841 | прямое |
| Bacillus subtilis | BACSU | AP012496 | 9810..11360 | прямое |
| Clostridium tetani | CLOTE | AE015927 | 8715..10223 | обратное |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421..11973 | прямое |
| Lactobacillus delbrueckii | LACDA | CP000156 | 35825..37395 | прямое |
| Lactococcus lactis | LACLM | CP004884 | 500104..501616 | прямое |
| Listeria monocytogenes | LISMO | CP007600 | 2063..3615 | обратное |
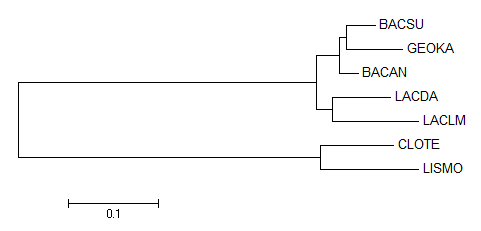
Полученные с помощью программы seqret пакета EMBOSS последовательности выровняли программой Muscle в JalView, в программе Mega построили дерево (см. рис. 1) с помощью алгоритма Neighbour-joining.
|
|
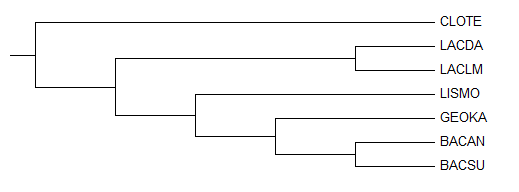
| Рис. 1. Филогенетическое дерево, построенное по последовательностям 16S рРНК. | Рис. 3. Правильное филогенетическое дерево исследуемых бактерий. |
Полученное дерево не совпадает с правильным, однако они достаточно похожи. Основное отличие - в дереве, построенном по последовательности рРНК, нет ветви, отделяющей всех Bacillales, {BACSU, BACAN, GEOKA, LISMO}, из-за того, что LISMO объединена в кладу с CLOTE. Это неудивительно, одна последовательность из всего генома вполне может выбиваться, а правильное дерево составлено по множеству последовательностей.
Построение и анализ дерева, содержащего паралоги
Для построения дерева нам необходимо найти белки, которые гомологичны CLPX_BACSU, из выбранных нами бактерий. С помощью программ makeblastdb и blastp найдём гомологи среди белков из файла proteo.fasta с E-value не больше 0,001 (выход blast в файле по ссылке). Из отобранных белков выберем только принадлежащие интересующим нас бактериям. Всего отобрали 31 белок, получили fasta-файл с их последовательностями. Сделали вырванивание программой Muscle в JalView, в программе Mega построили дерево (см. рис. 3) с помощью алгоритма Neighbour-joining.

|
| Рис. 3. Дерево гомологов белка CLPX_BACSU. |
По полученному дереву, можем сказать, какие белки являются паралогами, какие ортологами.
- Ортологи:
- CLPX_BACSU, CLPX_BACAN, CLPX_GEOKA, CLPX_LISMO, CLPX_CLOTE, CLPX_LACLM;
- CLPE_BACSU и Q8Y8B1_LISMO;
- Q5L436_GEOKA и B0AY46_BACAN.
- Паралоги:
- C2NBK7_BACAN, B1SHF4_BACAN, B0AWL5_BACAN;
- Q1G869_LACDA и Q1GBM8_LACDA
Соответствующие ветви на дереве отражают дупликацию гена внутри одного организма (в случае паралогов) или разделение путей эволюции белков в результате видообразования (в случае ортологов).