|
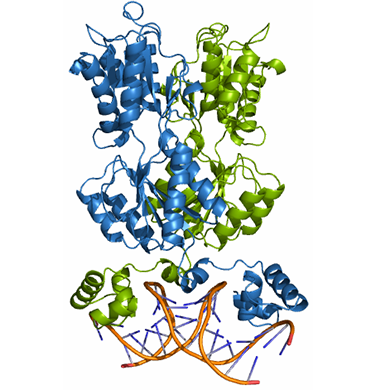
| Рис. 1. Структура комплекса димера пуринового репрессора с ДНК (1QP0). Мономеры окрашены синим и зелёным. |
Построение поверхности
Восстановление поверхностей различных контактов между молекулами проводилось для структуры комплекса пуринового репрессора с ДНК (1QP0). Димер былл восстановлен командой PyMOL symexp sym, 1qpz, all, 4. Общий вид структуры приведен на рисунке 1. Ниже приводятся команды для выделения различных множеств атомов, соответствующих контактам молекул. После выделения набор атомов отрисовывался как поверхность.
Поверхности контакта мономеров
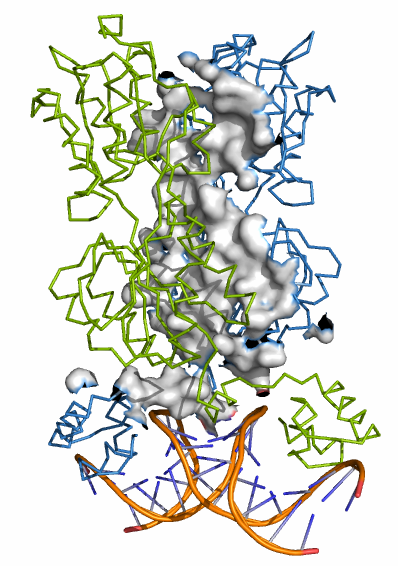
Изображение на рис. 4. Использовавшася команда: select c1, /1QP0//A near_to 5 of /sym//A
Поверхности контакта димера и ДНК
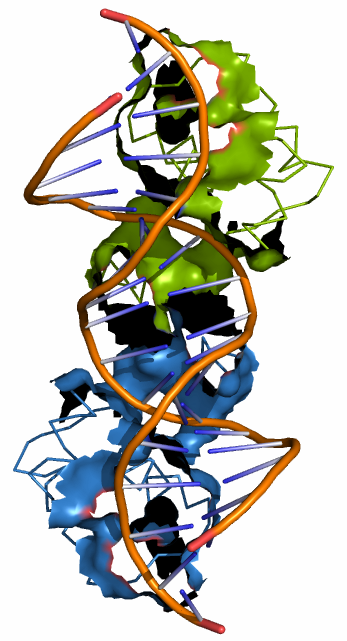
Изображение на рис. 5. Использовавшася команда: select c2, /obj01// near_to 5 of start or /obj02// near_to 5 of start # start - выделенные N-концы мономеров, obj01 и obj02 - цепи ДНК.
|
|
| Рис. 4. Поверхность контакта мономеров. Показаны остовы цепей А (синяя) и В (зелёная) и поверхность контакта (серым). | Рис. 5. Поверхность контакта димера с ДНК. Показаны остовы цепей А (синяя) и В (зелёная) около ДНК и поверхность контакта. |
Поверхности контакта ДНК и димера
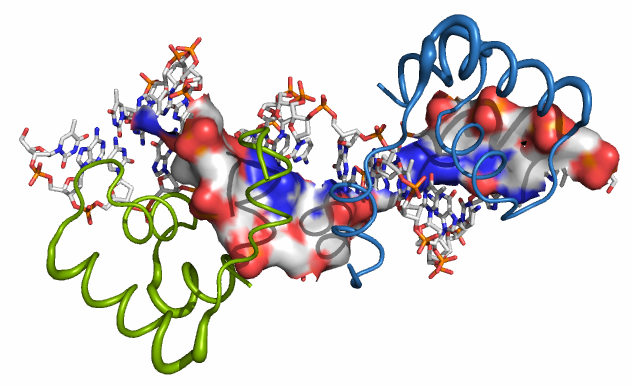
Изображение на рис. 6. Использовавшася команда: select c3, /obj01// near_to 5 of start or /obj02// near_to 5 of start # обозначения те же.
|
| Рис. 6. Поверхность контакта ДНК и димера. Показаны остовы цепей А (синяя) и В (зелёная) и поверхность контакта (покрашена по цветам атомов). |
Поверхности контакта мономеров и гидрофобные кластеры
С помощью сервиса Clud был произведён поиск гидрофобных кластеров между мономерами белка (для этого димер был сохранён в новый PDB-файл, а цепи переименованы, чтобы программа могла их отличить). Изображение найденных с порогом 4.7 Å трёх кластеров, включающих более 10 атомов, приведено на рис. 7. Из выдачи Clud были получены координаты атомов гидрофобных кластеров. Они были отмечены на изображении поверхности контакта мономеров на расстоянии 5 Å - рис. 8.
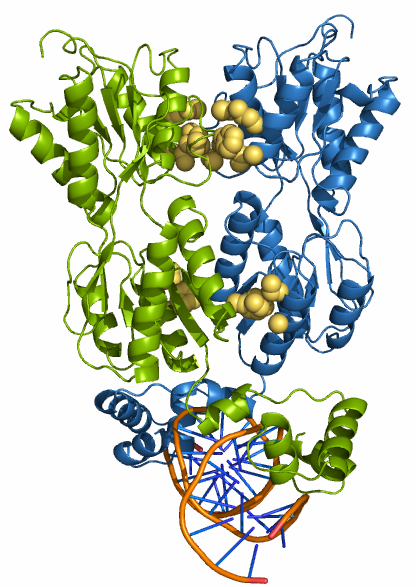
|

|
| Рис. 7. Гидрофобные кластеры между мономерами (жёлтые атомы). Цепи А и В - синим и зелёным. | Рис. 8. Поверхность контакта мономеров. Показаны остовы цепей А (синяя) и В (зелёная). На поверхности контакта атомы из гидрофобных кластеров показаны жёлтым. |