blastp
| Гомологи UVRB_BACSU | |||
| Поиск в Swiss-Prot | Поиск в PDB | Поиск в nr | |
| Самый хороший гомолог | |||
| Accession | P37954 | 2D7D | NP_391397 |
| E-value | 0.0 | ||
| Identity | 100% | ||
| Score | 3 485 | ||
| Другие хорошие гомологи (E-value < 1e-10) | |||
| Количество | 316 | 10 | 5 391 |
| Самый плохой из удовлетворительных гомолог (E-value < 1) | |||
| Номер | 1 035 | 61 | 19 921 |
| Accession | Q47VS0 | 2Z0M | XP_003996631 |
| E-value | 0.99 | 0.64 | 0.99 |
| Identity | 33% | 38% | 30% |
| Similarity | 56% | 52% | 45% |
| Score | 82 | 74 | 96 |
| Длина выравнивания | 85 | 53 | 109 |
| Координаты выравнивания | UVRB_BACSU: 428-511 Гомолог: 432-513 |
UVRB_BACSU: 491-543 Гомолог: 262-309 |
UVRB_BACSU: 445-552 Гомолог: 410-513 |
| Число гэпов | 4 | 5 | 6 |
* Поиск гомологов UVRB_BACSU проводился с помощью protein blast
(алгоритм blastp, запуск со стандартными значениями параметров, кроме следующих:
было установлено E-value = 1 и запрашивалось максимально возможное количество гомологов - 20 000).
** E-value - это ожидаемое количество находок в случайном банке последовательностей,
с весом парного выравнивания с белком таким же или большим, как вес парного выравнивания белка с находкой из определённого банка последовательностей.
Если E-value = 10, то ожидается найти 10 находок в случайном банке последовательностей с весом парного выравнивания с белком таким же или большим,
как вес парного выравнивания белка с находкой из определённого банка последовательностей.
Если E-value = 0.001, то ожидается найти лишь одну находку в 1000 или более случайных банках последовательностей с весом парного выравнивания с белком таким же или большим,
как вес парного выравнивания белка с находкой из определённого банка последовательностей.
Из приведённой выше таблицы видно, что в качестве самого хорошего гомолога UVRB_BACSU в Swiss-Prot, PDB и nr найден сам UVRB_BACSU.
Количество хороших гомологов и номер самого плохого гомолога UVRB_BACSU, по сравнению с результатами при поиске в Swiss-Prot, значительно меньше при поиске в PDB (т.к. лишь для немногих белков получена кристаллографическая структура) и значительно больше при поиске в nr (т.к. в этом случае поиск проводится в GenBank CDS translations, Swiss-Prot, PDB, PIR и SEQDB).
| Лучшие эукариотические гомологи UVRB_BACSU | |||
| Поиск в Swiss-Prot | Поиск в PDB | Поиск в nr | |
| Организм | Neurospora crassa | Saccharomyces cerevisiae | Cyanidioschyzon merolae |
| Название белка | ATP-dependent RNA helicase dbp3 | carboxyl-terminal domain of mRNA exporter Dbp5p |
excinuclease ABC subunit B |
| Accession | Q7S5R1 | 2KBF | BAM79644 |
| E-value | 2e-07 | 8e-08 | 0.0 |
| Identity | 33% | 35% | 50% |
| Similarity | 56% | 54% | 67% |
| Score | 135 | 123 | 1 647 |
| Длина выравнивания | 97 | 96 | 654 |
| Координаты выравнивания | UVRB_BACSU: 421-517 Гомолог: 426-522 |
UVRB_BACSU: 449-543 Гомолог: 39-134 |
UVRB_BACSU: 4-654 Гомолог: 227-871 |
| Число гэпов | 0 | 1 | 12 |
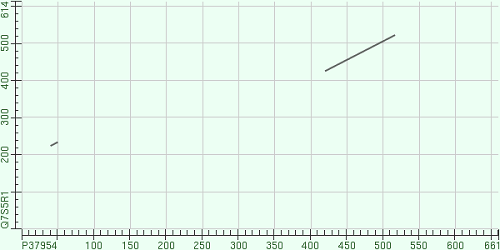
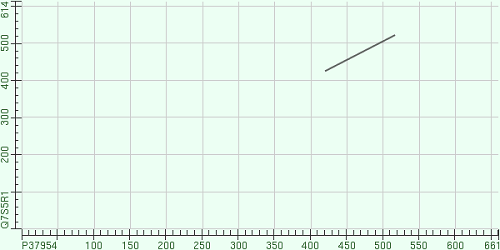
Рисунок 1. Карта локального сходства UVRB_BACSU и его эукариотического гомолога DBP3_NEUCR (E-value = 10).
Рисунок 2. Карта локального сходства UVRB_BACSU и его эукариотического гомолога DBP3_NEUCR (E-value = 0.01).
Из рисунков 1 и 2 видно, что чем больше E-value, тем большее локальное сходство приписывается сравниваемым аминокислотным последовательностям.
Наверх