Множественное выравнивание
Составление репрезентативной выборки гомологов UVRB_BACSU
| Поиск гомологов UVRB_BACSU с помощью protein blast | ||||||
| Алгоритм | База данных | Организмы | Установленное E-value | Запрашиваемое количество гомологов | Количество найденных гомологов | |
| blastp | RefSeq | Procaryota, за исключением Firmicutes | 1e-10 | 500 | 500 | |
| Eucaryota | 0.001 | 500 | 214 | |||
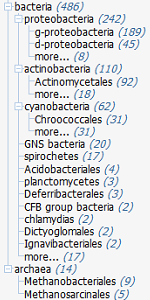
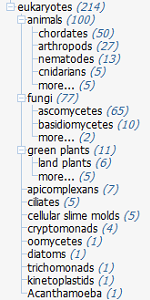
| Репрезентативная выборка гомологов UVRB_BACSU | |||
| Домен | Тип | Вид | Количество гомологов |
| Archaea | Euryarchaeota | Methanobacterium formicicum Methanobrevibacter smithii Methanolobus psychrophilus |
3 |
| Bacteria | Acidobacteria | Granulicella tundricola | 1 |
| Actinobacteria |
Arthrobacter gangotriensis Collinsella intestinalis Saccharomonospora viridis Slackia piriformis Streptomyces bottropensis Streptomyces somaliensis |
6 | |
| Chlamydiae | Parachlamydia acanthamoebae | 1 | |
| Cyanobacteria | Arthrospira maxima Chroococcidiopsis thermalis Microcystis aeruginosa Nostoc punctiforme Synechococcus elongatus |
5 | |
| Deferribacteres | Denitrovibrio acetiphilus | 1 | |
| Dictyoglomi | Dictyoglomus thermophilum | 1 | |
| Firmicutes | Bacillus subtilis | UVRB_BACSU | |
| Chlorobi | Ignavibacterium album | 1 | |
| Planctomycetes | Planctomyces limnophilus | 1 | |
| Proteobacteria |
Cupriavidus metallidurans Desulfovibrio desulfuricans Escherichia coli Geobacter sulfurreducens Marinobacter adhaerens Pseudomonas fluorescens |
6 | |
| Spirochaetes | Leptospira interrogans Leptospira weilii |
2 | |
| Thermodesulfobacteria | Thermodesulfatator indicus | 1 | |
| Eucaryota | Arthropoda | Drosophila melanogaster | 1 |
| Ascomycota | Saccharomyces cerevisiae | 1 | |
| Chordata | Human | 1 | |
| Mycetozoa | Dictyostelium discoideum | 1 | |
| Некоторые эукариотуческие гомологи UVRB_BACSU | ||||
| UniProt ID | FANCM_HUMAN | DDX17_DROME | DBP3_YEAST | Q54EH7_DICDI |
| Организм | Human | Drosophila melanogaster | Saccharomyces cerevisiae | Dictyostelium discoideum |
| Название белка | Fanconi anemia group M protein |
ATP-dependent RNA helicase p62 |
ATP-dependent RNA helicase DBP3 |
RNA-directed RNA polymerase |
| Хромосома | 14 | 3R | 7 | 6 |
Исходя из данных, приведённых в таблице выше, можно предположить, что UVRB_BACSU имеет эукариотическое происхождение, т.к. у различных эукариот гены, кодирующие его гомологов, локализованы в ядерной ДНК, а не в митохондриальной ДНК. Локализация кодирующих генов в митохондриальной ДНК могла бы свидетельствовать о прокариотическом происхождении UVRB_BACSU и появлении его гомологов в эукариотических организмах в результате эндосимбиоза.
Множественное выравнивание отобранных гомологов UVRB_BACSU

| Цветовая схема окрашивания множественного выравнивания | |||||||||||||||||||
| G | P | C | S | T | N | Q | D | E | R | K | M | I | L | V | A | F | Y | W | H |

• ADP-binding_site (аминокислты, связывающие АДФ, подписаны "S")
• Secondary_structure (аминокислоты, образующие α-спираль, отмечены красным цветом и подписаны "α". Аминокислоты, образующие β-тяж, отмечены зелёным цветом и подписаны "β")
• Blocks (участки множественного выравнивания, не содержащие гэпов, отмечены красным цветом и подписаны "Block X")
Результаты анализа множественного выравнивания
Множественное выравнивание прокариотических гомологов UVRB_BACSU достаточно консервативно, причём высококонсервативные участки выравнивания могут не соответствовать элементам вторичной структуры, и наоборот, элементы вторичной структуры могут быть образованы не очень консервативными аминокислотными последовательностями. Возможно, эти элементы вторичной структуры присутствуют не у всех гомологов, или для них не принципиально, какими аминокислотными последовательностями они образованы.
Множественное выравнивание содержит шесть больших блоков, пять из которых принадлежат цепи А и один принадлежит цепи В. Участок гэпов между первым и вторым блоками соответствует инсерции 132Gly в четвёртой α-спирали у Ignavibacterium album. Участки гэпов между вторым, третьим и четвёртым блоками соответствуют инсерциям нескольких аминокислот в β-поворотах у некоторых видов бактерий.
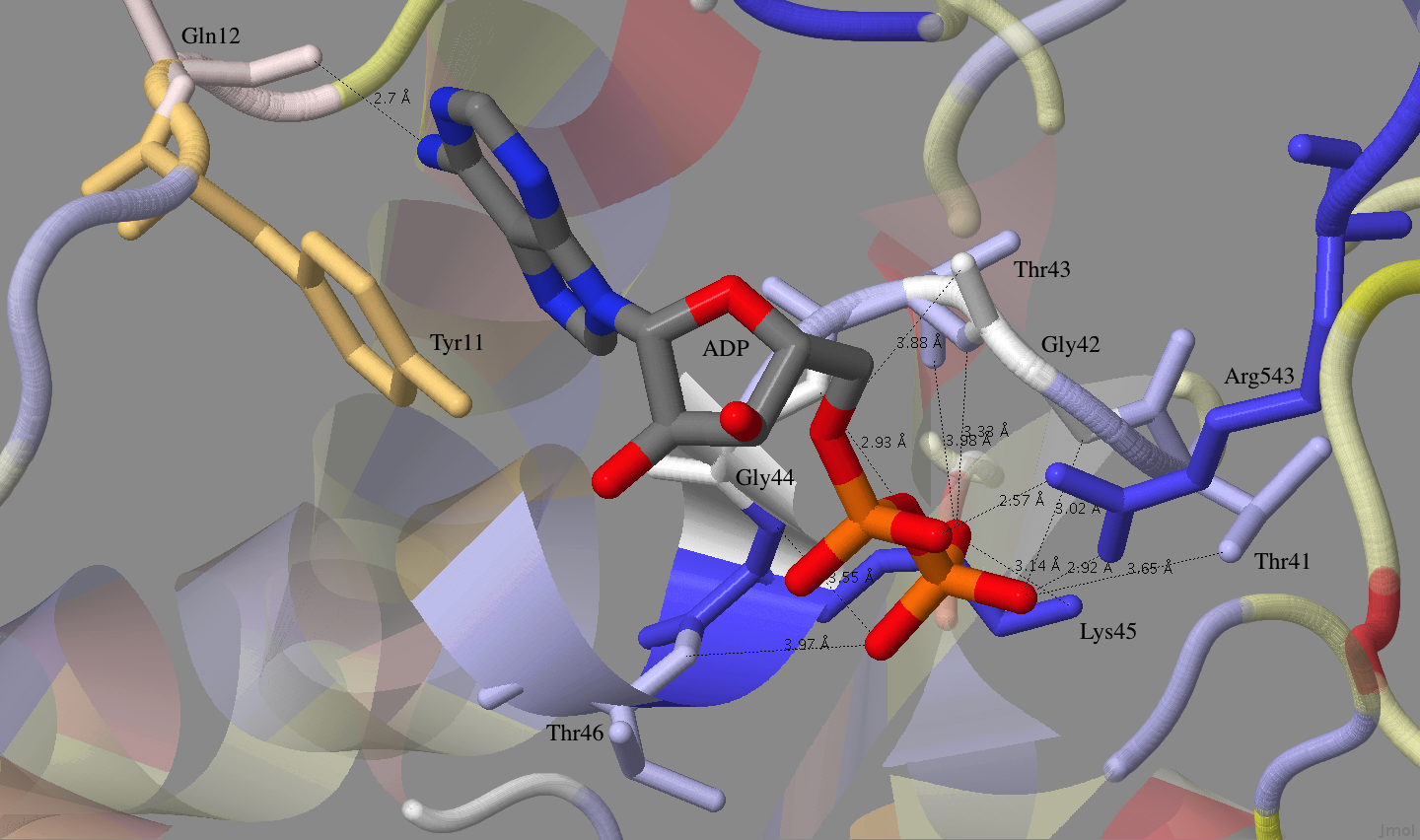
Пять взаимодействующих с АДФ аминокислотных остатков (Thr41, Gly42, Gly44, Lys45 и Arg543) консервативны у всех отобранных гомологов UVRB_BACSU. Два взаимодействующих с АДФ аминокислотных остатка (Thr43 и Thr46) у некоторых гомологов UVRB_BACSU заменены на Ser. Tyr11, взаимодействующий с пиримидиновым кольцом АДФ, у некоторых гомологов UVRB_BACSU заменён на Phe или His. Gln12, образующий водородную связь своей кето-группой, принадлежащей пептидному остову, с амино-группой АДФ, неконсервативен, т.к. в данном случае радикал аминокислоты не имеет функционального значения.
Наверх