Отчёт по 1 семестру
Мини-обзор генома Brucella canis
На этой странице вы можете ознакомиться с моей работой за 1 семестр - мини-обзором генома Brucella canis. В нём рассматриваются и анализируются стандартные данные о геноме и протеоме Brucella canis, приводится сравнительная характеристика выбранных кодирующих участков ДНК-гиразы родственных видов Brucella с помощью инструментария электронных таблиц, командной оболочки BASH, скриптов, написанных на языке программирования Pyhon.
Ссылка на мини-обзор: Мини-обзор генома Brucella canis
Ссылка на сопроводительные материалы: Сопроводительные материалы
1 Введение
Brucella canis – это грам-отрицательная коккобацилла из класса Alphaproteobacteria (Таблица 1), вызывающая бруцеллёз у собак и других псовых [1].
| Домен | Bacteria |
| Филлум | Pseudomonadota |
| Класс | Alphaproteobacteria |
| Порядок | Hyphomicrobiales |
| Семейство | Brucellaceae |
| Род | Brucella |
| Вид | B.canis |
Наиболее часто встречающимися симптомами бруцеллёза являются репродуктивные нарушения обоих полов собак и аборты [1]. Люди, занимающиеся разведением собак в питомниках и контактирующие с инфицированными тканями собак, подвержены риску заражения данной бруцеллой [2]. Так как Brucella canis патогенна и несёт значительные экономические потери для питомников, изучение её генома является важной задачей научных исследований.
2 Материалы и методы
Геномные данные B. canis ATCC 23365 (ID 483179) и родственных видов B.abortus 544 (ID 93016562) и B. melitensis bv. 1 str. 16M (ID 29593694) взяты с сайта Национального центра биотехнологической информации (NCBI). Для подсчёта количества нуклеотидов и GC-состава геномной последовательности Brucella canis, попарного сравнения аминокислотной последовательности белка гиразы родственных видов Brucella были использованы скрипты, написанные на языке программирования Python (задействовались методы работы с файлами, цикл for, условный оператор if) [S1]. Для подсчёта количества различных типов кодирующих элементов был использован инструментарий электронных таблиц Google sheets (создание фильтров и функция “СЧЁТЕСЛИ”) [S2]. Для построения диаграммы, иллюстрирующей количество старт-кодонов, гистрограммы длин белков и подсчёта статистических показателей распределения длин белков был так же использован инструментарий электронных таблиц Google sheets (метод построения диаграмм, функции “МИН”, “МАКС”, “СРЗНАЧ”, “МЕДИАНА”, “СТАНДОТКЛОН”) [S4],[S5]. Для определения количества различных старт-кодонов был использован скрипт, написанный для командной оболочки Bash [S3].
3 Результаты и обсуждения
3.1 Описание стандартных данных о геноме Brucella canis
Геном Brucella canis состоит из двух хромосом, причём хромосома II считается плазмидного происхождения [3], плазмиды в геноме отсутствуют (Таблица 2, данные получены с помощью S1).
| Репликон | Длина, п.н. | GC-состав, % |
|---|---|---|
| chr 1 | 2105969 | 57.2 |
| chr 2 | 1206800 | 57.3 |
| Всего | 4129470 | 57.25 |
По результатам обработки видно, что размер хромосомы I примерно в два раз больше размера хромосомы II. Содержание GC-пар в геноме Brucella canis примерно равно 57%, поэтому можно утверждать, что бактерия не является термофилом и живёт в умеренных температурных условиях [1]. При дальнейшем анализе генома было определено количество различных типов кодирующих элементов на хромосомах (Таблица 3, полученная с помощью S2)
| Seq_type | CDS_with_ protein | CDS_without_protein | tRNA | rRNA |
|---|---|---|---|---|
| chr1 | 1945 | 78 | 41 | 6 |
| chr2 | 1067 | 63 | 14 | 3 |
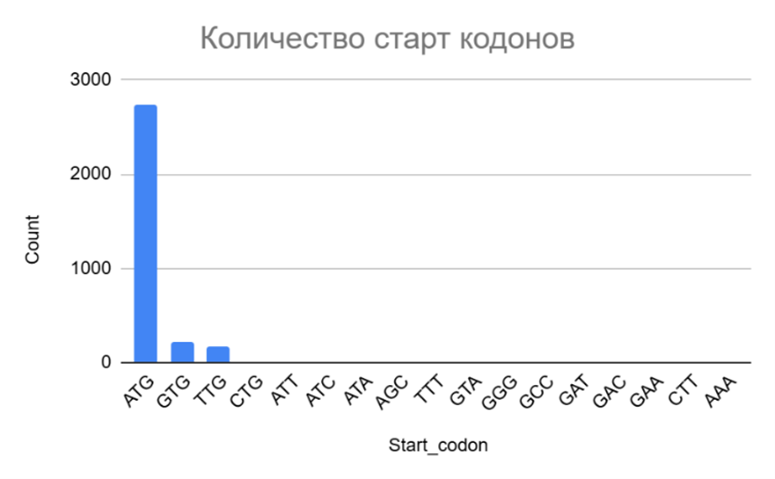
В процессе статистической обработки было выявлено наличие таких видов кодирующих элементов, как CDS (coding sequences - кодирующие последовательности), rRNA (рибосомальные РНК), tRNA (транспортные РНК). По результатам обработки было замечено, что доля генов, не кодирующих белки, так называемых псевдогенов, на хромосоме II больше, что, возможно, указывает на потерю функций этих генов в связи с переходом к внутриклеточному паразитическому образу жизни. Также было определено количество различных старкодонов для кодирующих последовательностей (Рисунок 1, результаты получены с помощью S3 и S4).
Видно, что преобладающим старт-кодоном остаётся кодон ATG, но иногда старт-кодонами могут являться и другие кодоны. Например, кодоны GTG и TTG, вероятно, появились в результате мутации первого нуклеотида кодона ATG и закрепились в некоторых генах, о чём свидетельствует их встречаемость в геноме B. сanis. Нетипичные кодоны могли возникнуть из-за ошибок секвенирования.
3.2 Описание стандартных данных о протеоме Brucella canis
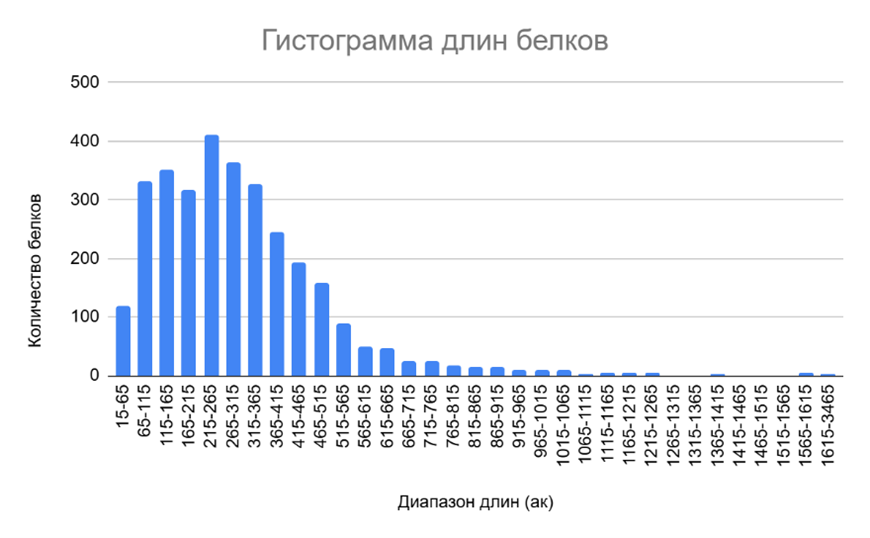
С помощью электронных таблиц найдено распределение белков по длине в аминокислотных остатках (Рисунок 2, данные получены с помощью S5), рассчитаны некоторые статистические показатели распределения (Таблица 4, данные получены с помощью S5).
| Показатель |
Значение |
|---|---|
| Минимальное значение |
19 |
| Максимальное значение |
3420 |
| Среднее значение |
306 |
| Медиана |
271 |
| Стандартное отклонение |
630 |
Наибольшее количество белков находится в диапазоне 65- 365 нуклеотидов. Небольшой размер белков, вероятно, свидетельствует об отсутствии многодоменной структуры. Самый короткий белок – гипотетический, самый длинный – ‘autotransporter adhesin’, участвующий в адгезии граотрицательных бактерий, в том числе Brucella canis, к внеклеточному матриксу клетки-хозяина.
3.3 Сравнительный анализ аминокислотной последовательности gyrA Brucella canis и родственных видов
Род Brucella разделён на 6 видов, среди которых B. melitensis и B. abortus патогенны для человека [3]. При лечении бруцеллёза человека в основном сочетают антибиотики разных классов, например, доксициклин и стрептомицин [4]. Для преодоления антибиотикорезистентности или развившейся токсичности ранее используемых препаратов, при лечении бруцеллёза можно применять флуорохинолоны, ингибирующие ДНК-гиразу [5]. Штаммы, которые приобрели устойчивость к антибиотикам, обладающим фторхинолоноподобным эффектом, вероятно, будут иметь мутации в гене белка гиразы субъединицы А (gyrA) [6]. В данной работе было проведено попарное сравнение аминокислотных последовательностей белка гиразы субъединицы А (gyrA) у бактерий B. canis, B. melitensis и B. abortus (Таблица 5, результаты получены с помощью S1).
| Сравниваемые виды |
Количество замен |
Замена аминокислоты |
Замена класса аминокислоты |
|---|---|---|---|
| B.canis – B.melitensis |
3 |
S – N F – S V – L |
НЕТ (Полярная) Ароматическая - полярная НЕТ (Гидрофобная) |
| B. canis – B.abortus |
2 |
S – N F – S |
НЕТ (Полярная) Ароматическая - полярная |
| B. abortus – B.melitensis |
1 |
V – L |
НЕТ (Гидрофобная) |
Поскольку длина белка gyrA у всех рассматриваемых видов Brucella одинаковая, можем судить о достаточной консервативности белка. Анализ выравнивания показал, что наибольшее количество различий обнаруживается при сравнении B. canis и B. melitensis, наименьшее – B. melitensis и B. abortus. Также можно сделать следующие выводы: B. melitensis и B. abortus более родственные друг другу виды, чем виду B. canis, что согласуется с существующими данными [7]; у общего предка B. melitensis и B. abortus произошла мутация в гене gyrA, приводящая к замене аминокислоты, но не её класса на 599 позиции; мутации в гене gyrA у общего предка B. melitensis, B. abortus, B. canis привели к замене аминокислоты на 580 позиции, к замене аминокислоты и её класса на 638 позиции, что отделило B. canis от двух других видов. Возможно, эти мутации повлияли на некоторые параметры гиразы, например, её конформационную устойчивость.
4 Сопроводительные материалы
S1. Python-скрипты, используемые в работе: подсчёт количества нуклеотидов, GC-cостава в геноме Brucella canis, попарное сравнение аминокислотной последовательности gyrA у родственных видов Brucella
https://colab.research.google.com/drive/1sDJSD1Fy8OqVeV0RLCtssLzVwzY6E5Ua#scrollTo=7VYK3YahMXHL
S2. Таблица Google sheet особенностей генома Brucella canis (лист gene, лист CDS_with, лист CDS_without): подсчёт количества различных типов кодирующих элементов
S3. Bash-скрипт, используемый в работе: определение количества различных старт-кодонов
https://drive.google.com/drive/folders/1_L8HOOBmzDeVB2JoVSxq6bw-gIr6Y0HY
S4. Таблица Google sheet для старт-кодонов в геноме Brucella canis: cтолбчатая диаграмма, иллюстрирующая количество старт-кодонов в геноме Brucella canis
S5. Таблица Google sheet для гистограммы длин белков Brucella canis:
5 Литература
[1] Статья в Википедии о бактерии
[2] Hollett RB. Canine brucellosis: outbreaks and compliance. Theriogenology. 2006 Aug;66(3):575-87. doi: 10.1016/j.theriogenology.2006.04.011. Epub 2006 May 23. PMID: 16716382.
[3] Ficht T. Brucella taxonomy and evolution. Future Microbiol. 2010 Jun;5(6):859-66. doi: 10.2217/fmb.10.52. Erratum in: Future Microbiol. 2010 Aug;5(8):1299. PMID: 20521932; PMCID: PMC2923638.
[4] Arijit Shil, Dr. Paramita Dey, Dr. Tapan Kumar Mandal. The role of fluoroquinolones for the treatment of brucellosis: An overview. Pharma Innovation 2020;9(10):10-13. doi: 10.22271/tpi.2020.v9.i10a.5353.
[5] Solera J, Martínez-Alfaro E, Espinosa A. Recognition and optimum treatment of brucellosis. Drugs. 1997 Feb;53(2):245- 56. doi: 10.2165/00003495-199753020-00005. PMID: 9028744.
[6] Chaoui I, Oudghiri A, El Mzibri M. Characterization of gyrA and gyrB mutations associated with fluoroquinolone resistance in Mycobacterium tuberculosis isolates from Morocco. J Glob Antimicrob Resist. 2018 Mar;12:171-174. doi: 10.1016/j.jgar.2017.10.003. Epub 2017 Oct 13. PMID: 29033301.
[7] Foster JT, Beckstrom-Sternberg SM, Pearson T, Beckstrom-Sternberg JS, Chain PS, Roberto FF, Hnath J, Brettin T, Keim P. Whole-genome-based phylogeny and divergence of the genus Brucella. J Bacteriol. 2009 Apr;191(8):2864-70. doi: 10.1128/JB.01581-08. Epub 2009 Feb 6. PMID: 19201792; PMCID: PMC2668414