UniProt
1. Выбор белка
Сначала с помощью расширенного поиска на сайте UniProt был сделан запрос с указанием исследуемой бактерии - Brucella canis strain ATCC 23365 и получено 3238 находки, из которых 364 записи проверены (Swiss-Prot) и 2874 записи не проверены (без указания штамма бактерии нашлось на 79 записей больше). Далее я захотела выбрать белок, участвующий в гликолизе, поэтому добавила указание Ключевого слова (Keyword) - Glycolisis: (taxonomy_id:483179)) AND (keyword:KW-0324)). Среди 10 полученных записей оказался один белок, участвующий не в гликолизе, а в цикле Кребса, и имеющий довольно большую длину в 1004 а.o. - это E1-субъединица 2-оксоглутаратдегидрогеназы (2-oxoglutarate dehydrogenase E1 component, AC: A9M8Q9). Далее в отчёте речь пойдёт об этом ферменте.
2. Информация о белке
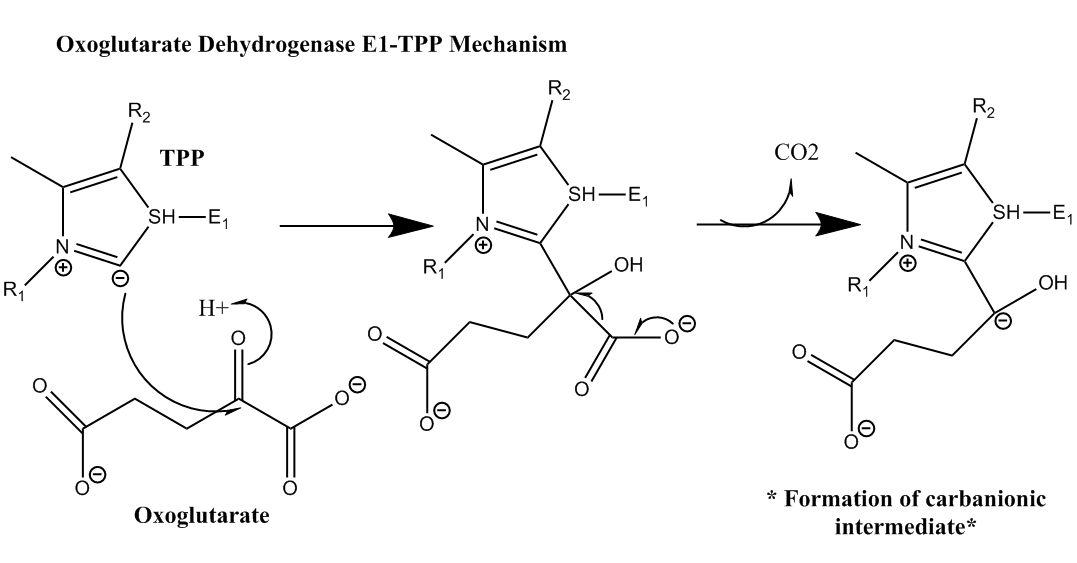
Функция: Катализирует необратимое декарбоксилирование 2-оксоглутарата (альфа-кетоглутарата) и последующий перенос декарбоксилированного ацильного интермедиата на окисленную дигидролипоильную группу, ковалентно присоединенную к Е2-субъединице комплекса с последующим образованием сукцинил-КоА. Играет ключевую роль в цикле Кребса - основном пути окисления углеводов, жирных кислот и аминокислот.
Кофактор: тиаминпирофосфат
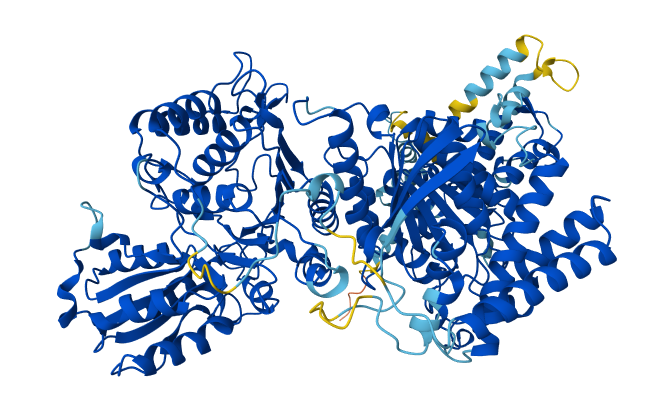
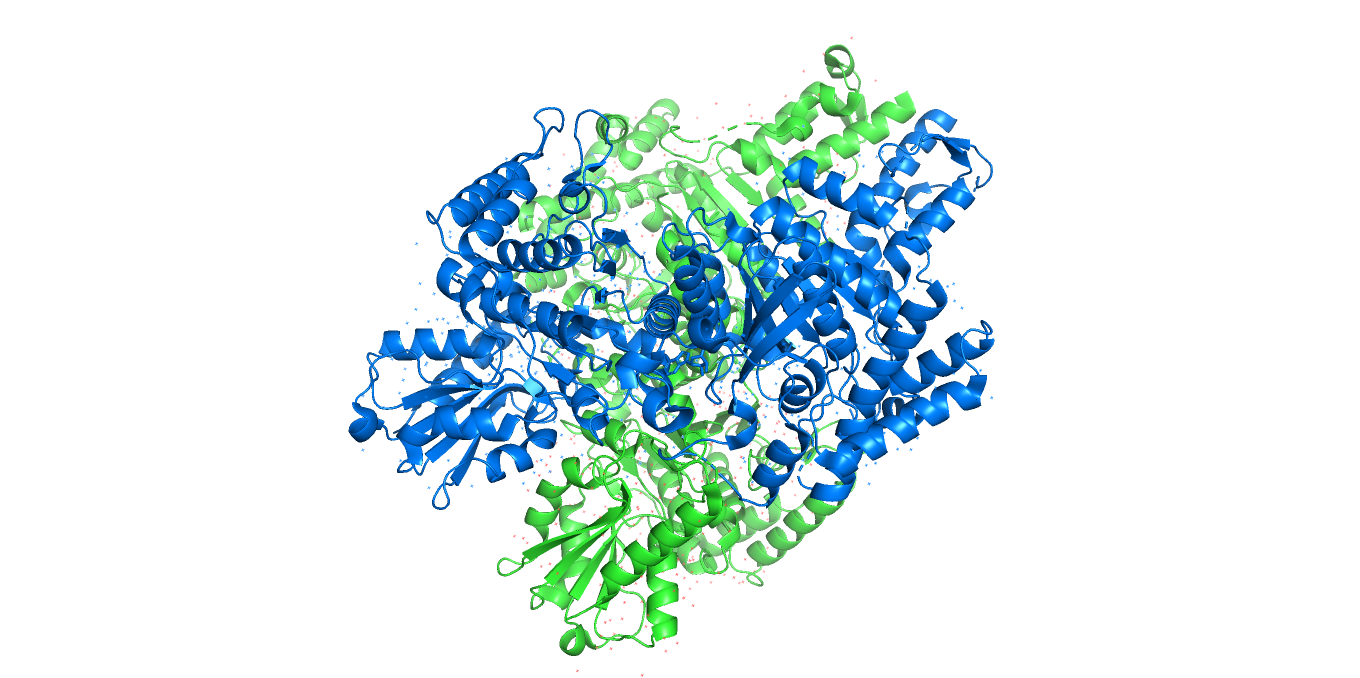
Структура: гомодимер
Длина: 1004 а.о.
3. Поисковые запросы
1. (taxonomy_id:483179) AND (go:0006099) NOT (cc_pathway:"tricarboxylic acid cycle") NOT (keyword:KW-0816)
Как было замечено ранее, даже расширенный поиск UniProt несовершенен, особенно для не очень хорошо аннотированных белков. Поэтому был сделан следующий запрос: в поле "Taxonomy[OC]" введена таксономия изучаемой бактерии, в поле "Gene Ontology (GO)" - GO-термин для "цикла трикарбоновых кислот", не включая находки поля "Pathway" - "tricarboxylic acid cycle" и поля Keyword - ключевое слово для "tricarboxylic acid cycle". Получили, что 5 находок, включая рассматриваемый белок ODO1_BRUC2, не попадают в поиск по полю Pathway и Keyword. Вывод: в этом примере поле "Gene Ontology (GO)", содержащее список GO-терминов, имеет более широкий охват поиска по метаболическому пути.
2. (ec:1.2.4.2) NOT (go:0005829)
Такой запрос находит белок по классификации ферментов (ec) и местонахождению белка не в цитозоле (важно, что не в цитоплазме) в основном в группе эукариот, ведь у них этот фермент функционирует в митохондриях, которых у бактерий, очевидно, нет.
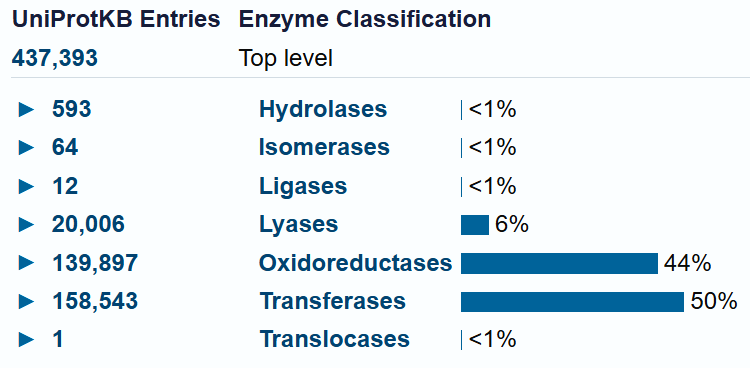
3. (cc_cofactor_chebi:"CHEBI:58937")
Указывая в поле "Cofactor" CheBI ID(Chemical Entities of Biological Interest) для кофактора тиаминпирофосфат и группируя по классам ферментов, получили следующий результаты встречаемости пирофосфата в следующих ферментах: окиредуктазы - реакции с альдегидной или оксо-группой доноров, трансферазы - перенос альдегидных или кетонных групп, лиазы - углеро-углерод. Действительно, такие ферменты, как Пируватдегидрогеназный комплекс (PDH), Транскетолаза (TK), Пируватдекарбоксилаза (PDC) используют в качестве кофактора тиаминпиросфат (он содержит тиазольное кольцо, которое может образовывать стабильные карбанионы, стабилизируют промежуточные соединения).
4. (taxonomy_id:2) AND (ec:1.2.4.2)
Указывая в поле "Enzyme Classification[EC]" ec изучаемого фермента и группируя по таксону Bacteria, получили 28,644 записи, результаты преобладающих таксонов приведены в Таблице 1, а также 42 записи для рода Brucella (Таблица 2).
| Кол-во записей | Таксон | % |
|---|---|---|
| 14,764 | Pseudomonadota | 52 |
| 7,259 | Bacillati | 25 |
| 3,641 | FCB group | 13 |
| Кол-во записей | Таксон | % |
|---|---|---|
| 6 | Brucella intermedia | 14 |
| 5 | Brucella abortus | 12 |
| 4 | Brucella suis | 10 |
5. (taxonomy_id:2) AND (ec:1.2.4.2) AND (structure_3d:true)
Нашлось 4 бактерии, для которых получена структура белка. Структура 2-оксоглутаратдегидрогеназы (также димера) из E.coli, полученная методом рентгеновской диффракции, представлена на Рис 5.