1. Глобальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | Identity | Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| 2-oxoglutarate dehydrogenase E1 component | ODO1_ECOLI | ODO1_BACSU | 1722.5 | 39.7% | 57.8% | 89 | 26 |
| Tryptophan-tRNA ligase | SYW_ECOLI | SYW_BACSU | 907.5 | 54.0% | 71.3% | 6 | 5 |
| DNA repair protein RecN | RECN_ECOLI | RECN_BACSU | 827.5 | 34.1% | 55.1% | 37 | 9 |
| Protein Name | ID 1 | ID 2 | Score | Identity | Similarity | Gaps | Indels | Coverage_1 | Coverage_2 |
|---|---|---|---|---|---|---|---|---|---|
| 2-oxoglutarate dehydrogenase E1 component | ODO1_ECOLI | ODO1_BACSU | 1724.5 | 39.9% | 58.0% | 87 | 24 | 99.57% | 99.36% |
| Tryptophan-tRNA ligase | SYW_ECOLI | SYW_BACSU | 910.5 | 54.7% | 72.2% | 4 | 3 | 99.10% | 99.10% |
| DNA repair protein RecN | RECN_ECOLI | RECN_BACSU | 829.5 | 34.7% | 56.0% | 29 | 8 | 99.64% | 98.26% |
По данным выравнивания можно полагать, что изучаемые белки гомологичны, но с меньшей степенью сходства у белков RECN, с немного большей - у ODO1 (стоит учитывать достаточно большую длину белка в ~930 aa и большее колиство гэпов) и самой большой - SYW. По большому счёту, глобальное и локальное выравнивание не имеют существенных различий, так как локальное выравнивание в приведённых примерах не включает первые и/или последние несколько аминокислотных остатков, за счёт этого оно становится немного короче, уменьшается количество гэпов и немного увеличивается процент идентичности: на 0.2%, 0.7%, 0.6% соответственно.
2. Выравнивание неродственных белков
Для выравнивания неродственных белков были выбраны флагеллярный биосинтетический белок FliP (ID: FLIP_ECOLI) кишечной палочки и метилентетрагидрофолат дегидрогеназа (ID: FOLD_BACSU) сенной палочки
| Алгоритм Выравнивания | Score | Identity | Similarity | Gaps | Indels | Coverage_1 | Coverage_2 |
|---|---|---|---|---|---|---|---|
| Needleman–Wunsch | 15.0 | 7.0% | 12.5% | 302 | 10 | - | - |
| Smith–Waterman | 28.5 | 30.8% | 53.8% | 4 | 1 | 8.98% | 9.19% |
Наиболее вероятно, что с процентом идентичности в 12.5% белки не обладают гомологией. Однако при локальном выравнивании нашёлся участок, демонстрирующий высокий процент сходства. На самом деле, этот участок совсем короткий ~20aa относительно всего белка и выровнялся случайно, поэтому мы по-прежнему не можем сделать вывод о гомологии белков.
3. Множественное выравнивание белков с помощью Jalview
Для множественного выравнивания был выбран белок - триптофановая аминоацил-тРНК-синтетаза (RecName: Tryptophan--tRNA ligase). По запросу (id:SYW_*) нашлось 153 записи Swiss-Prot с такой мнемоникой функции. Для выравнивания отобраны белки из следующих организмов: Escherichia coli (strain K12) - ID:SYW_ECOLI; Bacillus subtilis (strain 168) - ID:SYW_BACSU; Helicobacter pylori (strain ATCC 700392 / 26695) - ID:SYW_HELPY; Mycoplasma genitalium (strain ATCC 33530 / DSM 19775 / NCTC 10195 / G37) - ID:SYW_MYCGE; Caenorhabditis elegans - ID:SYWM_CAEEL; Mus musculus (Mouse) - ID:SYWM_MOUSE; Homo sapiens (Human) - ID:SYWM_HUMAN. Причём для эукариотных организмов выбрана митохондриальная тРНК-синтетаза, а не цитоплазматическая.
Jalview -> File -> Fetch Sequences -> меню: Uniprot -> меню слева: Entry name -> окно справа: SYW_ECOLI; SYW_BACSU; SYW_HELPY; SYW_MYCGE; SYWM_CAEEL; SYWM_MOUSE; SYWM_HUMAN -> окно Retreived from Uniprot -> Web service -> Alignment -> ClustalO with Defaults
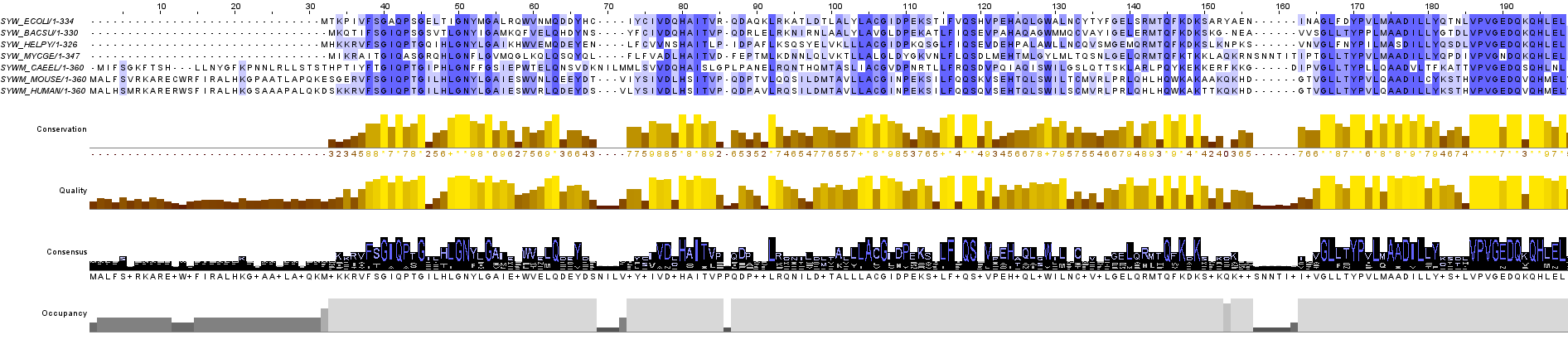
Аминоацил-тРНК-синтетазы — это ферменты, которые катализируют присоединение правильной аминокислоты к ее соответствующей тРНК. Происходит это в два этапа: активация аминокислоты (аминокислота реагирует с АТФ, образуя аминоацил-АМФ и пирофосфат) и перенос аминоацильного остатка на тРНК.
Наличие консервантивных и функционально-консервативных колонок 39-43, 49-51, 170-171, 175-179, 186-192, где последовательности практически полностью идентичны, свидетельствует о гомологии белков. Такие участки, вероятно, необходимы для правильной каталитической активности этого фермента, и действительно, в записи о белке в Uniprot указано, что эти участки являются сайтами связывания АТФ и L-триптофана - субстратов данной АРСазы, и поэтому сильно подвержены отбору. N- и C-концевые области aaRS менее консервативны и являются специфичными для разных организмов последовательностями, которые, возможно, участвуют в белок-белковых взаимодействиях, регуляции активности или локализации фермента, они не подвержены жёсткому отбору, что приводит к их дивергенции.