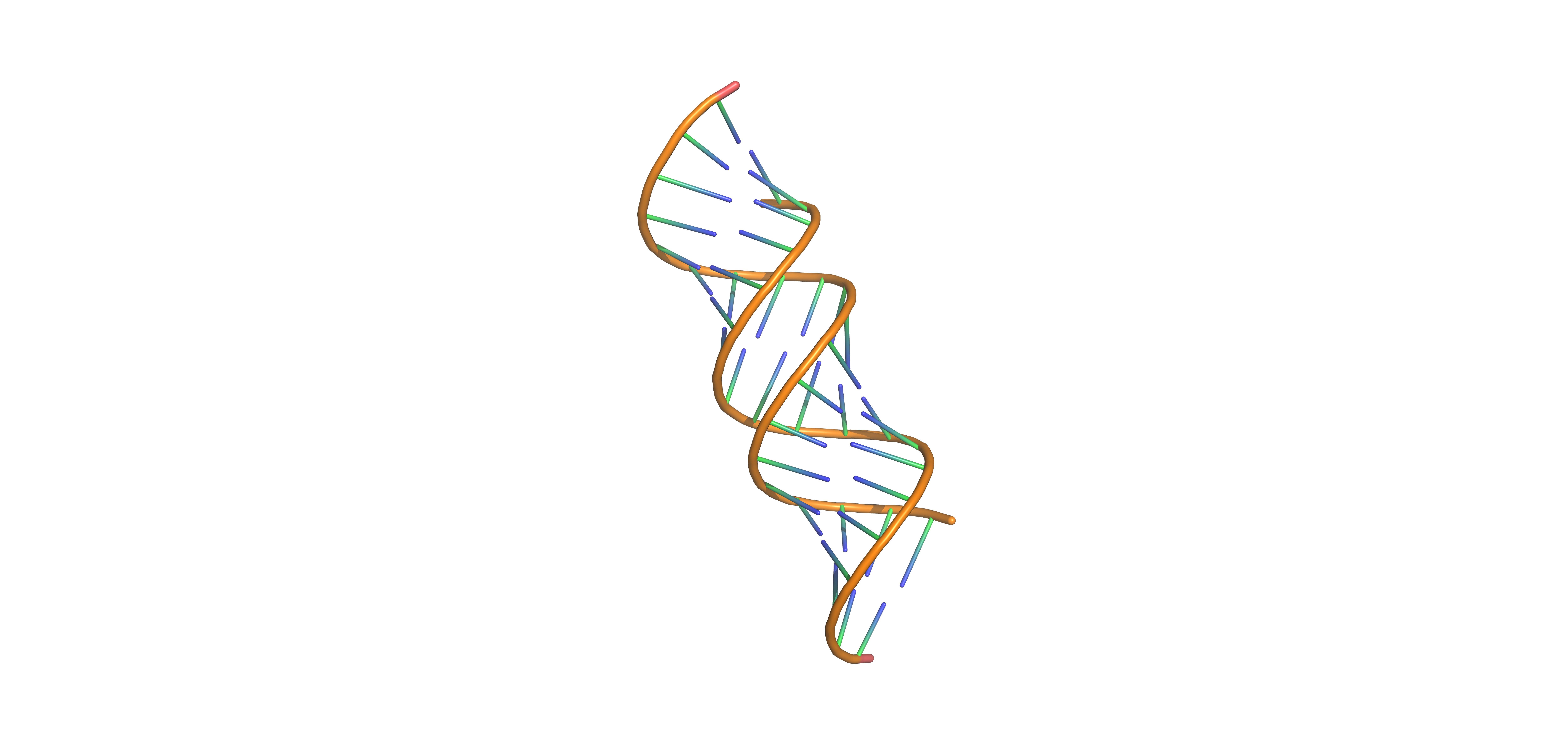

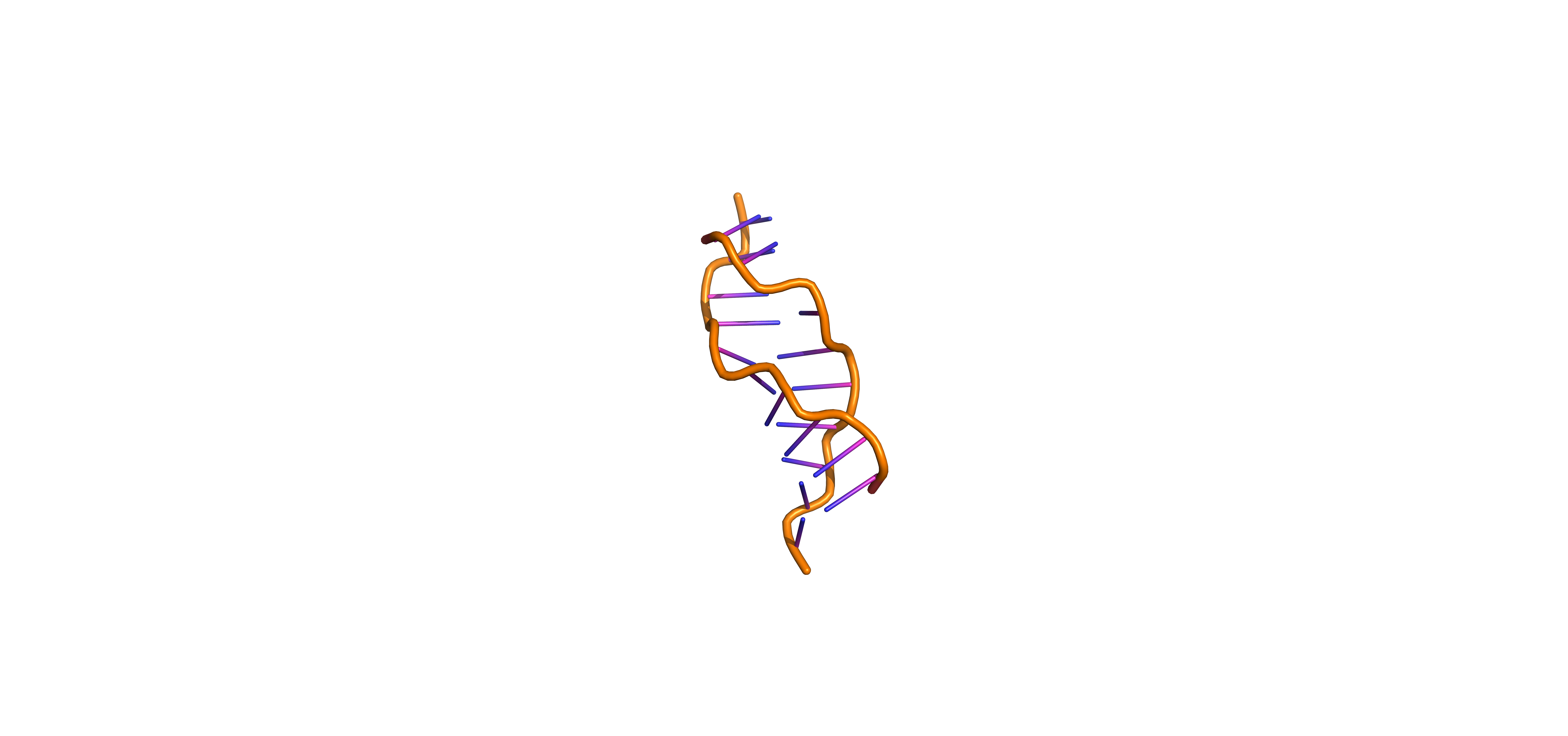
С помощью программного пакета fiber были получены структуры ДНК, состоящие из 5 повторов "GATC" в A, B и Z формах. Общий промт: fiber -тип_формы_ДНК -seq=gatc -rep=5 gatc-тип_формы_ДНК.pdb



Ссылки на файлы: A форма, B форма, Z форма
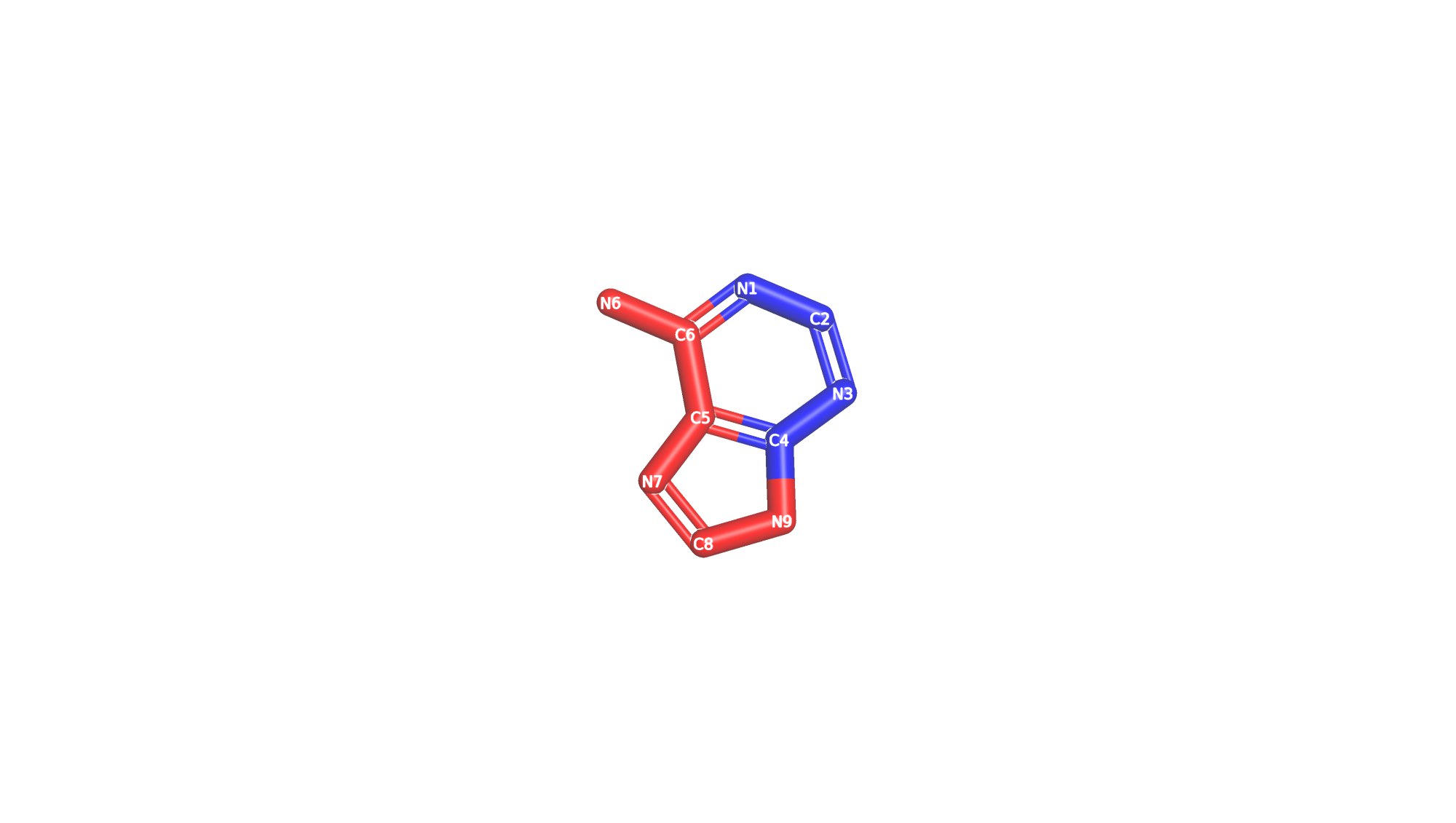
Далее был взят нуклеотид (A6) с предложенным мне основанием (аденин) на эксперементальной структуре B-формы ДНК, изучено, какие атомы основания смотрят в сторону большой бороздки, а какие в сторону малой. В итоге атомы, направленные в сторону малой бороздки были раскрашены в программе PyMol синий цвет (номера А6.С2, А6.N3, A6.C4, A6.N9), а в сторону большой - в красный (номера A6.N1, A6.C5, A6.N6, A6.C6, A6.N7, A6.C8; Рис. 4).
Ниже приведена информация о разных формах ДНК (Табл. 1)
| А-форма | В-форма | Z-форма | |
|---|---|---|---|
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали(Å) | 28.0 | 33.8 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16.5 | 20.6 | 22.9 |
| Ширина малой бороздки | 9.6 | 13.2 | 9.9 |
Для анализа мне была предложена структура с PDB ID 2cv0. Для начала был скачан файл с pdb, передён в старый pdb формат и с помощью программ find_pair и analize пакета 3DNA я изучила их вторичную структуру. Стебли тРНК образованы основаниями: 501-507 и 572-566; 549-553 и 565-561; 538-544 и 532-526; 510-514 и 525-521. Ниже приведена информация о неканонических парах и дополнительных водородных связях
2 G-*---U [2] O6 - N3 2.85 N1 - O2 2.91
13 U-**--A [2] O2 - N6 3.17 N3 - N7 3.19
14 U-**+-G [1] O2 - N2 2.70
15 A-**--C [1] N6 - O2 3.15
21 A-**--G [2] N6 - O6 3.10 N1 - N1 2.88
25 U-*---G [2] O2 - N1 2.65 N3 - O6 2.96
26 U-**--A [3] O3'- O2' 2.87 O2'- N3 3.22 O2 * N1 3.55
27 A-**--A [2] N6 - N3 3.03 N1 - O2' 2.74
28 G-**+-C [2] N1 - O2 3.24 N2 - N3 2.95
13 (0.007) ....>C:.554_:[..U]U-**--A[..A]:.558_:C<.... (0.003) |
14 (0.009) ....>C:.555_:[..U]U-**+-G[..G]:.518_:C<.... (0.014) x
28 (0.024) ....>C:.515_:[..G]G-**+-C[..C]:.548_:C<.... (0.013) x
29 (0.012) ....>C:.519_:[..G]G-----C[..C]:.556_:C<.... (0.003) +