1. Поиск гена дельта-субъединицы АТФ-синтазы
В текстовом файле с белковыми последовательностями (в формате FASTA) был найден белок, аннотированный как δ-субъединица АТФ-синтазы (ATP synthase F1 subunit delta). С помощью файла аннотированных последовательностей генома (GBFF) было установлено, что его ген располагается на 9 хромосоме геномной сборки VulVul3 (идентификатор нукдеотидной записи - NC_132788.1). На рисунке 1 положение гена на 9 хромосоме: зелеными прямоугольниками - экзоны гена и интроны между ними, фиолетовыми - РНК-транскрипт, красным - кодирующие участки (CDS).
2. Поиск гомологов гена дельта-субъединицы АТФ-синтазы с помощью Blast
Поиск гомологов Blast для лисы, относящейся к вторичноротым животным, производился по достаточно удалённому таксону Пауки (Araneae, Tax ID 6893) с помощью базы данных - refseq_genomes. Как и ожидалось, поиск гомологов с помощью megablast для сильно похожих последовательностей результатов не дал, ведь изучаемые нуклеотидные последовательности, во-первых, дивергировали давно, во-вторых, стоит учитывать, что множественные мутации в слабо подверженных отбору интронах и синонимичные замены, не изменяющие аминокислотную последовательность, будут сильно изменять нуклеотидную последовательность и ухудшать работу megablast, делая его не лучшим алгоритмом для поиска дальних гомологов.
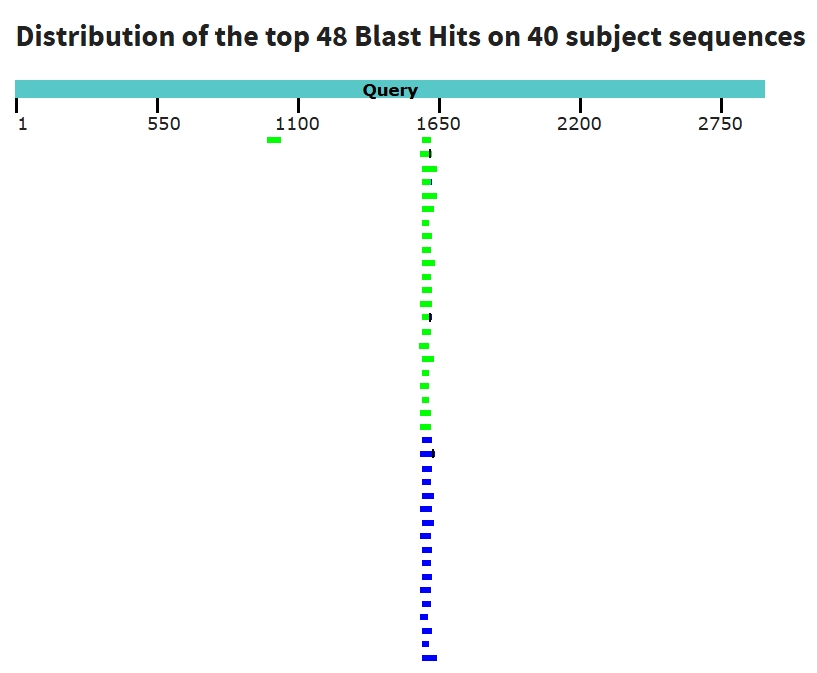
Если же вместо megablast использовать алгоритм blastn (для отчасти схожих последовательностей) и уменьшить word size до 7, значение e-value = 0.05 оставить прежним, то количество находок возрастёт до 40. На рисунке 2 графически обозначены хиты, выровнявшиеся на исходную последовательность гена дельта-субъединицы АТФ-синтазы. Их длина совсем небольшая, e-value таких находок не превышает 5e-04.
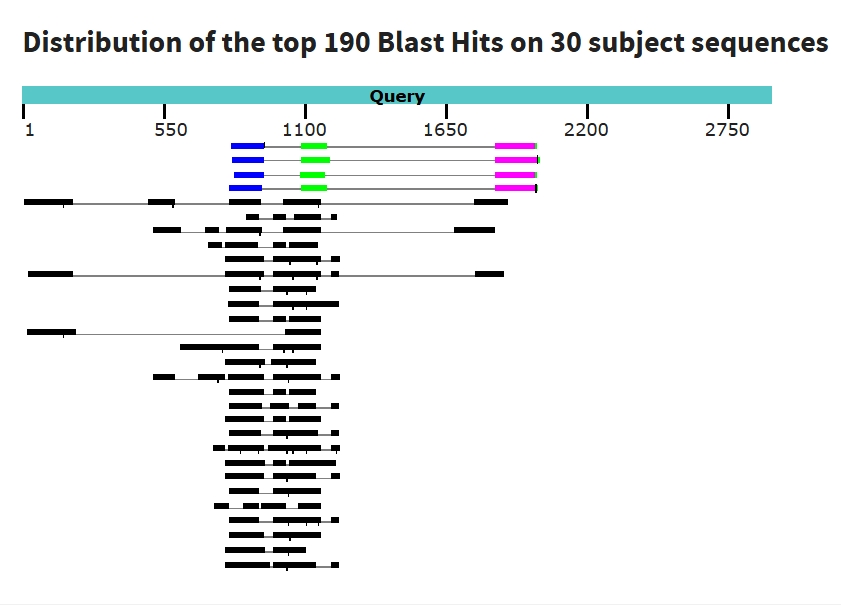
Для нахождения более достоверных гомологов будем использовать другой алгоритм - tblastx, который переводит и запрос (нуклеотидную последовательность), и базу данных ДНК в 6 белковых последовательностей и сравнивает их. Результат работы - 30 находок, но уже с большей длиной хита, меньшим значением e-value = 3e-26 (более статистически значимо) и большим весом выравнивания. Можно заметить, что хиты хорошо выравниваются с экзонами гена, тем самым прослеживается соответствие между более консервативными участками query и экзон-интронной организацией гена.
Задание 3. Поиск генов рРНК по далекому гомологу
Следующей задачей был поиск гомологов структурных рРНК E. coli в геноме Vulpes vulpes. Исследовали 16S-рРНК (компонент малой субъединицы, ответственный за распознавание мРНК) и 23S-рРНК (компонент большой субъединицы, обладающий пептидилтрансферазной активностью).
Для начала нужно проиндексировать последовательности генома для работы локального BLAST (опция -in для имени входного файла генома, -dbtype nucl для нуклеотидной базы данных).
makeblastdb -in .\ncbi_dataset\data\GCF_048418805.1\GCF_048418805.1_VulVul3_genomic.fna -dbtype nucl
Далее используем алгоритм blastn для поиска гомологичных последовательностей в исследуемой сборке VulVul3, в качестве запросов (query) использовались нуклеотидные последовательности 16S и 23S рРНК E. coli в fasta-формате.
blastn -task blastn -query .\16S_Ecoli.fasta.txt -db .\ncbi_dataset\data\GCF_048418805.1\GCF_048418805.1_VulVul3_genomic.fna -out Vulpes_Ecoli_16S.txt -outfmt 7
blastn -task blastn -query .\23S_Ecoli.fasta.txt -db .\ncbi_dataset\data\GCF_048418805.1\GCF_048418805.1_VulVul3_genomic.fna -out Vulpes_Ecoli_23S.txt -outfmt 7
Пояснения:
-task blastn — программа по умолчанию использует megablast
-query .\16S_Ecoli.fasta.txt — имя fasta-файла с последовательностью 16S рРНК E. coli.
-db .\GCF_048418805.1_VulVul3_genomic.fna — имя раннее созданной базы данных по последовательности генома Canis lupus dingo.
-out Vulpes_Ecoli_23S.txt — имя выходного файла blastn в формате 7
Файл выдачи blastn по 16S рРНК E. coli
Файл выдачи blastn по 23S рРНК E. coli
В случае поиска гомологов 16S рРНК blastn нашел 99 хитов, для 23S рРНК - 225 хитов. После фильтрации хитов с e-value < 0.05 для 16S рРНК осталось 44 хита - все они являются гомологами участка 1494-1536 у E.coli и комплементарны разным участкам хромосом и скэффолдов в геноме лисы. На рисунке 4 графически изображён результат (взят скэффолд NW_027325924.1, но для остальных последовательностей картина выглядит аналогично):
Для 23S рРНК после такой же фильтрации осталось 210. Для визуализации гомолога я взяла скэффолд NW_027325867.1, на котором нашлось 3 комплементарных участка:
Задание 4. Карты локального сходства двух геномов
Для сравнительного анализа были отобраны референсные геномы (хромосомы) двух видов рода Mycoplasmoides: Mycoplasmoides genitalium strain G37 (RefSeq sequence ID: NZ_CP159789.1) и Mycoplasmoides pneumoniae (RefSeq sequence ID: NZ_LR214945.1). Сравнение проводилось тремя разными алгоритмами: megablast, blastn, tblastx, результаты их работы представлены в виде карт локального сходства (dotplot).


Пояснение: нетрудно заметить, что предназначенный для поиска близких гомологов алгоритм blastn выровнял больше сходных участков и выдал более информативный результат, чем megablast, использующийся в основном для поиска почти идентичных последовательностей. Также можем наблюдать, что большая часть участков генома хорошо выравнивается, образуя почти сплошную диагональ, за исключением некоторых "разрывов", которые могут свидетельствовать о делециях (выделены синей рамкой).
Особо интересны участки, соответствующие различным генетическим перестройкам. Выделенный красной рамкой участок является транслокацией. Участок, выделенный коричневой рамкой - дубликацией. Многочисленные точки свидетельствуют о частых повторах в сравниваемых геномах.