Гомологичное моделирование комплекса белка с лигандом
Шестой семестр
Выравнивание лизоцима форели (с известной структурой 1LMP) с лизоцимом LYS_CLOAB из Clostridium acetobutylicum.
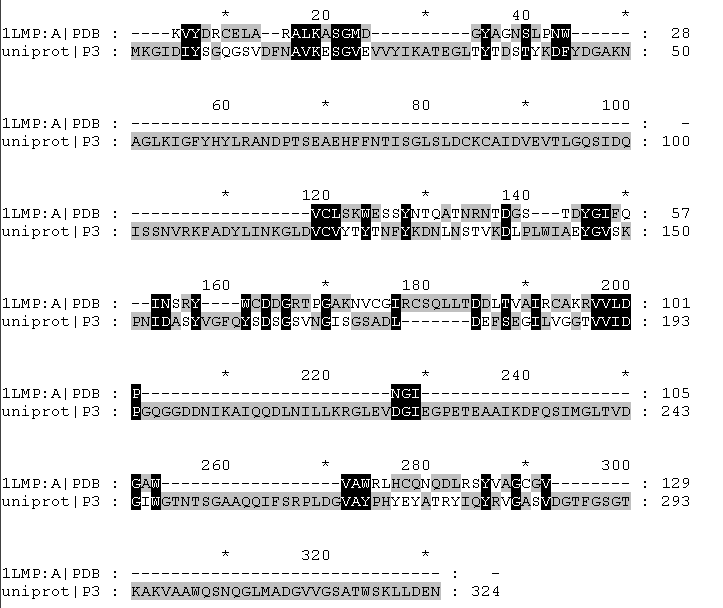
Файл выравнивания был сохранен с раширением .pir и модифицирован. Номера остатков и названия цепей в файле с координатами изменены.
Для анализа были выбраны 3 водородные связи, изображенные на рисунке:
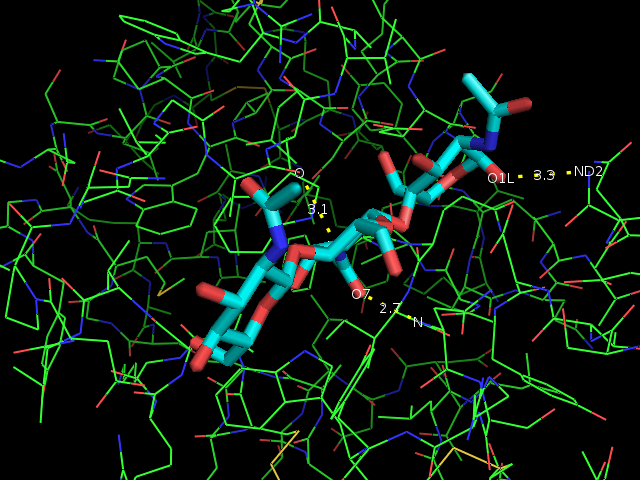
В белке форели:
N:59:A, O7:130:B
O:107:A, N2:130:B
N:109:A, O6:130:C
В гомологичном белке:
N:154:A, O7:325:B
O:245:A, N2:325:B
N:279:A, O6:325:C
Для моделирования исползовался следующий скрипт: lys_cloab.py
mod9v7 lys_cloab.pyВ результате моделирования были получены 5 структур:
seq01.pdb , seq01.pdb , seq03.pdb, seq04.pdb, seq05.pdb
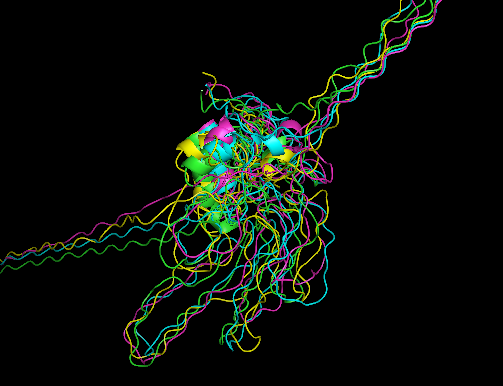
С помощью сервиса WHATIF (swift.cmbi.ru.nl/servers/html/index.html) оценим качество структур.
| Модель | Ramachandran Z-score | Z-score for bond angles |
| 1lmp | -0.965 | 2.718 |
| seq1 | -4.081 | 5.978 |
| seq2 | -4.467 | 6.613 |
| seq3 | -4.125 | 6.29 |
| seq4 | -4.945 | 6.334 |
| seq5 | -4.444 | 5.780 |
Чем меньше Z-score, тем лучше структура. Самые лучшие структуры 1ая и 5ая.