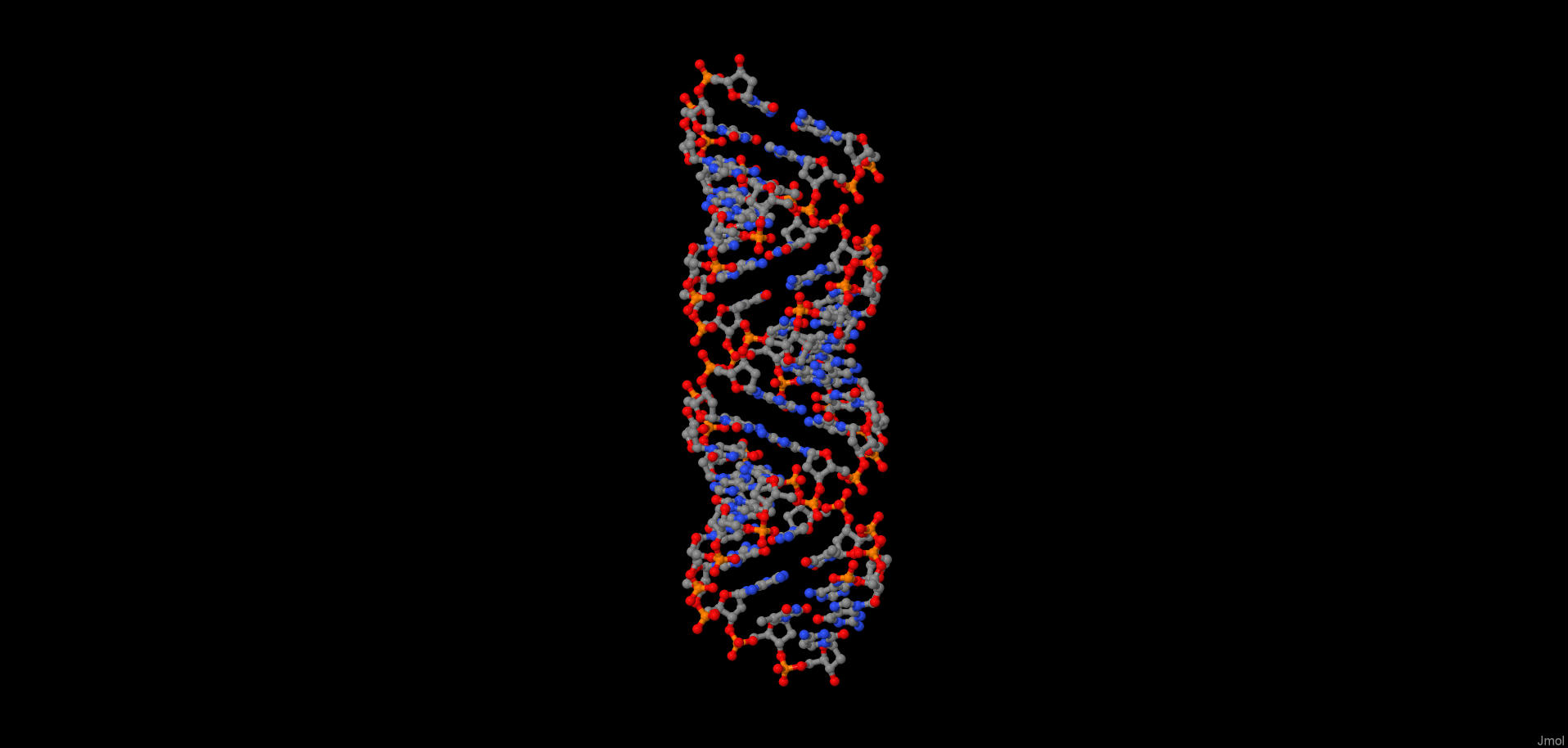
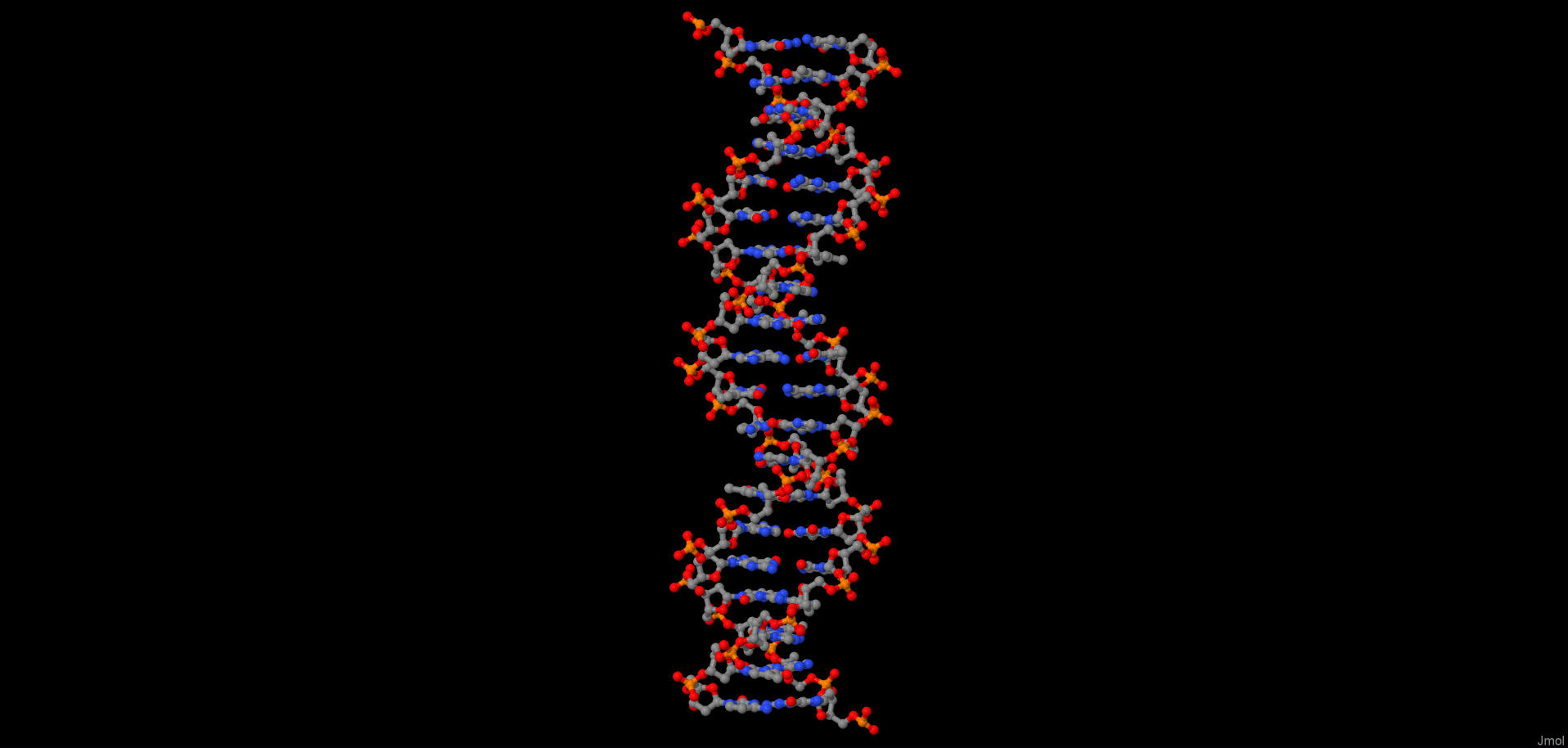
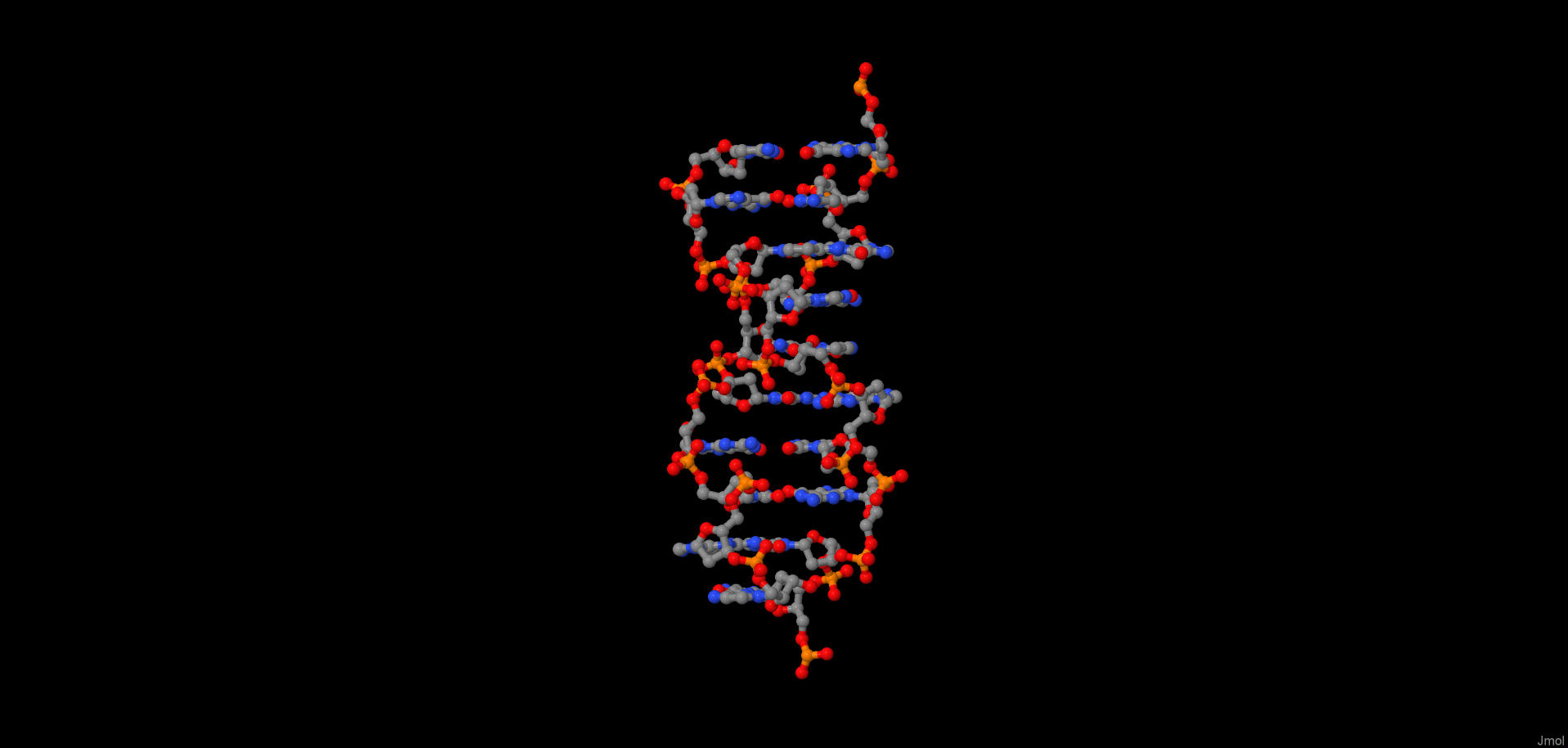
С помощью инструментов пакета 3DNA были построены модели структур A-, B- и Z-формы ДНК. Пакет 3DNA - один из популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот.
С помощью программы fiber пакета 3DNA были построены A-, B- и Z-формы дуплекса ДНК (файлы gatc_a.pdb, gatc_b.pdb, gatc_z.pdb, содержащие смоделированные структуры ДНК в А, В и Z формах соответственно),
последовательность одной из нитей которого представляет собой 5 раз повтореную последовательность "gatc".
Изображение форм ДНК
Для визуального определения принадлежности атомов аденина большой и малой бороздке разных форм ДНК структуры были открыты в JMol. В таблице ниже приведены координаты соответствующих атомов.
| Атомы тимина в трех формах ДНК |
|---|
| A-форма | B-форма | Z-форма |
| В сторону большой бороздки обращены атомы: | N1, C2, N3, C4 | C5, C6, N6, N7, C8 | C4, C5, C6, O6, N7, C8, N9 |
| В сторону малой бороздки обращены атомы: | N6, N7, C8, N9 | C2, N3, C4, N9 | C2, N2 |
| Остальные атомы основания: | C5, C6 | N1 | N1, N3 |
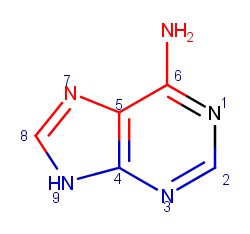
С помощью MarvinSketch были получены изображения аденина, где красным цветом выделены атомы, смотрящие в сторону большой бороздки, а синим в сторону малой.
С помощью программы Jmol я также сравнила основные спиральные параметры разных форм ДНК. Результаты исследования приведены в таблице 2.
| Форма ДНК |
A |
B |
Z |
| Тип спирали |
правая |
правая |
левая |
| Шаг спирали (A) |
28.03 |
28.03 |
43.50 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки |
1.681 нм |
1.721 нм |
1.830 нм |
| Ширина малой бороздки |
0.798 нм |
1.169 нм |
0.720 |
Для A-формы ширина большой бороздки измерялась от фосфата C (8 нуклеотид, цепь A) до фосфата T (35 нуклеотид, цепь В); ширина малой бороздки - от фосфата G (9 нуклеотид, цепь A) до фосфата A (26 нуклеотид, цепь B).
Для B-формы ширина большой бороздки измерялась от фосфата C (8 нуклеотид, цепь A) до фосфата A (30 нуклеотид, цепь В); ширина малой бороздки - от фосфата A (6 нуклеотид, цепь A) до фосфата T (39 нуклеотид, цепь B).
Для Z-формы ширина большой бороздки измерялась от фосфата C (12 нуклеотид, цепь A) до фосфата C (26 нуклеотид, цепь В); ширина малой бороздки - от фосфата G (9 нуклеотид, цепь A) до фосфата G (35 нуклеотид, цепь B).
С помощью команды Torsion в программе Jmol я измерила торсионные углы нуклеотида, содержащего аденин, в A- и B-формах ДНК. Результаты приведены в таблице 3.
| Таблица 3. Сравнение торсионных углов нуклеотида, содержащего аденин, в разных формах ДНК |
| Мои значения |
| Форма ДНК |
α |
β |
γ |
δ |
ε |
ζ |
Χ |
| А |
64.1 |
174.8 |
41.7 |
79.0 |
-147.8 |
-75.1 |
-157.2 |
| В |
85.9 |
136.4 |
31.1 |
143.4 |
140.8 |
-160.5 |
-98.0 |
| Значения из презентации |
| А |
62 |
173 |
52 |
88 |
178 |
-50 |
-160 |
| В |
63 |
171 |
54 |
131 |
155 |
-90 |
-117 |
С помощью программ find_pair и analyze пакета 3DNA я сравнила значения торсионных углов в структурах А-, В- и Z-форм ДНК и тРНК с идентификатором 1EHZ. Для тРНК приведены средние значения торсионных углов. Результаты
представлены в файлах ниже.
DNA, самый отклоняющийся 4-й тимин.
RNA, самый отклоняющийся 11-й гуанин.
Рассмотрим структуру тРНК. Из выходного файла find_pair можно выяснить позиции нуклеотидов, образующих водородные связи.
Акцепторный стебель состоит из участка 501-507 и комплементарного ему участка 566-572:
|
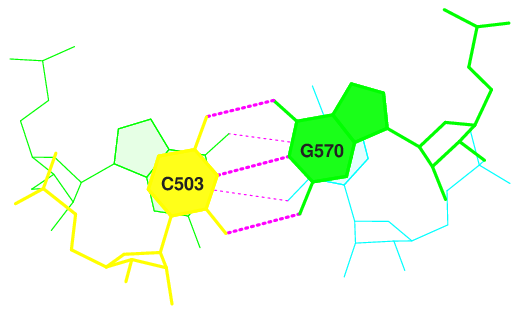
(0.011) C:.501_:[..G]G——-C[..C]:.572_:C (0.008)
(0.008) C:.502_:[..G]G-*—-U[..U]:.571_:C (0.007)
(0.005) C:.503_:[..C]C——-G[..G]:.570_:C (0.004)
(0.005) C:.504_:[..C]C——-G[..G]:.569_:C (0.008)
(0.007) C:.505_:[..C]C——-G[..G]:.568_:C (0.009)
(0.006) C:.506_:[..C]C——-G[..G]:.567_:C (0.004)
(0.004) C:.507_:[..A]Ax——U[..U]:.566_:C (0.004) |
Т-стебель из 549-553 и 561-565:
|
(0.008) C:.549_:[..G]G——-C[..C]:.565_:C (0.003)
(0.006) C:.550_:[..G]G——-C[..C]:.564_:C (0.005)
(0.005) C:.551_:[..G]G——-C[..C]:.563_:C (0.002)
(0.003) C:.552_:[..G]G——-C[..C]:.562_:C (0.004)
(0.008) C:.553_:[..G]G——xC[..C]:.561_:C (0.004) |
D-стебель из 510-513 и 522-525:
|
(0.012) C:.510_:[..G]G——-C[..C]:.525_:C (0.005)
(0.005) C:.511_:[..U]U——-A[..A]:.524_:C (0.006)
(0.005) C:.512_:[..C]C——-G[..G]:.523_:C (0.010)
(0.007) C:.513_:[..U]U-*—xG[..G]:.522_:C (0.013) |
Антикодоновый стебель из 539-544 и 526-531:
|
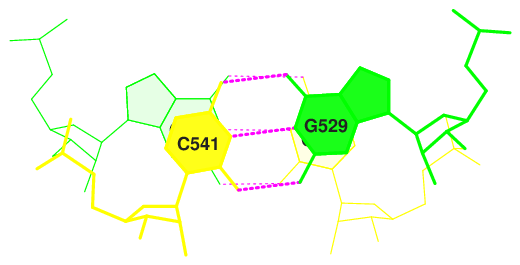
(0.007) C:.539_:[..G]G——-C[..C]:.531_:C (0.003)
(0.009) C:.540_:[..G]G——-C[..C]:.530_:C (0.007)
(0.003) C:.541_:[..C]C——-G[..G]:.529_:C (0.006)
(0.008) C:.542_:[..C]C——-G[..G]:.528_:C (0.007)
(0.007) C:.543_:[..G]G——-C[..C]:.527_:C (0.009)
(0.011) C:.544_:[..A]Ax*—-G[..G]:.526_:C (0.009) |
Остальные водородные связи, стабилизирующие третичную структуру тРНК (не образуют стебли):
|
(0.009) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.014)
(0.024) C:.515_:[..G]Gx**+xC[..C]:.548_:C (0.013)
(0.012) C:.519_:[..G]Gx—-xC[..C]:.556_:C (0.003) |
Также в структуре есть 5 неканонических пар оснований: 520G-571U, 513U-522G, 544A-526G, 555U-518G, 538A-532C.
В выходном файле выделим данные о величине площади "перекрывания" двух последовательных пар азотистых оснований. Пары с наибольшими значениями имеют большую площадь "перекрывания", наиболее вероятное стекинг-взаимодействие:
|
step i1-i2 i1-j2 j1-i2 j1-j2 sum
2 GC/GU 7.14( 4.30) 0.00( 0.00) 0.00( 0.00) 6.63( 4.10) 13.76( 8.40)
17 GC/GC 6.19( 3.17) 0.00( 0.00) 0.00( 0.00) 6.51( 3.37) 12.69( 6.53)
30 GC/GU 7.17( 4.39) 0.00( 0.00) 0.00( 0.00) 6.49( 4.01) 13.66( 8.40)
43 AG/CC 4.74( 2.33) 0.00( 0.00) 0.00( 0.00) 5.68( 2.31) 10.42( 4.64) |
Для получения изображений использовались следующие запросы:
ex_str -i stacking.pdb stepi.pdb, где i - номер шага в stacking.pdb
stack2img -cdolt stepi.pdb stepi.ps - получение изображения
convert stepi.ps stepi.png - конвертирование изображения в png
Шаг 2
Шаг 17
Шаг 30
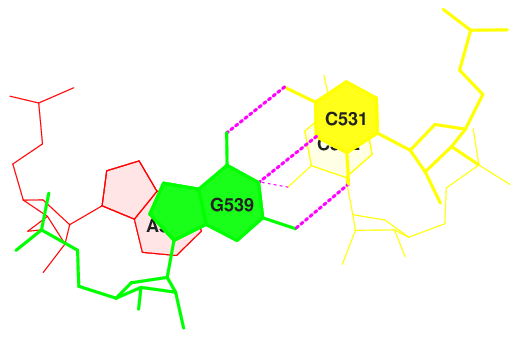
Шаг 43
Источники:
MarvinSketch
©
Avdiunina Polina, 2015