Практикум 8
Резюме: В ходе работы над данным практикумом была продолжена работа с геномом косатки (Orcinus Orca). Первая часть - в практикуме 7. Освоены базовые навыки работы с веб-интерфейсом BLAST и standalone BLAST.
Поиск гена дельта-субъединицы АТФ-синтазы
С помощью текстового поиска в файле с последовательностями белков (см пр7, пункт про файлы) был найден белок, аннотированный как δ-субъединица АТФ-синтазы (ATP synthase F1 subunit delta). Далее было установлено, что этот ген располагается на 3 хромосоме (напомню, что эта сборка довольно хорошая и содержит целые последовательности всех хромосом - см. пр7). Идентификатор нуклеотидной записи - NC_064561.1.
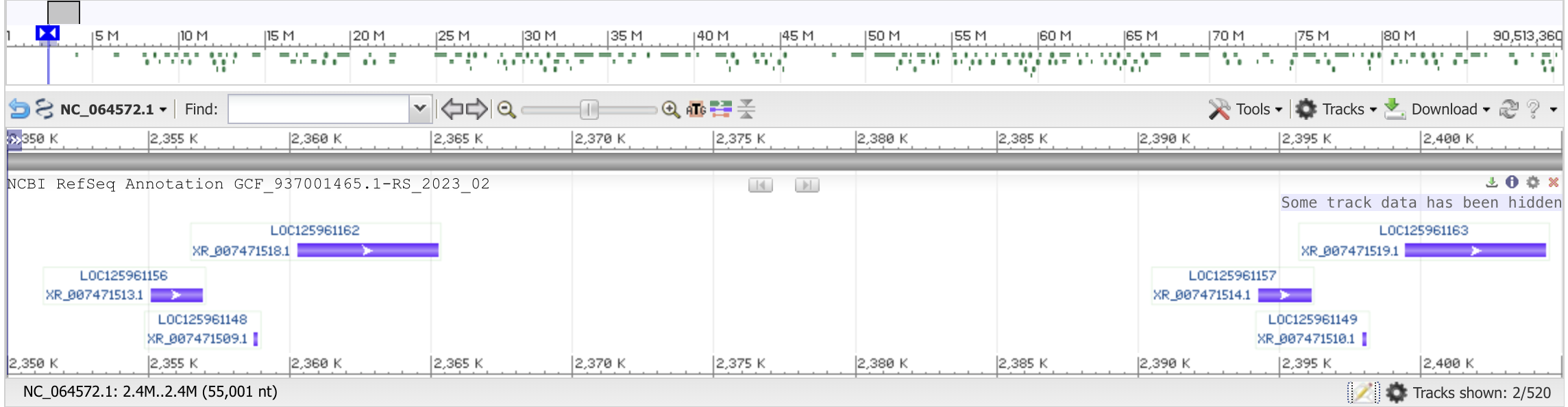
Пояснения к Рис.1 (подробнее)
Верхнее поле - карта целой хромосомы (зеленые прямоугольники - гены, синие штуки показывают положение показываемого фрагмента относительно всей последовательности). Ниже располагается поле выделенного фрагмента. Зеленым цветом обозначен целевой ген, выделены интроны и экзоны (более светлый блок - нетранслируемая область, насколько я понял). Стрелочки вправо обозначают то, что ген нахродится на (+)цепи (в случае интронов - направление сплайсинга). Еще ниже - какие-то графики, тоже связанные с экзон-интронной организацией гена.
Поиск гомологов гена дельта-субъединицы АТФ-синтазы косатки
Следущей задачей стало найти в относительно отдаленном таксоне последовательности, гомологичные гену δ-субъединицы АТФ-синтазы из Orcinus orca. Так как косатки относятся к вторичноротым, в качестве таксона для поиска я взял надсемейство Apoidea (это клада перепончатокрылых, к которой относятся, в том числе, пчелы). База данных, которая использовалась - refseq_genomes. Если искать в NCBI Datasets, то для данной клады в RefSeq включены 28 сборок геномов, однако в выдаче BLAST пишет, что их 29 (в целом, небольшая разница). Поиск гомологов с помощью blastn/megablast не дал результатов (вне зависимости от параметров). Это неудивительно, так как гены эукариот содержат интроны, на которые слабо действует отбор (а значит они сильно вариабельны в далеких таксонах, что делает поиск по нуклеотидным последовательностям эукариот плохим способом поиска гомологов). Также, blastn не учитывает вырожденность генетического кода (одна аминокислота может кодироваться несколькими кодонами). А значит множество синонимичных мутаций, меняющие нуклеотидную последовательность, но слабо подверженные отбору, будут ухудшать работу blastn.
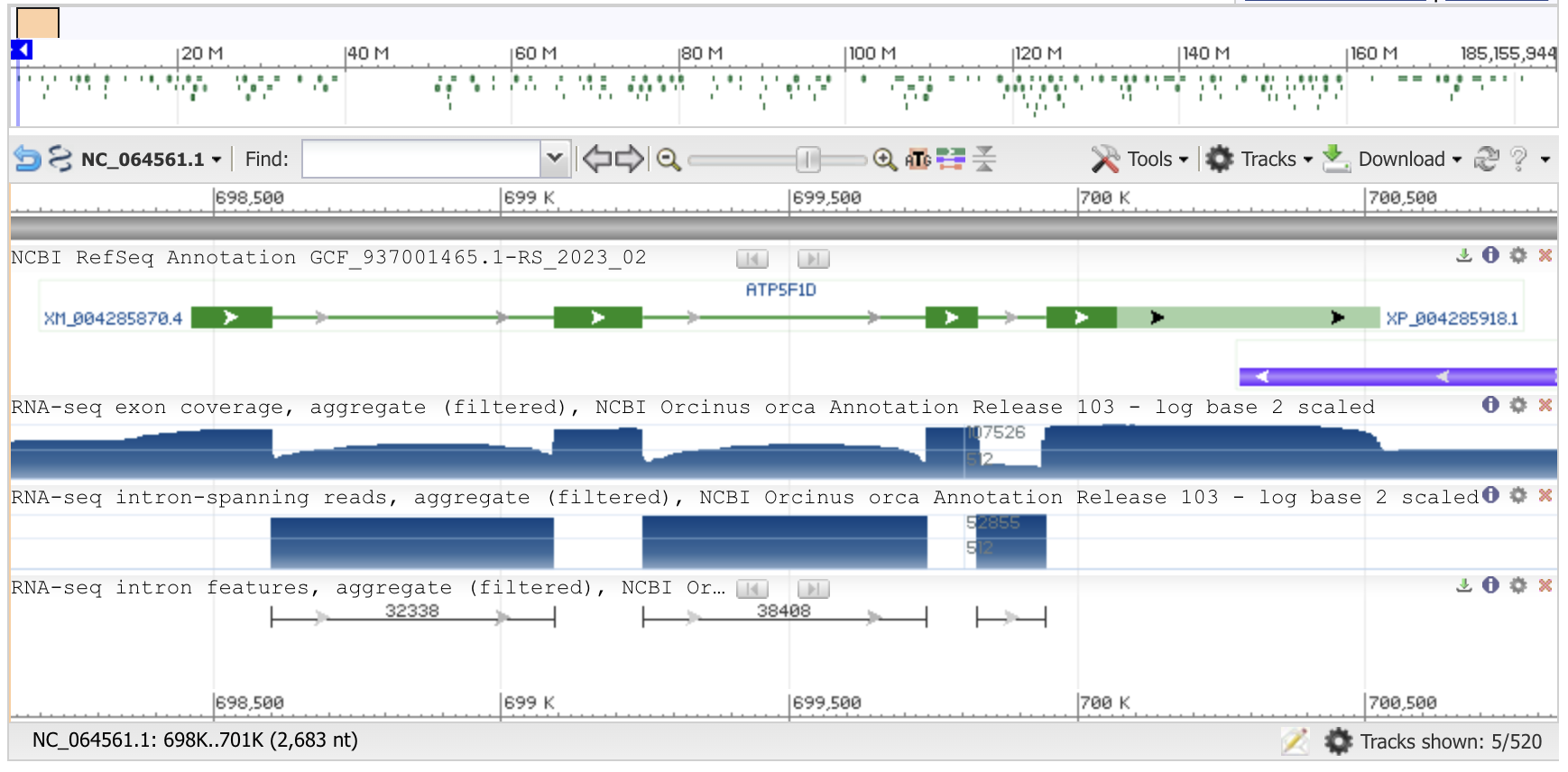
Дальше я решил использовать метод tblactx, так как мне хотелось давать на вход нуклеотидную последовательность, а не белковую. Были использованы следующие параметры: word size - 3; threshold - 0.05. Количество находок - 28 (соответсвует количеству сборок, включенных в RefSeq согласно NCBI Datasets). Мои ожидания были примерно такие же, так как АТФ-синтаза - это очень важный белок, а значит его аминокислотная последовательность должна быть довольно консервативной (то есть гомологи должны находиться без проблем).
При уменьшении word_size до 2, находятся еще две последовательности (какие-то другие хромосомы из этих же видов), но их E-value значительно больше, чем у других (0.048 по сравнению с 2e-09 - еле перешли порог), а значит значимость меньше. Четко прослежвается соответствие выравниваемых участков query с экзон-интронной организацией гена (Рис. 2).Поиск генов рРНК по далекому гомологу
Следующей задачей стало найти в сборке генома косатки гены рРНК по далекому гомологу - генам рРНК из E. coli, используя standalone (локальный) BLAST. Поиск проводился по последовательностям генов 23S и 16S рРНК. Обе молекулы входят в состав рибосомы (16S - малой субъединицы, 23S - большой), выполняют структурную функцию. 16S рРНК участвует в инициаторном скрининге за счет способности комплементарно связываться с последовательностью Шайна-Дальгарно в мРНК. 23S рРНК участвует в пептидилтрансферазной реакции (рибозим).
Для начала нужно было проиндексировать последовательности сборки, чтобы получить локальную базу данных. Команда:
makeblastdb -in GCF_937001465.1_mOrcOrc1.1_genomic.fna -dbtype nucl
Поиск производился с помощью blastn, так как, во-первых, запросом здесь является нуклеотидная последовательность, во-вторых, E.coli и O.orca - это очень далекие по систематическому положению организмы, поэтому даже такие консервартивные последовательности, как рРНК будут несколько отличаться между ними (поэтому megablast не подходит). Размер слова - 7 (повысим шансы найти что-то), пороговое значение E-value - 0,05 (остальные параметры по умолчанию). Команды:
blastn -task blastn -query 16S.txt -db GCF_937001465.1_mOrcOrc1.1_genomic.fna -out 16_blast7.out -word_size 7 -evalue 0.05 -outfmt 7
blastn -task blastn -query 16S.txt -db GCF_937001465.1_mOrcOrc1.1_genomic.fna -out 16_blast.out -word_size 7 -evalue 0.05
blastn -task blastn -query 23S.txt -db GCF_937001465.1_mOrcOrc1.1_genomic.fna -out 23_blast7.out -word_size 7 -evalue 0.05 -outfmt 7
blastn -task blastn -query 23S.txt -db GCF_937001465.1_mOrcOrc1.1_genomic.fna -out 23_blast.out -word_size 7 -evalue 0.05
Результаты:
- 16S (текст | таблица). Количество находок: 2, количество гомологичных участков (hits): 7.
- 23S (текст | таблица). Количество находок: 10, количество гомологичных участков (hits): 36.
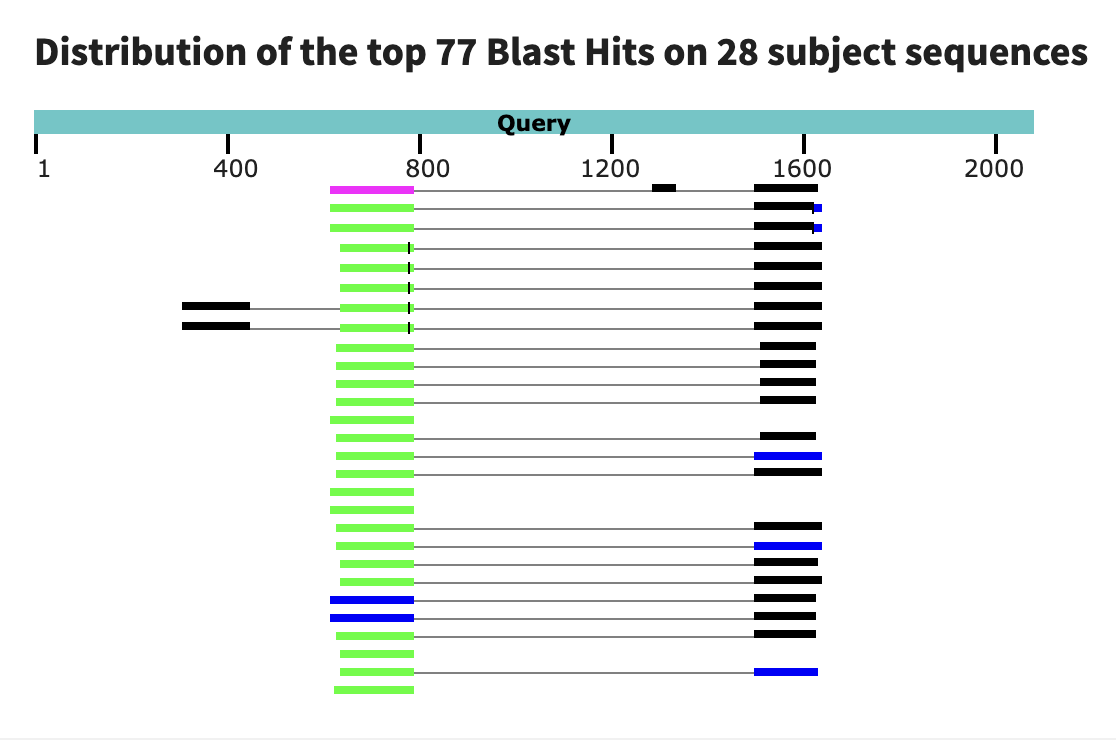
Для обеих рРНК наиболее значимые хиты были на 14 хромосоме, причем некоторые принадлежали одному кластеру (Рис. 3). Можно отметить, что при поиске по 23S рРНК одной из находок стала последовательность митохондриального генома (нашелся гомолог бактериальной 23S рРНК - 16S митохондриальная рРНК).