Практикум 7
Резюме: В ходе работы над данным практикумом были освоены базовые навыки работы с базой данной NCBI Datasets Genome и предпринята попытка анализа cборки генома косатки (Orcinus Orca). Продолжение работы с геномом костаки - в практикуме 8
Выбор сборки
По какой-то причине я решил, что буду искать сборку генома кого-то из дельфинов (они классные). Прочитав пару слов про систематику, я сделал запрос по семейству Delphinidae (морские дельфины - marine dolphins). По этому запросу было найдено 33 сборки, из которых 7 имели аннотированные гены. Я остановился на референсной сборке генома косатки (Orcinus orca), потому что мне понравилось ее английское название - killer whale (Рис. 1). Косатки - это вид-космополит морских сверх-хищников, единственный представитель китообразных, питающийся теплокровными животными. Как мне кажется, наиболее примечательна популяционная структура косаток. Так, только среди антарктических косаток встречается 5 форм (рас/подвидов), отличающихся по морфологии, кормовой базе и социальным особенностям [источник]. Еще, косатки - это одни из очень немногих животных, для самок которых характерна менопауза (помимо косаток менопазуза характерна для некоторых других видов зубатых китов и для некоторых приматов). "Бабушки-косатки", прошедшие менопаузу помогают молодым особям в своих семьях и увеличивают их шансы на выживание [источник]. Косатки имеют 44 хромосомы в диплоидном наборе.

Уровень сборки - Chromosome, то есть сборка генома содержит последовательность одной или нескольких хромосом (либо полностью секвенированная хромосома без гэпов, либо хромосома, содержащая гэпы). Также в сборке могут быть нелокализированные скэффолды (последнее, как раз, характерно для этой сборки - она содержит 425 нелокализированных скэффолда).
Характеристики сборки
Таблица 1. Некоторые характеристики сборки
| Характеристика | Значение |
| Идентификатор GenBank | GCF_937001465.1 |
| Идентификатор RefSeq | GCF_937001465.1 |
| Общий размер генома (п.н.) | 2.6 Gb |
| Число фрагментов генома в сборке | 447 (448 - см. последний пункт) |
| N50 для контигов | 45.6 Mb |
| L50 для контигов | 16 |
| N50 для скэффолдов | 114.2 Mb |
| L50 для скэффолдов | 9 |
Параметр N50 означает длину контига/скэффолда, для которого половина (50%) всех нуклеотидов сборки содержится в контигах/скэффолдах такой и большей длины. Параметр L50 - это число контигов/скэффолдов (наименьшее), в которых содержится половина (50%) всех нуклеотидов сборки.
Таким образом, хотя сборка и не обладает наивысшим уровнем, мне кажется, она довольно хорошо отражает биологическую информацию о геноме косатки. Например, все 22 хромосомы гаплоидного набора аннотированы, а также эта сборка признана референсной (то есть проверена человеком и используется как стандарт). Согласно BUSCO анализу в сборке содержится 97,8% предполагаемых генов - поле Complete (если я правильно понимаю суть BUSCO анализа), что, несомненно, является очень хороших показателем.
Файлы сборки
С помощью NCBI FTP были скачаны следующие файлы сборки:
GCF_937001465.1_mOrcOrc1.1_genomic.fna.gz - Нуклеотидные последовательности генома (в формате FASTA)
GCF_937001465.1_mOrcOrc1.1_genomic.gbff.gz - Последовательности генома с аннотацией (в формате GBFF)
GCF_937001465.1_mOrcOrc1.1_protein.faa.gz - Последовательности белков (в формате FASTA)
Органнелы
В выбранной сборке есть геном митохондрии. Понять это можно, посмотрев в поле Chromosomes (Рис. 2), там же есть ссылки на записи в GenBank и RefSeq.
Таблица 2. Некоторые характеристики сборки митохондриального генома косатки
| Характеристика | Значение |
| Идентификатор GenBank | OW443360 |
| Идентификатор RefSeq | NC_064559 |
| Тип органеллы | Митохондрия |
| Число кодирующих последовательностей (CDS) | 13 |
| Число генов рРНК | 2 |
| Число генов тРНК | 22 |
| Число псевдогенов | 0 |
Данные были получены "ленивым путем": сочетанием клавиш Ctr+F на странице записи RefSeq и несложными арифметическими операциями.
Анализ длин фрагментов генома
C помощью скриптов на Python был проанализирован файл с нуклеотидными последовательностями генома. Оказалось, что в этом файле лежат последовательности целых хромосом (22 аутосомы и X), последовательность митохондриального генома и 425 последовательностей нелокализированных скэффолдов. Всего 448 фрагментов (в таком случае, видимо, митохондриальный геном не считается скэффолдом, наверное, потому что выделение и секвенирование митохондриальной ДНК происходит отдельно от от ядерного генома). Было построено три графика:
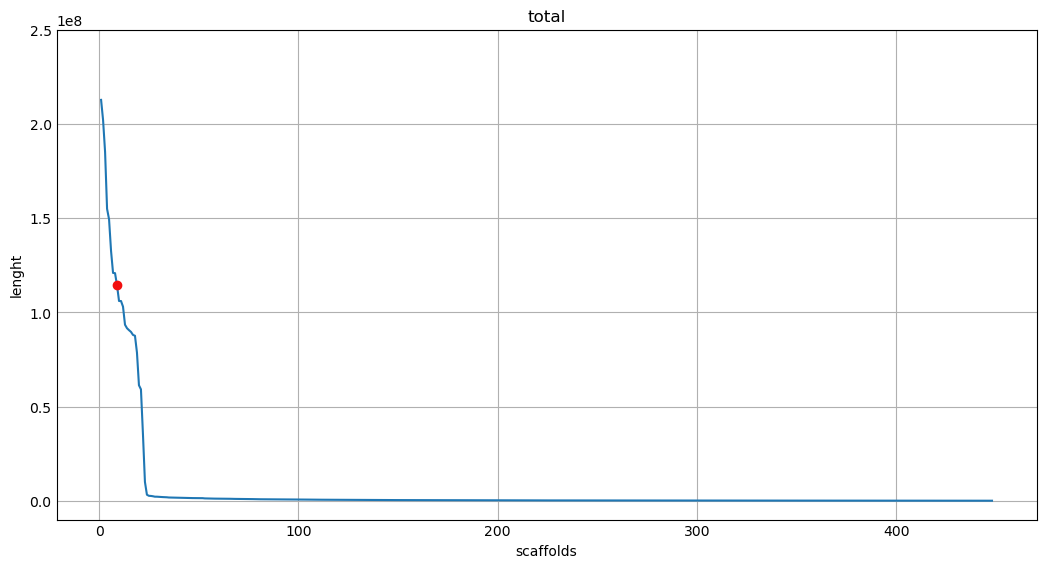
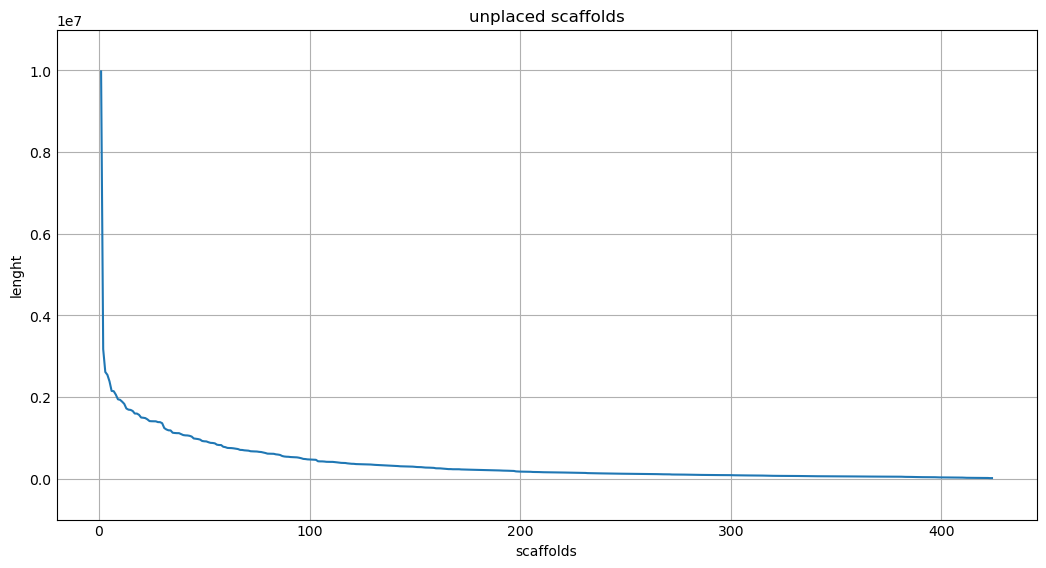
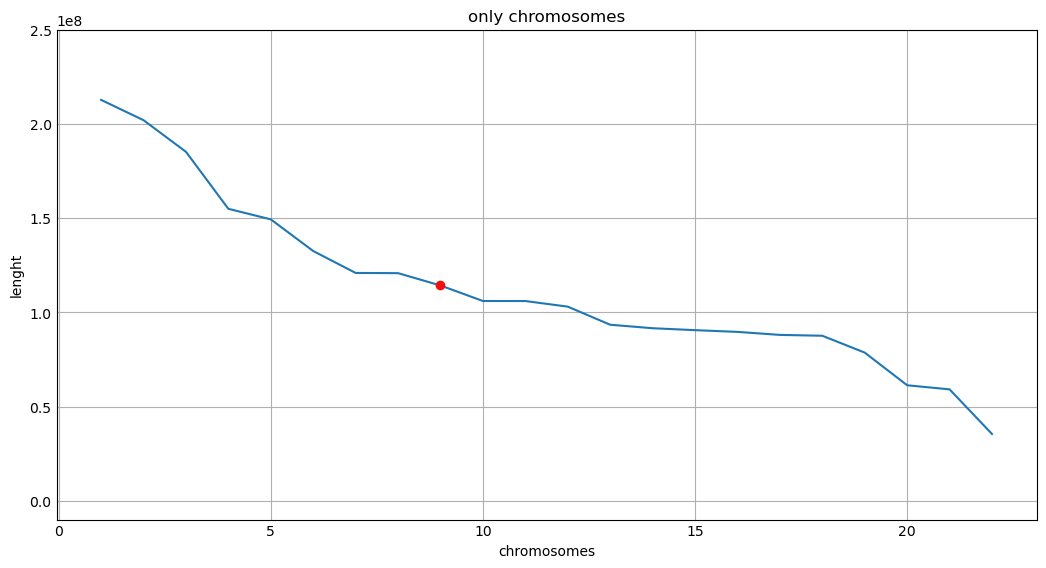
//я отмечал точку, соответствующую L50 и N50 для скэффолдов, а не контигов, так как у меня в файле последовательности именно скэффолдов, а значит она будет нести больше информации
Выводы
Во-первых, я понял, что значения Scaffold L50 и N50 соответствуют 8 хромосоме (Рис. 5, скрипт). Во-вторых, я еще больше удостоверился в том, что сборка очень хорошая, так как содержит целые последовательности всех хромосом, а большинство нелокализированных скэффолдов имеют очень маленькую длину относительно хромосомных последовательностей (Рис. 3, Рис. 4). С другой стороны, несколько нелокализированных скэффолдов имеют довольно большую длину, и мне совершенно непонятно, как скэффолд длиной 10 миллионов пар нуклеотидов может быть нелокализированным.
P. S. Я специально не стал менять ничего в других пунктах после выполнения этого, потому что так более понятен ход моих рассуждений.