Создание репрезентативной выборки гомологов белка PDXS_BACSU
Целью представленной работы является создание репрезентатичной, но не очень большой выборки гомологов PDXS_BACSU по различным таксонам прокариот и эукариот для последующего множественного выравнивания.
Бактерия (Bacillus Subtilis), которой принадлежит изучаемый белок, относится к филуму Firmicutes (об этом свидетельствуют данные NCBI Taxonomy, январь 2013). Был осуществлен поиск BLAST (алгоритм BlastP) с использованием исходной последовательности PDXS_BACSU. При этом на поиск были наложены следующие параметры:
- Ограничение по таксонам. В одном случае был совершен поиск гомологов у прокариотических организмов не из Firmicutes, во втором – поиск гомологов у эукариотических организмов (Eukaryota).
- Порог e-value (до которого выравнивания из выдачи BLAST могут считаться надежными). Был подобран эмпирически при поиске гомологов сначала с большим порогом.
- Максимальное число хитов. Было выбрано значение 5000.
Параметры финального поиска представлены в таблице 1.
Таблица 1. Параметры финального поиска BLAST.
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | BlastP | RefSeq | Не Eukaryota, не Firmicutes | 2е-04 | 5000 |
| По эукариотам | BlastP | RefSeq | Только Eukaryota | 2е-05 | 5000 |
Количество выданных записей:
1) По первому поиску - 818 (прокариотические гомологи)
2) По второму запросу - 149 (эукариотические гомологи)
Для всех последовательностей в обоих случаях была получена выдача GenBank и изображения таксономических деревьев гомологов. Изображения представлены ниже. Деревья содержат названия организмов, которым принадлежат искомые гомологи, и показывает выраженность различий (и, соответственно, сходства) между их последовательностями.
Необходимо сразу отметить, что ни для одного эукариотического гомолога изучаемого белка не была выявления локализация кодирующих генов в митохондриях или хлоропластах. Наиболее вероятным является независимое происхождение белков в различных организмах (про- и эукариотических) для выполнения одинаковых (или сходных) функций за счет определенной близости в их последовательностях и структурах.
Ссылки на выдачу BLAST для обоих запросов можно скачать из данного файла.
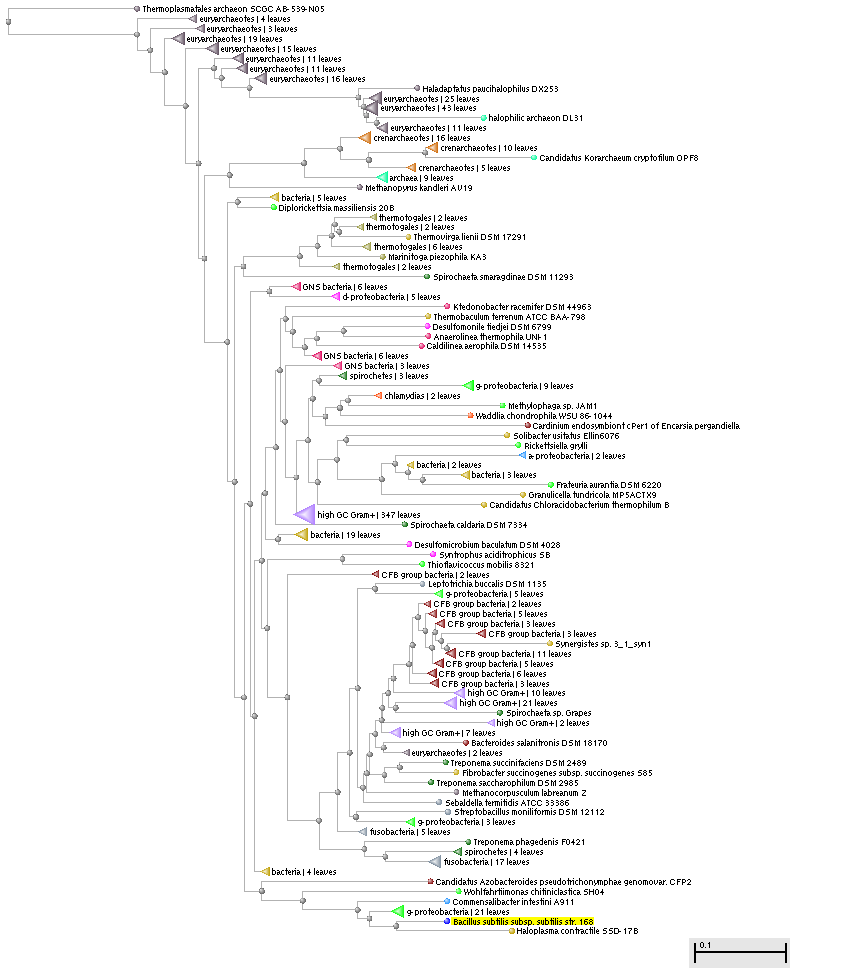
Рис. 1-1. Таксономическое дерево гомологов у прокариотических организмов.
Рис. 1-2. Таблица обозначений некоторых таксонов различными цветами для рис 1-1.
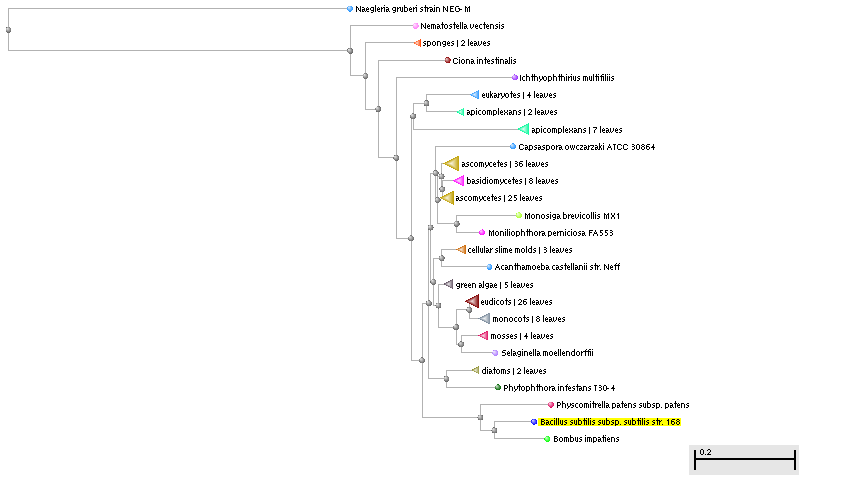
Рис. 2-1. Таксономическое дерево гомологов у эукариотических организмов.
Рис. 2-2. Таблица обозначений некоторых таксонов различными цветами для рис 2-1.
Были отобраны последовательности гомологов организмов, принадлежащих разным таксонам. Названия организмов, их принадлежность к филумам/царствам, количество отобранных последовательностей для разных случаев отражены в таблице 2.
Таблица 2. Встречаемость белков, гомологичных PDXS_BACSU, в разных таксонах. Приведены названия организмов, последовательности гомологов исходного белка которых были взяты для репрезентативной выборки и последующего множественного выравнивания.
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | euryarchaeotes | Natronomonas moolapensis 8.8.11 | 4 |
| (Halobacteriales) | Halococcus thailandensis JCM 13552 | ||
| Halobiforma nitratireducens JCM 10879 | |||
| Haloarcula japonica DSM 6131 | |||
| euryarchaeotes | Acidianus hospitalis W1 | 3 | |
| (Сrenarchaeotes) | Pyrobaculum sp. 1860 | ||
| Caldivirga maquilingensis IC-167 | |||
| euryarchaeotes | Candidatus Nitrosopumilus sp. AR2 | 3 | |
| (Nitrosopumilales) | Nitrosopumilus maritimus SCM1 | ||
| Candidatus Nitrosopumilus salaria BD31 | |||
| Bacteria | firmicutes | Bacillus subtilis strain 168 | 1 (исходный) |
| actinobacteria | Mycobacterium abscessus | 5 | |
| Streptomyces davawensis JCM 4913 | |||
| Rhodococcus ruber BKS 20-38 | |||
| Nocardia brasiliensis ATCC 700358 | |||
| Micromonospora lupini str. Lupac 08 | |||
| proteobacteria | Bibersteinia trehalosi USDA-ARS-USMARC-192 | 5 | |
| Wohlfahrtiimonas chitiniclastica SH04 | |||
| Actinobacillus suis H91-0380 | |||
| Pasteurella multocida 36950 | |||
| Francisella cf. novicida Fx1 | |||
| CFB group bacteria | Prevotella nigrescens F0103 | 4 | |
| (Bacteroidales) | Odoribacter laneus YIT 12061 | ||
| Prevotella oralis ATCC 33269 | |||
| Prevotella buccae ATCC 33574 | |||
| CFB group bacteria | Cardinium endosymbiont cEper1 of Encarsia pergandiella | 1 | |
| (Candidatus Cardinium) | |||
| Eukaryota | Fungi | Ascomycetes: | 2 |
| Aspergillus oryzae RIB40 | |||
| Basidiomycetes: | |||
| Cryptococcus gattii WM276 | |||
| Green plants | Arabidopsis thaliana | 1 | |
| Apicomplexans | Plasmodium chabaudi chabaudi | 1 | |
| Animals | Nematostella vectensis | 2 | |
| Bombus impatiens | |||
Последовательности белков, отобранных для выравнивания представлены в следующих файлах:
"/~pankevich-ev/term2/infoseq/infoseq_help.txt" infoseq/infoseq_help.txt">iМножественное выравнивание гомологов белка PDXS_BACSU
Выравнивание полученных полноразмерных последовательностей (из репрезентативной выборки) было проведено с помощью программы Muscle. Последующая обработка выдачи происходила в программе JalView. Общий вид выравнивания можно увидеть на рисунке 3. Полноразмерное изображение откроется в новом окне при клике на картинку.
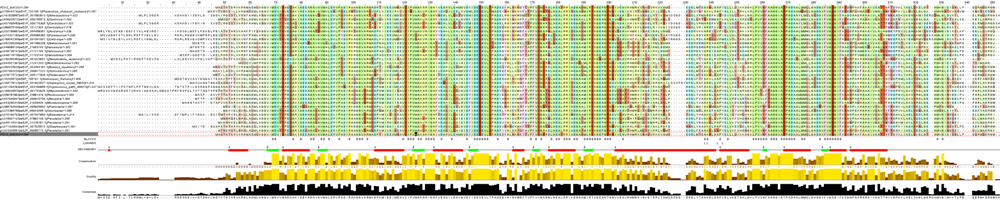
Рис. 3. Выравнивание последовательностей гомологов PDXS_BACSU с исходной последовательностью (для наглядности она приведена в первой и последней строках выравнивания). Полноразмерное изображение можно увидеть кликнув по картинке. Слева от выравнивания - подписи последовательностей (gi-номер, АС, название организма), первые 5 последовательностей (после исходной) - гомологи из эукариот, остальные - из прокариот. Для окраски выравнивания была создана цветовая схема, основанная на свойствах аминокислот (по их радикалам), выбран режим, при котором столбцы, в которых по всей толщине процент идентичности меньше 15% - не окрашиваются (cut-off 15%), яркость окраски столбцов также зависит от количества совпадений. Под выравниванием располагаются следующие строки аннотации:
1. BLOCKS. Показывает блоки выравнивания (B).
2. LIGANDS. Показывает местa связывания лигандов EDO.
3. SECONDARY. Cодержит информацию о известной трехмерной структуре белка PDXS_BACSU (альфа-спирали обозначены красным цветом, бета-листы - зеленым).
4. Conservation. Показывает консервативность каждой позиции выравнивания(по толщине), показывает количество не сходящихся с большинством остатков.
5. Quality. Цветом отражает сходство аминокислот в каждой позиции выравнивания.
6. Consensus. Показывает консервативность каждой позиции выравнивания (по толщине).
Результаты анализа множественного выравнивания гомологов белка PDXS_BACSU
Проведенное выравнивание репрезентативной выборки белков-гомологов PDXS_BACSU дает возможность сделать некоторые выводы. В целом, по общему виду вырванивания можно определить, что последовательность является умеренно консервативной. Далеко не все стоблцы содержат большое колиество идентичных остатков, некоторые белки имеют длинные хвосты в начале, зато не имеют в конце. Это коррелирует с особенностями вторичной стркутры белка. Цепь имеет консувидную форму, причем в ее вершине, как было обнаружено, встречаются более консервативные участки из альфа-спиралей. Там связываются лиганды EDO (этандиол-1,2 или этиленгликоль) при кристаллизации белка. Там же происходит связывание с субъединицами белка (подробнее это описано в работе о внутримолекулярных взаимодействиях в структуре 2NV1 (PDSX_BACSU). Следует отметить, что в целом позиции аминокислот, отвечающих в PDXS_BACSU за образование вторичной структуры, довольно консервативны. Вероятно, гомологи имеют подобную конусовидную форму. Ее функция в образовании третичной и четвертичной структур белка. "Хвосты" в начале последовательности укладываются в основание "конуса", чье значение играет меньшую роль. Соответственно, в начале выравнивание является наихудшим. Также неудачные места выравнивания составляют колонки с гэпами в середине выраванивания. Их наличие обусловлено определенным числом инсерций в геномах организмов-обладателей некоторых гомологов. Интересно, что в первом таком месте "лишние" аминокислотые остатки появляются там, где идет альфа-спираль PDXS_BACSU. Возможно, что эти остатки входят в альфа-спирали вторичной структуры гомологов. Во втором же случае рядом стоящие колонки с гэпами приходятся на линейную часть структуры и, вероятно, не изменяют ее.
В процессе работы были проставлены "блоки", они обозначены буквой "В" в первой строке аннотации. Большинство блоков попадает на позиции аминокислот, которые обусловливают вторичную структуру PDXS_BACSU. Тем не менее, обратного видеть мы не можем: далеко не все позиции, с которыми связана вторичная структура, дают блоки. Это вызвано тем, что блоки выбирались вручную и по весьма жестким критериям. Столбец считался блоком, если идентичность остатков в нем была очень значительной. Возможно, вернее было бы ослабить критерии выбора и уменьшить человеческий фактор при помощи написания специальных скриптов, обозначающих блоками позции с определенной идентичностью остатков по толщине.
Следует оценить консервативность остатков, которые связывают лиганды в белке.
На рисунках 5 и 6 представлены соответственно общий вид расположения лигандов EDO в структуре белка и увеличенный вид с указанием на места связывания лиганда определенными аминокислотами. Отметим, что картинки были получены с помощью интерфейса JMol в программе JalView, которая позволяет с большим удобством определять связь между консервативностью определенных позиций аминокислот и их расположения в пространственной структуре молекулы (этим можно пользоваться при связывании последовательности белка с его структурой PDB, в том числе можно переносить окраску выравнивания на трехмерную структуру).
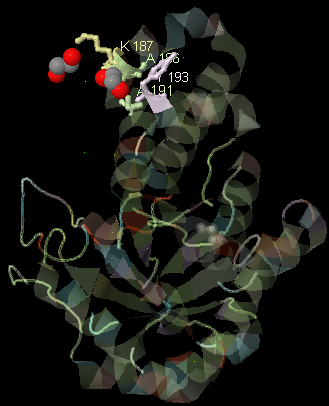
Рис. 5. Общий вид связываения EDO с PDXS_BACSU.

Рис. 6. 2 молкулы EDO и аминокислоты, связывающие эти лиганды.
Подробно о связывании EDO можно прочитать в работе о области контакта белка и лиганда EDO в структуре 2NV1(PDSX_BACSU). Отметим, что для лиганда, связываемого аланином-186 и лизином-187 (в выравнивании - позиции 238, 239), столбцы выравнивания весьма консервативны и могут составлять блоки. Можно заметить, что лизин часто заменяется на аргинин, который в приниципе может выполнять сходные функции. Для второго лиганда, который в PDXS_BACSU связывается аланином-191 и тирозином-187 (в выравнивании позиции 243, 245), столбцы имеют различную степень консервативности. Позиция аланина является слабо консервативной, часто аминокислота заменяется на валин, который (как в случае замены лизина на аргинин) может выполнять ту же функцию, включая в себя неполярный алифатический радикал. Позиция тирозина же имеет слабую консервативность. Этот остаток заменяется на остатки аминокислот иной природы и иными функциями. Можно предположить, что EDO в данном случае вообще не связывается с белком в этом месте. Из этого не может следовать, что белок теряет свою биологическую функцию, т.к. EDO является лигандом, который служит исключительно для кристаллизации данного белка. Если связь у некоторых гомологов не происходит, то можно только предположить, что для этих белков существует несколько иной способ кристаллизации.