Структуры ДНК
Построение моделей структур A-, B- и Z-формы ДНК возможно с помощью инструментов пакета 3DNA. Пакет 3DNA - один из пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот.
В ходе работы были получены А- и В-структуры ДНК с повторяющейся пять в раз последовательностью 4-х нуклеотидов "ctag" в каждой цепи, а также Z-структура ДНК с повторяющимися GC:GC парами. С pdb-файлам можно можно ознакомиться здесь: A-структура, B-структура, Z-структура.
А-структура была выбрана для работы в JMol. Результат работы представлен на рисунке 1.
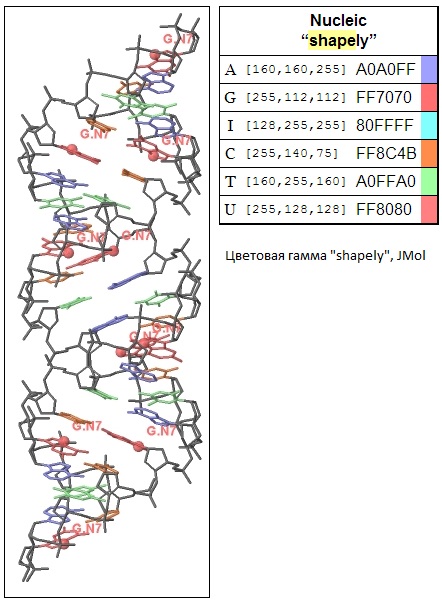
Рис. 1. А-структура ДНК. Сахарофосфатный остов покрашен серым. Выделены различные азотистые основания (отдельное выделение нуклеотидов не является таким информативным), выбрана стандартная цветовая гамма JMol (RasMol) "shapely", обозначения справа от структуры. Крупно выделены и подписаны все атомы N7 у гуанина.
Glutaminyl-tRNA synthetase (1GTR) и Bacteriophage Lambda Excisionase (Xis)-DNA Complex (1RH6)
В данном разделе представлена некоторая информация о структурах РНК и ДНК из pdb-файлов 1GTR и 1RH6 соответственно.
В некоторых структурах PDB есть разрывы, которые могут быть связаны с тем, что структура в самом деле содержит несколько фрагментов или, например, рентгеноструктурный анализ не позволил определить координаты части атомов. Второе для полученных в последнее время структур практически не актуально.
Структуры 1GTR и 1RH6 разрывов не содержат, их проволочные модели представлены на рисунке 2.
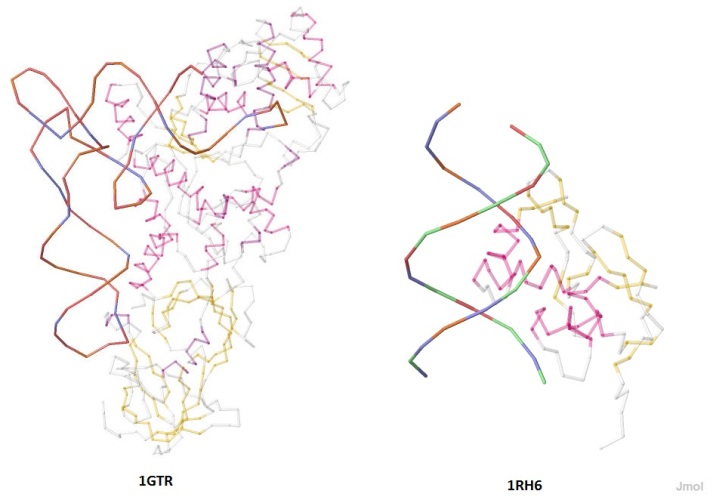
Рис. 2. Проволочные модели РНК (1GTR) и ДНК (1RH6) с окружающими структурами (более прозрачные).
Координаты атомов только РНК и ДНК из pdb-файлов структур 1GTR и 1RH6 можно увидеть в следующих файлах: 1GTR-RNA; 1RH6-DNA.
Сравнение структур 3-х форм ДНК с помощью средств JMol
Расположение тимина в А-, В-, Z-формах ДНК
На рисунке 3 представлена В-структура ДНК из 5 повторяющихся последовательностей нуклеотидов ctag и расположение тимина 6 (T6) в ней.
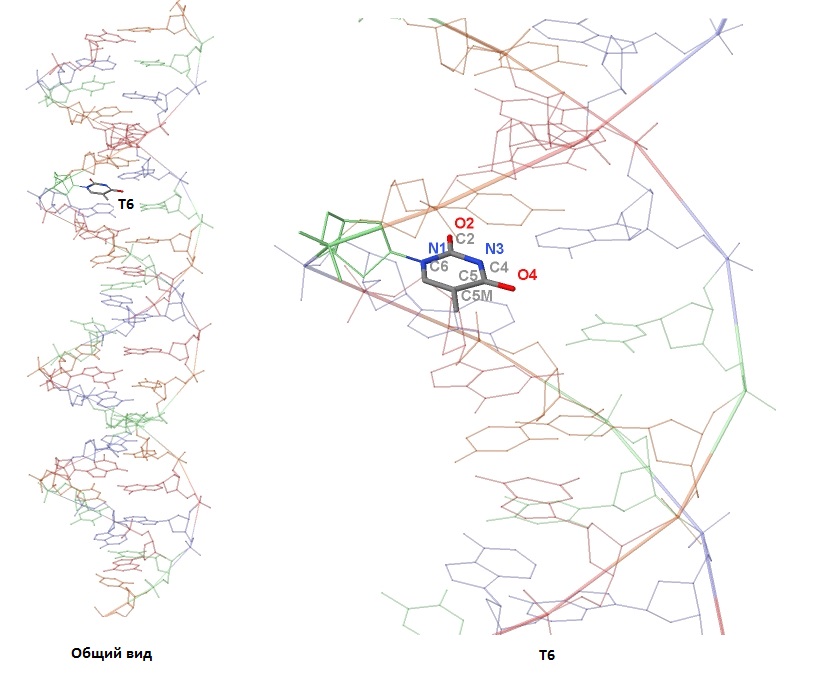
Рис. 3. Тимин в структуре В-формы ДНК. Сверху - большая бороздка, снизу - малая бороздка. Можно видеть, какие атомы обращены к разным бороздкам. Пояснения в тексте.
В сторону большой бороздки расположены атомы: С6, С5, С4, О4, С5M.
В сторону малой бороздки расположены атомы: О2, С2, N3.
Атом N1 находится практически в плоскости между бороздками, но имеет небольшой сдвиг в сторону малой бороздки.
Для А-структуры ДНК расположение атомов тимина аналогично.
В Z-структуре ДНК нет нуклеотидов с азотистым основанием тимином, в образовании этой структуры участвуют цитозин и гуанин. Помимо этого, многие источники утверждают, что Z-структура имеет только малые бороздки. В самом деле, те, бороздки, которых часто называют большими, не достаточно глубокие и визуально похожи на малые.
На рисунке 4 представлена структура тимина с выделенными обращенными к разным бороздкам атомами.
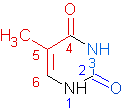
Рис. 4. Тимин. Атомы, обращенные к большой бороздки выделены красным, обращенные к малой - синим.
Характеристики различных структур ДНК
В таблице 1 представлены основные характеристики А-, В- и Z-структур ДНК.
Таблица 1. Характеристики структур ДНК.
| A-форма | B-форма | *Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (nm) | 2.803 | 3.375 | 4.35 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (nm, от гуанина) | 1.697 | 1.721 | 0.720 |
| Ширина малой бороздки (nm, от гуанина) | 0.798 | 1.169 | 0.987 |
Примечание по поводу ширины бороздок ДНК. Ширина бороздки определена для конкретного нуклеотида в достаточно регулярной структуре. Это расстояние от атома фосфора данного нуклеотида до некоторого фосфора комплементарной цепи - такого, что расстояние до соседних фосфоров несколько больше. В нашем случае ширина измерялась от гуанина.
Отметим также, что ширина большой бороздки не всегда больше ширины малой (бороздки оцениваются по глубине). Для Z-формы ширина большой бороздки (если считать, что она вообще есть в этой структуре) меньше, чем у малой.
На рисунке 5 показаны измерения параметров для А-структуры ДНК.
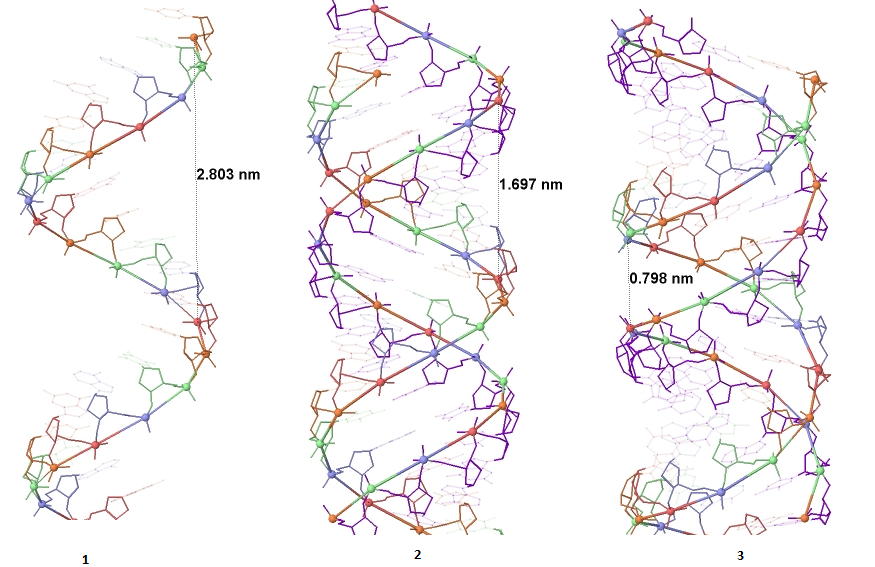
Рис. 5. Длина витка (1), ширина большой (2) и малой (3) бороздок А-структуры ДНК.
Измерение торсионных углов для тимина 6 в А- и В-структурах ДНК
Были измерены торсионные углы при тимине 6 в А- и В-структурах ДНК из 5 повторяющихся последовательностей нуклеотидов ctag. Данные приведены в таблице 2. Также в таблице указаны аналогичные данные из другого источника (см. презентацию). Изображения измеренных торсионных углов представлены на рисунке 6.
Таблица 2. Торсионные углы при тимине 6.
| α (P-O5') | β (O5'-C5') | γ (C5'-C4') | δ (C4'-C3') | ε (C3'-O3') | ζ (O3'-P) | χ (C1'-N) | |
| А-ДНК | -51.7 | 174.8 | 41.7 | 79.0 | 147.8 | -75.1 | -157.2 |
| А-ДНК (презентация) | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| В-ДНК | -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98 |
| В-ДНК (презентация) | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
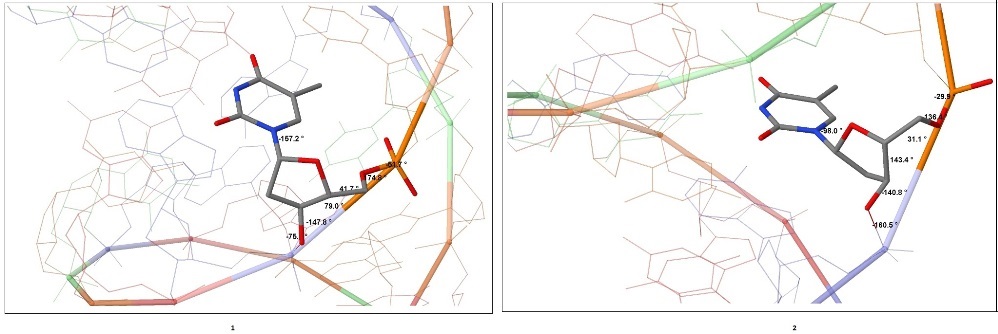
Рис. 6. Торсионные углы тимина 6 в А-структуре (1) и В-структуре (2) ДНК.
Для дальнейшей работы с помощью команд биоинформатического пакета 3DNA была получена различная информация, в том числе значения торсионных углов в А-, В- и Z-структурах ДНК в файлах ctag-a-old.out, ctag-b-old.out и ctag-z-old.out соответственно.
В Z-структуре не встречаются тимины. Для А- и В-структур было проведено сравнение подсчитанных вручную (в JMol) торсионных углов при тимине 6 и их значений из программного файла (табл. 3). Все значения совпали, потому что при измерении вручную выбирались четверки атомов по правилу (при наличии разных вариантов - по старшенству по правилам IUPAC) (рис. 5 и табл. 2).
Таблица 3. Значения торсионных углов при тимине 6 в А- и В-структурах ДНК.
| α (P-O5') | β (O5'-C5') | γ (C5'-C4') | δ (C4'-C3') | ε (C3'-O3') | ζ (O3'-P) | χ (C1'-N) | |
| А-ДНК(out) | -51.7 | 174.8 | 41.7 | 79.0 | -147.8 | -75.1 | -157.2 |
| В-ДНК(out) | -29.9 | 136.4 | 31.1 | 143.4 | 140.8 | -160.5 | -98 |
Работа со структурой ДНК из 1RH6
С помощью команд пакета 3DNA была получена информация о торсионных углах ДНК из структуры 1RH6. Увидеть эти данные можно в таблицах ниже или в файле 1RH6_DNA_torsion.xlsx.
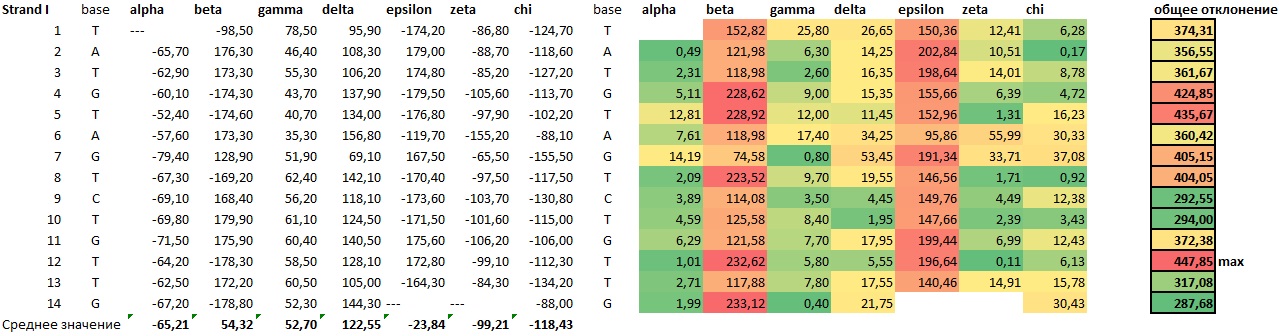
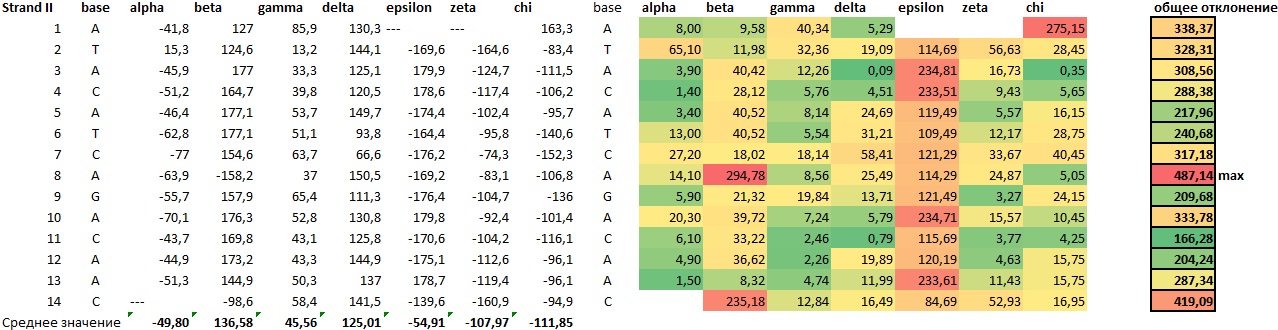
Видно, что в цепи I наиболее деформированными основаниями (с наиболее отклоняющимся средним значением какого-либо из торсионных углов или сразу нескольких углов) можно назвать T12, T5, G4, а в цепи II - 8A, 14C. Отметим, что при подсчете среднего значения торсионных углов нуклеотидов не учитывались углы для краевых оснований.
Работа со структурой РНК из 1GTR
При помощи уже изученной программы find_pair из пакета 3DNA, мы определили возможные связи между азотистыми основаниями в структуре тРНК из 1GTR. Информация представлена в файле 1GTR-old.out.
Стебли тРНК представлены спаренными основаниями (участками одной цепи и комплементарных им участками другой цепи; см. рис. 7):
- акцеторный стебель: участок 2-7 и комплементарный 66-71 (выделен розовым цветом);
- T-стебель: участок 49-53 и комплементарный 61-65 (выделен оранжевым цветом);
- D-стебель: участок 39-43 и комплементарный 31-27 (выделен голубым цветом);
- антикодоновый стебель: 10-12 участок и комплементарный: 25-23 (выделен зеленым цветом).
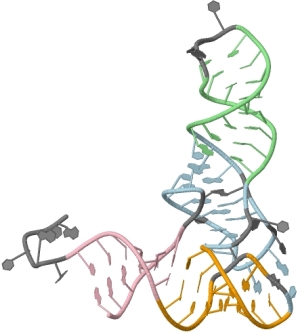
Рис. 7. Структура тРНК из 1GTR: показаны стебли (пояснения в тексте).
Водородные связи образуются в структуре и между неканоническими парами (отмечены * в файле). Таких пар 8:
12 (0.011) B:..54_:[..U]U-**-xA[..A]:..58_:B (0.010) | 13 (0.017) B:..55_:[..U]Ux**+xG[..G]:..18_:B (0.009) x 14 (0.010) B:..37_:[..A]A-*---U[..U]:..33_:B (0.014) | 15 (0.016) B:..38_:[..U]U-*---U[..U]:..32_:B (0.016) | 21 (0.015) B:..44_:[..C]Cx*---A[..A]:..26_:B (0.012) | 25 (0.012) B:..13_:[..A]A-**+xA[..A]:..45_:B (0.012) | 26 (0.011) B:..14_:[..A]A-*--xA[..A]:..21_:B (0.014) | 27 (0.008) B:..15_:[..G]Gx**+xC[..C]:..48_:B (0.009) x
Также были обнаружены дополнительные водородные связи, не имеющие отношение к стеблям и стабилизирующие третичную структуру тРНК. Это каноническая пара (можно сказать, она создает стебель длиной в одну пару нуклеотидов):
28 (0.009) B:..19_:[..G]G-----C[..C]:..56_:B (0.011) +
Исследование третичной структуры
В файле 1GTR-old.out имеется информация о перекрывании (overlap) последовательных пар азотистых оснований и о площади этих "перекрываний". Она представлена ниже.
Values in parentheses measure the overlap of base ring atoms only.
Those outside parentheses include exocyclic atoms on the ring.
Intra- and inter-strand overlap is designated according to the following diagram:
i2 3' 5' j2
/|\ |
| |
Strand I | | II
| |
| |
| \|/
i1 5' 3' j1
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 4.05( 2.85) 0.00( 0.00) 0.09( 0.00) 0.63( 0.00) 4.76( 2.85)
2 GG/CC 3.65( 2.31) 0.00( 0.00) 0.35( 0.00) 0.06( 0.00) 4.06( 2.31)
3 GG/CC 3.96( 2.48) 0.00( 0.00) 0.52( 0.00) 0.00( 0.00) 4.49( 2.48)
4 GU/AC 6.66( 3.82) 0.00( 0.00) 0.00( 0.00) 4.45( 2.93) 11.11( 6.75)
5 UA/UA 0.62( 0.00) 0.00( 0.00) 1.16( 0.94) 0.13( 0.00) 1.91( 0.94)
6 AC/GU 2.76( 1.60) 0.00( 0.00) 0.00( 0.00) 6.27( 3.25) 9.03( 4.85)
7 CG/CG 0.00( 0.00) 0.00( 0.00) 5.60( 2.70) 0.00( 0.00) 5.60( 2.70)
8 GA/UC 4.18( 2.38) 0.00( 0.00) 0.00( 0.00) 2.65( 0.35) 6.82( 2.73)
9 AG/CU 3.53( 3.27) 0.00( 0.00) 0.23( 0.00) 0.33( 0.00) 4.09( 3.27)
10 GG/CC 3.60( 2.09) 0.00( 0.00) 0.92( 0.00) 0.00( 0.00) 4.52( 2.09)
11 GU/AC 6.60( 3.77) 0.00( 0.00) 0.00( 0.00) 4.43( 1.73) 11.04( 5.50)
12 UU/GA 4.81( 2.33) 0.00( 0.00) 0.00( 0.00) 6.06( 3.01) 10.87( 5.35)
13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
14 AU/UU 4.84( 1.19) 0.00( 0.00) 0.00( 0.00) 3.97( 1.95) 8.80( 3.14)
15 UU/AU 2.33( 0.94) 0.00( 0.00) 0.00( 0.00) 4.93( 3.75) 7.25( 4.68)
16 UC/GA 0.11( 0.01) 0.00( 0.00) 0.00( 0.00) 2.57( 1.07) 2.68( 1.07)
17 CC/GG 0.65( 0.02) 0.00( 0.00) 0.30( 0.00) 3.18( 1.73) 4.12( 1.75)
18 CG/CG 0.00( 0.00) 0.00( 0.00) 5.88( 2.66) 0.00( 0.00) 5.88( 2.66)
19 GG/CC 3.83( 2.44) 0.00( 0.00) 0.57( 0.00) 0.07( 0.00) 4.47( 2.44)
20 GC/AC 6.92( 3.76) 0.00( 0.00) 0.00( 0.00) 5.28( 3.35) 12.21( 7.11)
21 CG/CA 0.00( 0.00) 0.00( 0.00) 0.52( 0.00) 0.69( 0.21) 1.21( 0.21)
22 GC/GC 4.19( 1.51) 0.00( 0.00) 0.00( 0.00) 6.41( 3.26) 10.61( 4.77)
23 CC/GG 0.05( 0.00) 0.00( 0.00) 0.52( 0.00) 3.10( 1.58) 3.66( 1.58)
24 CA/AG 1.89( 0.97) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 1.89( 0.97)
25 AA/AA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 5.05( 4.27) 5.05( 4.27)
26 AG/CA 4.22( 1.87) 0.00( 0.00) 0.00( 0.00) 5.49( 2.42) 9.71( 4.29)
27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Для пары с наибольшим значением (20 GC/AC) было получено стандартное изображение стекинг-взаимодействия, оно представлено на рисунке 8. Стекинг-взаимодействия возможны и для пары с несколько меньшей площадью перекрывания.
20 GC/AC 6.92( 3.76) 0.00( 0.00) 0.00( 0.00) 5.28( 3.35) 12.21( 7.11)
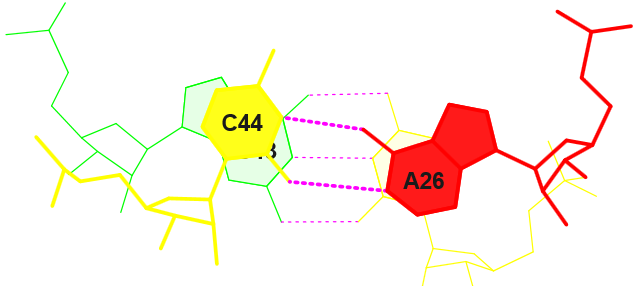
Рис. 8. Изображение стекинг-взаимодействий для пары 20 GC/AC РНК из 1GTR.
Данные о стекинг-взаимодействиях найдены в файле stacking.pdb, необходимая стркутура 20 GC/AC (Section #0020) была вырезана в отдельный файл step20.pdb, с помощью которого было получено изображение step20.ps, переведенное в формат jpg. Использованные команды:
Нормальный online-конвертер так и называется: online-convert.com.