Киотская энциклопедия генов и геномов (KEGG)
Метаболические пути с GALK2
В Swiss-Prot была найдена ссылка на киотсткую энциклопедию генов и геномов KEGG: hsa:2585. Данные о метаболических путях фермента GALK2 человека, приведенные в KEGG, указаны в таблице 1.
Таблица 1. Метаболические пути GALK2 человека из KEGG.
| KEGG ID | Англоязычное название | Русскоязычное название | |
| 1 | hsa00052 | Galactose metabolism | Метаболизм галактозы |
| 2 | hsa00520 | Amino sugar and nucleotide sugar metabolism | Метаболизм аминосахаров и фосфорных эфиров нуклеозидов |
| 3 | hsa01100 | Metabolic pathways | Метаболические пути (общая карта для человека) |
Можно посмотреть карту метаболического пути галактозы.
Cтруктурные формулы соединений в KEGG
С помощью сервиса KEGG LIGAND была выяснена информация о двух выбранных веществах.
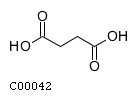
1. Сукцинат (янтарная кислота)
- Английское название: Succinate (Succinic acid)
- Идентификатор: C00042
- Структурная формула:
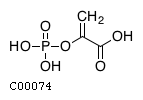
2. Фосфоенолпируват
- Английское название: Phosphoenolpyruvic acid
- Идентификатор: C00074
- Структурная формула:
Метаболический путь между двумя веществами
С помощью сервиса KEGG PATHWAYS были найдены карты метаболических путей, в которых участвуют сукцинат и фосфоенолпируват. Один из таких путей был выбран для изучение.
Выбранная цепочка ферментативных реакций:
Карта: метаболизм углерода (carbon metabolism, идентификатор: ko01200)
Путь: фосфоенолпируват > сукцинат
Кратчайший путь включает в себя 4 интермедиата (C00022, C00036, C00149, C00122). Их названия можно увидеть на рисунке 1. Этот путь имеет направление от фосфоенолпирувата к сукцинату, но только реакция между первым и вторым интермедиатом идет необратимо: превращение пирувата в оксалоацетат.
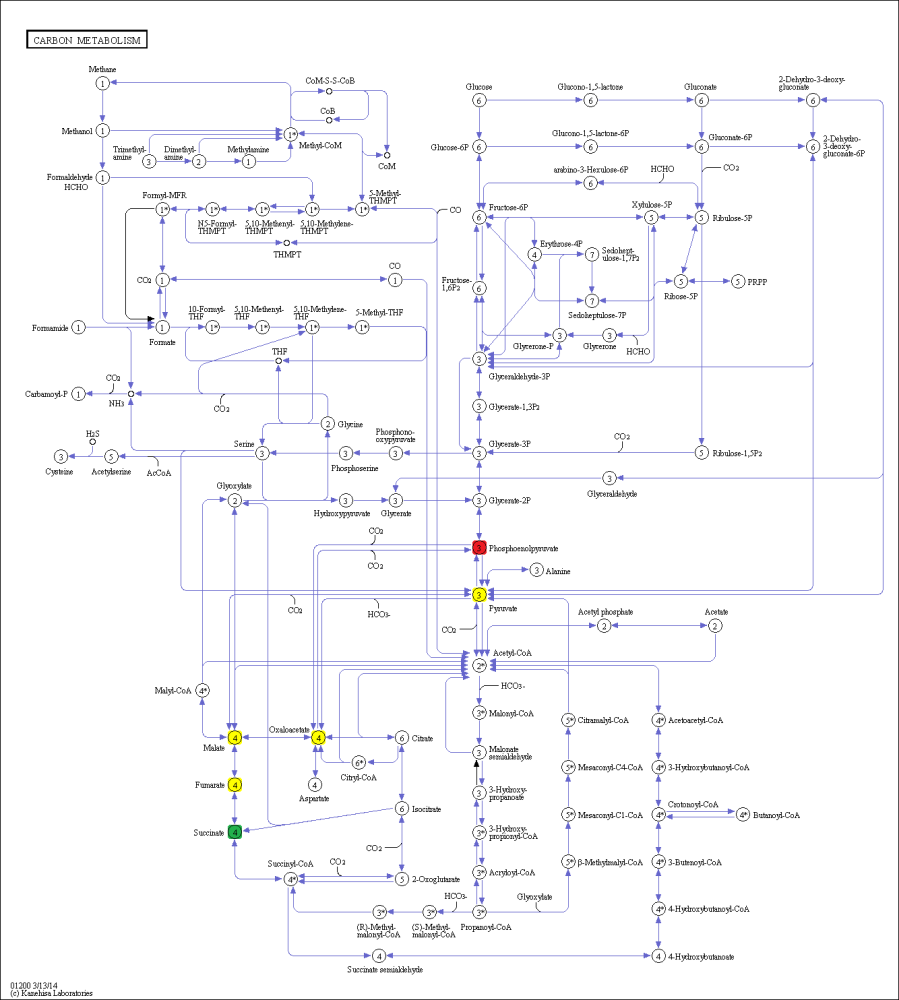
Рис. 1. Карта метаболизма углерода. Начальное соединение (фосфоенолпируват) отмечен красным, конечное соединение (сукцинат) - зеленым. Интермедиаты отмечены желтым цветом.
База данных KEGG - необходимые инструменты
KEGG предлагает также множество сервисов, которые пока что мы не использовали. Тем не менее, они часто могут быть полезными для решения задач, возникающих в работе молекулярных биологов.
Кратко о возможностях и сервисах базы можно прочитать в разделе KEGG Mapper. Здесь же автору данной работы хотелось бы рассказать о том, что база данных KEGG интегрируется в различные биоиформатические ресурсы. Одним из таких является DAVID Bioinformatics Resources, очень полезного для работы с большими листами генов. DAVID выполняет функциональную классификацию групп генов, например, по ключевым словам (и по многим другим критериям). Для каждой группы генов производится поиск по базе KEGG, после чего выдается сигнальный каскад, в котором участвуют все (или наибольшая часть) генов, интересующих исследователя. Это оказывается важным для первичного понимания возможных связей в сигнальных путях клетки.
Описанный процесс опирается на сервис Reconstruct Pathway базы данных KEGG. Так, будучи интегрированной в биоинформатические программы, KEGG может добавлять удобный и доступный функционал. Но также отдельные сервисы могут быть использованы для описанных целей самостоятельно с сайта KEGG.
Также необходимо отметить высокую скорость обработки данных в KEGG. Это одно из главных преимуществ данной системы. Также она обладает проработанным веб-интерфейсом, благодаря чему практически не приходится искать то, что необходимо для решения той или иной задачи.