Алгоритмы реконструкции деревьев. Укоренение и бутстреп.
Укоренение в среднюю точку
Укоренение в среднюю точку удобно и важно делать для деревьев, которые не являются ультраметрическими, т.е. ветви имеют длины, а корней нет. Темпы эволюции всех ветвей дерева предполагают равными. При укоренении таким методом корень помащается в середине самого длинного "маршрута".
С использованием программы retree пакета PHYLIP, было укоренено в среднюю точку дерево, построенное методом Neighbor-joining tree using PID (% Identity) ("Реконструкция филогенетических деревьев"). Полученное с помощью программы MEGA6 изображение представлено на рисунке 1. Файл в формате nwk с результатом: out.nwk.
На рисунке 2 приведено правильное дерево (см. "Филогенетические деревья").
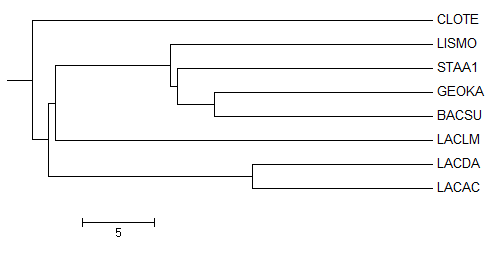
Рис. 1. Укоренение дерева в среднюю точку.
Рис. 2. Правильное дерево.
Использование внешней группы
Деревья, построенные методом максимальной экономии ("Maximum parsimony") невозможно укоренить в среднюю точку, из-за того, что метод не реконструирует длины ветвей. Метод направлен на анализ данных о последовательностях и определение оптимального дерева с минимизированным количеством мутаций. Однако можно воспользоваться укоренением с помощью внешней группы.
На данном этапе работы была произведена рекострукция укорененного дерева отобранных бактерий с использованием в качестве внешней группы белок шаперонного семейства (HSLO) из кишечной палочки (Escherichia coli, ECOLI).
Все отобранные бактерии принадлежат типу Firmicutes (грамположительные), а ECOLI относится к Proteobacteria (грамотрицательные).
Систематическое положение ECOLI: Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia.
Проведено множественное выравнивание MUSCLE последовательностей шапероновых белков 9 бактерий (8 отобранных и ECOLI). Результат можно увидеть в приложенных файлах в aln-формате и в fasta-формате.
По построенному выравниванию с помощью MEGA6 методом maximum parsimony реконструировано дерево. ECOLI использована в качестве внешней группы (Subtree, Root, выбор ветви). На рисунке 3 приведено полученное дерево без ECOLI (Show subtree separately).
Рис. 3. Реконструкция: maximum parsimony, ECOLI использована в качестве внешней группы.
Наблюдения. Все листья на одном расстоянии от корня, т.к. не учитываются расстояния. Полученное дерево очень сильно отличается от правильного. С последним всего одна общая ветвь: {LACDA,LACAC}, остальные ветви не совпадают. Укоренение пришлось в неправильное место. Все это является следствием того, что при использовании выбранного метода учитывается лишь относительное число мутаций. Реконструкция только по одному семейству белков не показательна.
Bootstrap analysis
Был проведен бутстрэп-анализ филогении белков. Дерево было реконструировано в MEGA6 с использованием метода Neighbour Joining (меню: Test of Phylogeny, Bootstrap method). Было взято число реплик: 100. Каждая бутстрэп-реплика получается в результате случайного удаления половины столбцов из выравнивания с заменой их копиями других (тоже случайно выбранных) столбцов.
Получено два дерева: "Original tree" и "Bootstrap consensus tree", они приведены на рисунке 4.
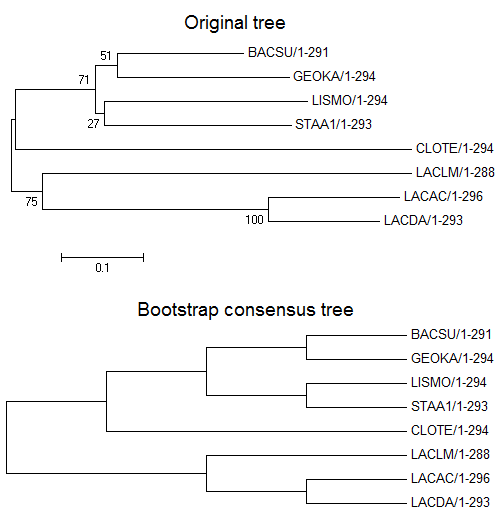
Рис. 4. Деревья: Neighbour Joining "Original tree" и "Bootstrap consensus tree"
Видно, что деревья, "Original tree" и "Bootstrap consensus tree" не отличаются.
Это говорит о том, что построение по части данных (100 реплик по случайной половине имеющихся столбцов выравнивания) в принципе такое же по достоверности, как и по полноценным данным. Тем не менее, дерево не очень похоже на правильное; это обусловлено выбором исходного метода построения дерева (мы уже видели, что он давал не совсем достоверный результат).
Правильные ветви имеют высокую поддержку. Чем число ближе к 100, тем правильнее ветвь, так, ветвь {LACAC, LACLM} против {LACDA, CLOTE, BACSU, GEOKA, LISMO, STAA1} с числом 100 воспроизводилась всеми испробованными на занятии 2 методами. Неправильные ветви имеют меньшую поддержку.