Анализ трехмерных структур
Определение вторичной структуры
Белок 3tui (3tui.pdb) - мембранный белок E.coli, который уже рассматривался в предыдущих работах, в частности в работе по изучению особенностей мембранных белов (ссылка на работу). В данном разделе рассматривается предсказание его вторичной структуры по структуре с помощью метода DSSP (запуск на kodomo с помощью команды: mkdssp -i 1bl8.pdb -o 1bl8.dssp).
Была получена выдача DSSP для белка 3tui. Было проанализировано соответствие полученного предсказания аннотации PDB на примере 4-х альфа-спиралей цепи А и 2-х бета-тяжей в цепи G. Данные представлены в таблице 1.
Предсказание для бета-тяжей полностью совпало с аннотацией. Краевые остатки спиралей в нескольких случаях предсказаны как изгибы (маркировка S в выдаче DSSP), однако в остальном предсказание альфа-спиралей правильное.
Таблица 1. Предсказанные DSSP и проаннотированные в PDB элементы вторичной структуры 3tui.
| 1 | α | 2 | α | 3 | α | 4 | α | 1 | β | 2 | β | |
| Н | К | Н | К | Н | К | Н | К | Н | К | Н | К | |
| PDB | 2 | 41 | 49 | 65 | 67 | 75 | 75 | 84 | 161 | 165 | 193 | 198 |
| DSSP | 3 | 40 | 50 | 64 | 68 | 74 | 76 | 83 | 161 | 165 | 193 | 198 |
С помощью сервиса SheeP были построены карты бета-листов белка 3tui. В качестве "&beta-sheet detector" был выбран "Original Stride". Карта одного из листов (лист 7, в таблице 1 - два бета-тяжа из этого листа) приведена на рисунке один. На составленной карте, приведенной на рисунке 8, красным выделены остатки, боковые цепи которых "смотрят" наружу баррели, жёлтым – внутрь. Нерегулярности (buldges) присутствуют в листе: Thr198 и Glu221 в четвертом столбце в карте не связаны - это вставки, которые не связаны водородными связями в листе.
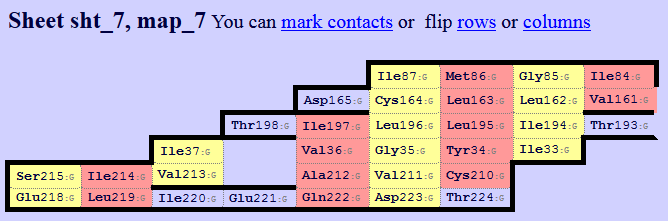
Рис. 1. Карта листа 7, выдача сервиса SheeP (пояснения в тексте).
На рисунке 2 показан лист, где остатки, сморящие внутрь, выделены зеленым, а смотрящие вперед - красным (неопределившиеся остатки - желтого цвета). Показаны также водородные связи.
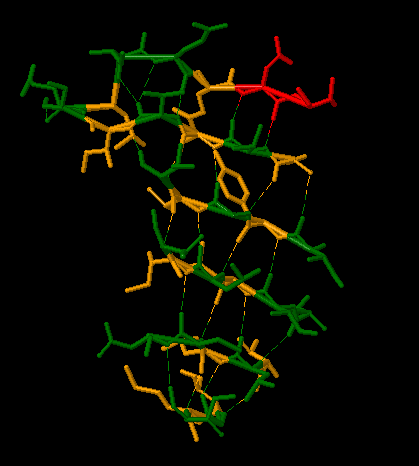
Рис. 2. Изображение листа 7, определенного SheeP.
Соответствие одного столбца на карте и хребта в бета-листе
На рис. 2-2 цветом показан один "хребет" бета-листа, показанный на карте (остатки 86, 163, 195, 34, 210). Как видно, эти остатки действительно расположены в одном хребте.
Рис. 2-2. Бета-лист структуры 3tui, найденный SheeP. "Хребет" бета-листа, показанный на карте SheeP (остатки 86, 163, 195, 34, 210) покрашен красным.
Совмещение структур
С помощью PDBeFold (с параметрами по умолчанию) были найдены гомологи белка 3tui, а затем выбраны и совмещены 5 структур. Автоматически были выбраны только некоторые (по одной) цепи белков. Последовательности белков можно найти в fasta-файле по ссылке.
Последовательности были выровнены с помощью Muscle, оно представлено в файле, а выравнивание по структуре можно найти во втором файле, само совмещение представлено на рисунке 3.

Рис. 3. Совмещение структуры 3tui и гомологов (с помощью PDBeFold).
Можно обнаружить некоторые несоответствия множественного выравнивания по последвательности и по структуре. 44 остаток и 101-102 остатки белка 2onk (D) выровнены по структуре, но не выровнен по последовательности. Таких примеров можно обнаружить достаточно много.
Вывод: изначально не очевидно, какое из выравниваний более правильное - по структуре или по последовательности, каждый случай необходимо разбирать отдельно, однако, биологическая интуиция подсказывает, что чаще выравнивание по структуре будет более правильным (исследуемый случай - не исключение). В целом, выравнивания по структуре - золотой стандарт для выравнивания по последовательности.
Поиск по сходству структур в PDBeFold
Был осуществлен поиск структурных гомологов для домена A:203-315 белка 1b0m со следующими параметрами: поиск производился по всему архиву PDB, отменено: "если хитов нет, то показать то, что больше всего похоже". Результатов не было обнаружено. Было предположено, что это связано с порогом сходства (70% по умолчанию). Далее этот параметр был изменен на 30%, и были получены результаты. Они были отсортированы по RMSD. Первым хитом (кроме исходного белка с RMSD = 0) был белок (его часть) 1nit:A с RMSD = 0.27.
Совмещение по заданному выравниванию
В данном разделе показано совмещение по заданному выравниванию структур константного домена T-клеточного рецептора из цепочки альфа 1j8h:d:118-203 и из цепочки бета 2bnu:b:114-242 (домен и границы из SCOP).
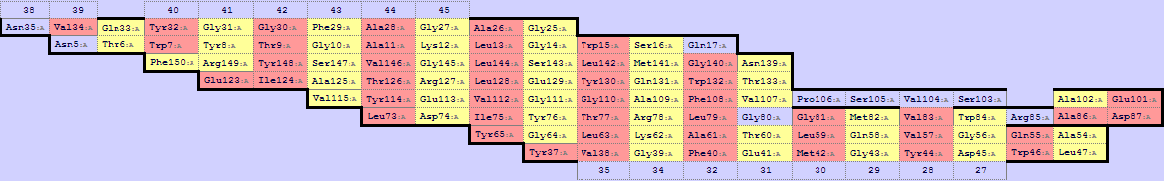
Для выбранных структур были построены карты бета-листов с помощью SheeP (рис.1-2). Для альфа-цепи найден один лист (рис.1), для бета-цепи - два листа (рис.2). Из карт бета-листов трудно сделать заключение о том, насколько листы из разных структур похожи. Чтобы понять, какой лист в цепочке бета соответствует листу в цепочке альфа, необходимо дальнейшее рассмотрение листов. Можно предположить, что первая карта (карта 0) для бета-цепи (12 хребтов) соответствует карте из альфа-цепи (14 хребтов), исходя из количества хребтов.

Рис. 1. Лист альфа-цепочки, найденный Sheep (пояснения в тексте).
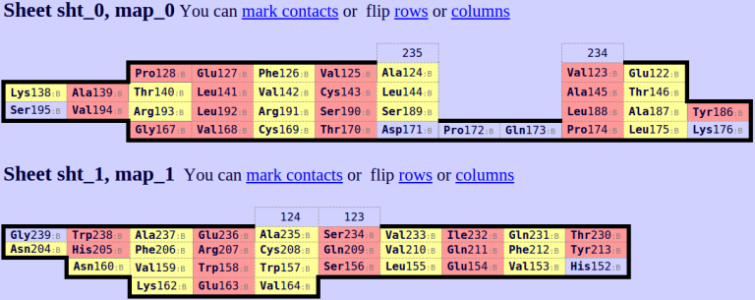
Рис. 2. Листы бета-цепочки, найденные Sheep (пояснения в тексте).
По всей видимости, предположение верно. Дополнительные перестановки в листе альфа-цепочки и первом листе бета-цепочки позволяют добиться того, чтобы указанные листы были в одной ориентации, то есть они соответствуют друг другу. Кроме того, можно обнаружить консервативный остаток цистеина (известно, что такой должен быть и должен образовывать дисульфидную связь). В альфа-цепочке это Cys139 (5-ая колонка), в бета-цепочке - Cys143 (6-ая колонка).
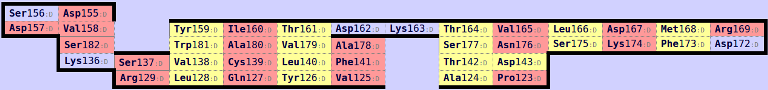
Рис. 3. Лист альфа-цепочки после дополнительных перестановок.
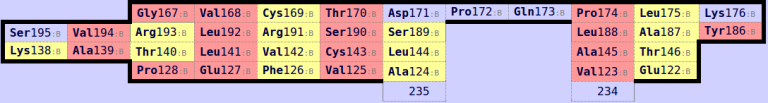
Рис. 4. Лист (карта 0) бета-цепочки после дополнительных перестановок.
Далее на основе листов было построено совмещение с помощью pymol - по нескольким хребтам. Использовались остатки, спаренные с цистеином (вероятно, цистеины должны быть выровнены), а также тяжи (их остатки), в которые эти остатки входят (до появления всяких нерегулярностей: в листе, в направлении боковых радикалов). Выравнивание представлено на рисунке 5. Команды в pymol для построения выравнивания с номерами использованных для выравнивания остатков:
select alpha_align, (resi 137-140 or resi 159-161 or resi 179-181 or resi 126-129) and 1J8H_mod and name CA
select beta_align, (resi 141-144 or resi 189-191 or resi 169-171 or resi 124-127) and 2BNU_mod and name CA
pair_fit alpha_align, beta_align
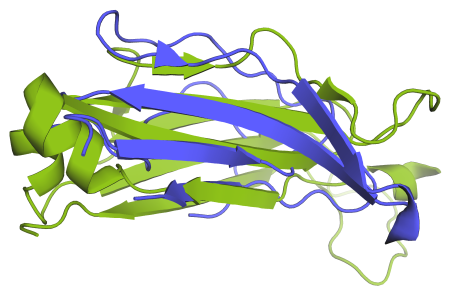
Рис. 5. Совмещение.
Как видно, выравнивание по бета-тяжам помогает получить хорошее совмещение структур. В двух местах в совмещении (на рис.5) видно, что в одной структре - неупорядоченные петли, а в другой - вторичные структуры - альфа-спирали, но ход цепи по тяжам сохраняется для обеих структур, что означает сходство топологий.
Нахождение гидрофобных кластеров
В данном разделе работы был проведен анализ структуры белка 2jmm (который уже рассматривался ранее, в разделе 4 семестра). Для этого был использован сервис SheeP На составленной карте, приведенной на рисунке 8, красным выделены остатки, боковые цепи которых "смотрят" наружу баррели, жёлтым – внутрь.
С помощью сервиса Clud были найдены гидрофобные кластеры белка. При параметрах по умолчанию находятся 22 кластера, 7 из которых составляют более 11 остатков и 2 из которых составляют 42 остатка. Кластеры из более, чем 11 остатков, визуализированы на рисуке 5.
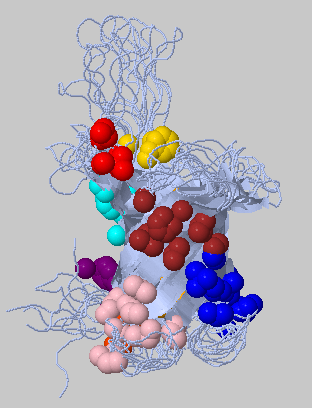
Рис. 5. Гидрофобные кластеры 2jmm (более 11 остатков, выделены ярко), найденные с помощью Clud.
На риснуке 6 представлены: локализация белка в мембране (из OPM); изображение 3D структуры, на котором смотрящие наружу боковые цепи указаны красным цветом, а также приведена структура, в которой коричневым отмечены полярные остатки. Видно, что они в не входят в трансмембранную часть белка. Они либо близко расположены к поворотам, либо находятся в цитоплазматической части белка, либо не являются обращенными в мембрану. В это время обращенные в мембрану аминокислотные остатки гидрофобны.
Гидрофобные кластеры, найденные с помощью Clud, как видно, обращены в мембарну.
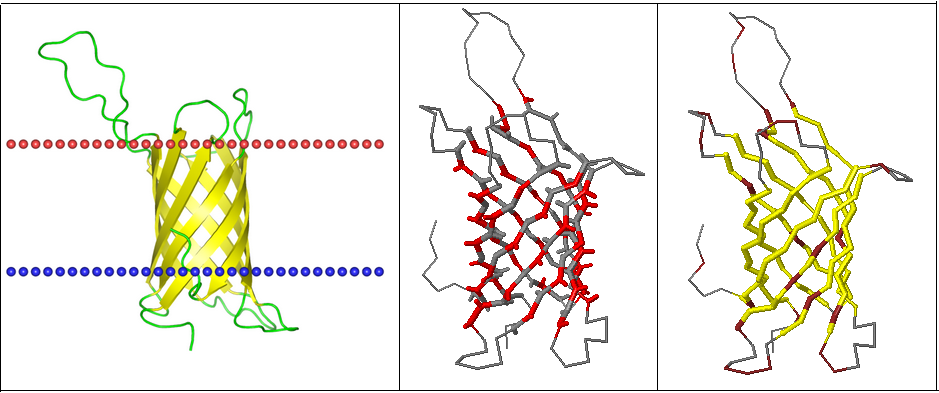
Рис. 6. 3D структуры белка 2jmm. Пояснения в тексте.
Построение поверхности, раскраска участка поверхности: PyMol
Для комплекса димера пуринового репрессора 1QP7 с ДНК в PyMol были созданы изображения:
- поверхности контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера (рис. 2);
- поверхности контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт (рис. 3);
- поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали (рис.4).
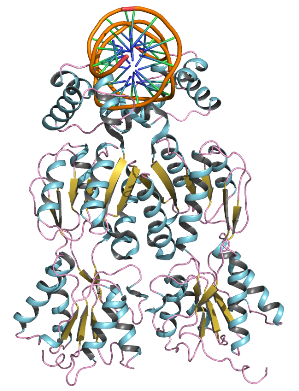
Рис. 1. Изображение димера белка в комплексе с ДНК.
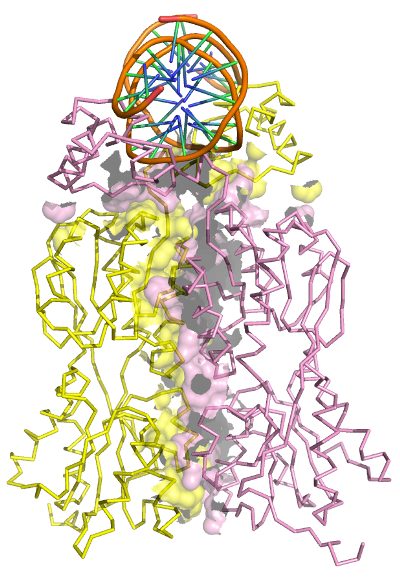
Рис. 2. Изображение поверхности контакта (менее 5 ангстрем) между мономерами белка.
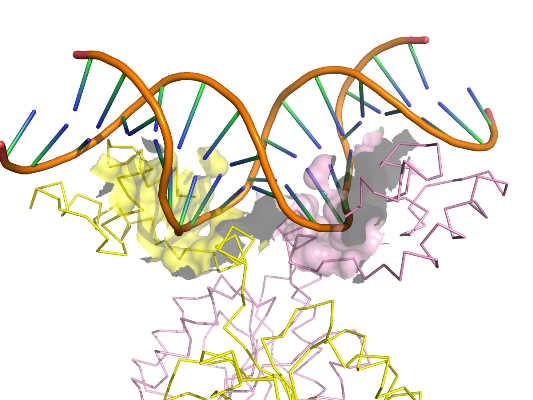
Рис. 3. Изображение поверхности контакта димера белков с двойной спиралью ДНК.
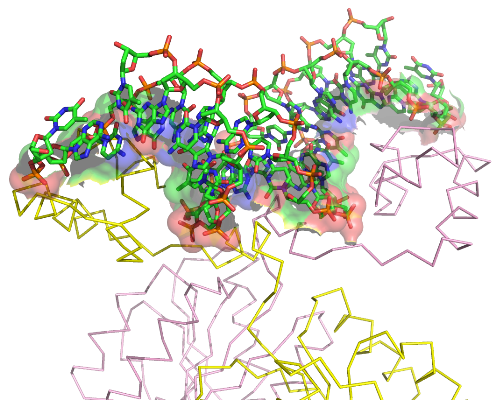
Рис. 4. Изображение поверхности контакта ДНК с димером белков.
Далее с помощью сервиса CluD были найдены гидрофобные кластеры объёмом не менее 10 атомов на интерфейсе мономеров белка в том же комплекс (рис. 5). Была вновь визуализирована поверхность контакта мономера белка с симметричным мономером на фоне остовной модели мономера, но дополнительно была выделена поверхность (синим цветом), относящаяся к атомам, входящим в найденные гидрофобные кластеры (рис. 6)
Рис. 5. Изображение гидрофобных кластеры между мономерами, найденных с помощью CluD.
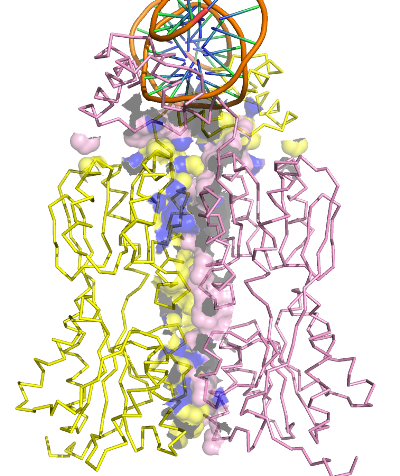
Рис. 6. Изображение поверхностей атомов мономера белка, образующих гидрофобные контакты с другим мономером. Атомы, входящие в гидрофобные кластеры, найденные CluD, окрашены синим.
Сравнение доменов SCOP/SCOPe, ECOD, CATH и Pfam
С помощью SCOP/SCOPe, ECOD, CATH и Pfam была произведена попытка определить домены для структуры мембранного белка 3tui. В SCOP информации о белке нет. В ECOD, Pfam и CATH обнаруживается по 3 домена. В таблице 1 представлены данные о границах домена ABC-транспортера, полученные разными способами. В CATH не выходит посмотреть на найденные домены (ссылка), поэтому в таблице стоит "?", но указан CATH-код. Границы домена, определенные ECOD и Pfam, практически совпадают.
Таблица 1. Границы домена белка 3tui, найденные с помощью SCOP/SCOPe, ECOD, CATH и Pfam.
| Классификация | ECOD | Pfam | CATH |
| Название домена | Type I ABC importer transmembrane domain fold | Binding-protein-dependent transport system inner membrane component | CATH code: 1.10.3720.10 |
| Границы домена | 1-216 | 27-217 | ? |
Использование сайта PDB
Сайт PDB позволяет производить поиск структур по различным параметрам. В этом разделе приведена некоторая демонстрация этого.
С помощью "Advanced search" были скачены все последовательности белков, структуры которых определенны при помощи метода электронной микроскопии, их последовательности в виде одного fasta-файла были скачены из PDB, и они представлены в файле: EM_PDB.fasta.
Далее был проведен поиск структурных гомологов для цепи А белка 3tui с помощью PDBeFold и jFATCAT.
С помощью PDBeFold (с параметрами по умолчанию) было найдено 26 гомолога: по сути это разные цепи 12-ти белков (результат в таблице).
В PDB был найден результат работы алгоритма jFATCAT (ссылка). Показаны только результаты с P-value < 0.001, также установлен порог на идентичность последовательностей 40%. Список включает в себя 76 гомологов. Ни один хит не совпал с таковым из PDBeFold. Однако если в PDBeFold взять порог идентичности по полседовательности 50% (результат в таблице), то некоторые хиты совпадают.