Алгоритмы и программы множественного выравнивания
Петренко Павел
Факультет биоинженерии и биоинформатики, Московский Государственный Университет имени М.В.Ломоносова
Сравнение выравнивания одних и тех же последовательностей тремя разными программами
При выполнении этого задания я решил сравнить работу таких программ множественного выравнивания, как MAFFT, T-coffee и MUSCLE. При этом в качестве референсного выравнивания использовалось выравнивание, полученное при использовании программы MAFFT, так как согласно информации с лекции она имеет наибольший средний вес сравнения с выравниванием BAliBASE, которое принято за "идеальное выравнивание". Выравнивал я белки из домена Cytochrome С (AC: PF00034, ID: Cytochrom_C), с которым я столкнулся в практикуме 3. Все сравниваемые программы были запущены в Jalview со стандартными параметрами. Далее для сравнения выравниваний использовалась программа MACHO на kodomo, разработанная моими однокурсниками Маслениковым Всеволодом, Гагарочкиным Виталием и Нагорным Даниилом.
| Программы | Длина выравнивания MAFFT | Доля одинаково выровненных колонок | Длина второго выравнивания | Доля одинаково выровненных колонок | Одиночные колонки | Координаты блоков в первом выравнивании | Координаты блоков во втором выравнивании |
|---|---|---|---|---|---|---|---|
| MAFFT и T-coffee |
253 | 7.11% | 265 | 6.79% | (63,63) | (13-15) (21-26) (43-44) (107-109) (126-128) |
(13-15) (22-27) (43-44) (112-114) (141-143) |
| MAFFT и MUSCLE | 253 | 2.77% | 144 | 4.86% | - | (14-15) (21-25) |
(16-17) (23-27) |
В результате двух выравниваний видно, что выравнивание программы T-coffee больше похоже на выравнивание программы MAFFT, чем MUSCLE (это видно даже из длины выравниваний, так как выравнивание программы MUSCLE заметно короче). Так, если принимать выравнивание MAFFT за наиболее близкое к "идеалу" BaliBASE, можно сделать вывод, что программа T-coffee работает лучше, чем MUSCLE. Это может быть обусловлено тем, что программа T-coffee комбинирует несколько результатов разных выравниваний (например, ClustalW, LALIGN), создавая библиотеку попарных выравниваний, на основе которых строится итоговое множественное выравнивание. Это позволяет учитывать как глобальные, так и локальные сходства. Однако стоит заметить, что программа MUSCLE работает быстрее, а также она была выпущена позже, чем T-coffee (2004 и 2000 гг).
Fasta-файл с результатом работы программы MAFFT
Fasta-файл с результатом работы программы T-COFFEE
Fasta-файл с результатом работы программы MUSCLE
Построение выравнивания по совмещению структур и сравнение его с выравниванием программой MAFFT из MSA
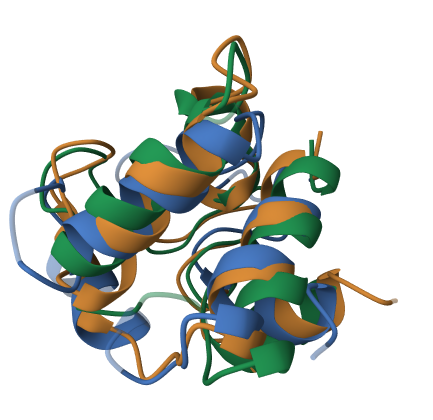
Для выполнения этого пункта я выбрал тоже семейство белков Cytochrome С (AC: PF00034, ID: Cytochrom_C), что и в предыдущем пункте. Затем я выбрал три белка: Cytochrome C-551 from P.Stutzeri Zobell(1CCH), Cytochrome C-553 from Desulfovibrio Vulgaris (1C53) и Ferricytochrome C-552 from Nitrosomonas Europaea (1A56). Затем с помощью подсказок в задании практикума я получил множественное выравнивание на основе совмещения структур, а также выровнял эти последовательности программов MAFFT в JalView. Сравнение двух выравниваний было, как и в предыдущем случае, сделано с помощью программы MACHO на kodomo, сделанной моими однокурсниками. В результате выравнивания видно, что есть одинаковые участки выравнивания, которые соответствуют участкам связывания с лигандом.
| Программы | Длина выравнивания MAFFT | Доля одинаково выровненных колонок | Длина второго выравнивания | Доля одинаково выровненных колонок | Одиночные колонки | Координаты блоков в первом выравнивании | Координаты блоков во втором выравнивании |
|---|---|---|---|---|---|---|---|
| PDB и MAFFT |
92 | 35.87% | 91 | 36.2% | (10,10) (23,23) (36,37) |
(6-8) (12-19) (72-73) (76-92) |
(6-8) (12-19) (72-73) (75-91) |
Описание программы MUSCLE
MUSCLE (Multiple Sequence Comparison by Log-Expectation) — это программа для множественного выравнивания последовательностей, разработанная Робертом Эдгаром в 2004 году. Она сочетает высокую скорость работы с хорошей точностью, и её основным преимуществом является работа с большими объёмами данных. Также MUSCLE поддерживает разные матрицы замен и параметры выравнивания.
Как работает MUSCLE?
Сначала MUSCLE выполняет попарное сравнение последовательностей, вычисляя меру сходства между всеми парами последовательностей (использует при этом метод слов, подобно тому, что мы рассматривали в BLAST). Затем на основе матрицы расстояний строится дерево кластеризации, которое определяет порядок добавления последовательностей в выравнивание. Последовательности добавляются в множественное выравнивание одна за другой согласно дереву, начиная с наиболее похожих.
После этого MUSCLE рекурсивно разбивает дерево на две части и перевыравнивает подгруппы. Алгоритм пытается улучшить вес выравнивания, переставляя последовательности и изменяя gaps. Для более точного учета вероятностей аминокислотных замен используется алгоритм "Log-Expectation" (логарифмического ожидания).
MUSCLE использует эвристики для выделения высококонсервативных участков и их выравнивания. Также происходит параметризация: пользователь может настраивать штрафы за гэпы, матрицы замен и некоторые другие параметры.
Статья, которой я пользовался для выполнения этого пункта задания: MUSCLE: multiple sequence alignment with high accuracy and high throughput Open Access; Robert C. Edgar; Nucleic Acids Research, Volume 32, Issue 5, 1 March 2004, Pages 1792–1797