
В практикуме использовалась программа BLAST, служащая для поиска гомологов белков или нуклеиновых кислот. BLAST является одним из основных инструментов базового биоинформатического поиска, так как позволяет осуществлять поиск по многочисленным базам данных, выравнивать последовательности, строить карты локального сходства и деревья. На вход программе подаётся одна (или несколько, для выравнивания) последовательностей, вносимых в поле Query Sequence. В поле Choose Search Set можно выбирать используемую базу данных, ограничивать и всячески улучшать поиск. Также во вкладке Algorithm parameters можно более тонко регулировать параметры работы алгоритма.
Для выполнения задания была использована последовательность белка сиалидазы Clostridium beijerinckii
С помощью BLAST был произведён поиск по банку SwissProt с различными параметрами. Так как E-value оказывался чрезвычайно высоким, было решено изменить базу поиса на Refseq proteins.
В бнке, после нескольких вариантов поиска, были найдены последовательности, изображённые на рисунке 1.
Для выравнивания последовательностей, 9 избранных были скачаны одним файлом fasta формата с помощью выделения и опции Download.
Из рисунка 1 видно, что выравнивания, в большинстве своём, более идентичны при движении к С-концу. Возможно, блоки впоследствии стоит искать именно там. Из последовательностей было выбрано девять записей с разными значениями E-value (Таблица 1). Условия поиска: Organism: Clostridia (taxid:186801), Max target sequences: 500, Expect threshold: 10, Word size: 3.
| ID/AC | Название белка | Coverage | E-value | Identity | Гомология |
| WP_012058883.1 | coagulation factor 5/8 type domain-containing protein [Clostridium beijerinckii] | 1 | 0.0 | 0,98 | да |
| WP_066463255.1 | adenylyl cyclase [Sanguibacter suarezii] | 0,17 | 1E-150 | 0,47 | нет |
| WP_054244579.1 | licheninase [Actinobacteria bacterium OV320] | 0,15 | 4E-28 | 0,47 | да |
| WP_037851527.1 | coagulation factor 5/8 type domain protein [Streptomyces sp. NRRL S-340] | 0,16 | 7E-24 | 0,46 | да |
| WP_002612399.1 | carbohydrate-binding protein [Stigmatella aurantiaca] | 0,15 | 1E-23 | 0,47 | нет |
| WP_013223239.1 | oxidoreductase [Amycolatopsis mediterranei] | 0,16 | 2E-23 | 0,46 | да |
| WP_076303791.1 | glycosyl hydrolase [Paenibacillus odorifer] | 0,16 | 0.001 | 0,33 | условно |
| WP_076966773.1 | xylosidase [Streptomyces sp. IB2014 011-1] | 0,09 | 1.0 | 0,35 | нет |

Выравнивание 2 демонстрирует результат работы программы выравнивания и даёт представление о возможных гомологичных участках, в которых можно искать блоки.

Для получения наглядных блоков в выравнивании 3 последовательности были построчно поменяны, что не изменило выравнивание. Теперь белки идут в следующем порядке:
Я выделил эти блоки, поскольку каждый из них содержит минимум по три асолютно косервативные позиции, и одну функционально консервативную (третий блок). В блоки входит пять последовательностей. Такое расположение может говорить в пользу гомологии первых пяти и против гомологии оставшихся.
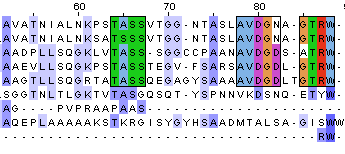
На данном участке можно выделить четыре блока. Четвёртый- длинный удовлетворяет всем критериям блока и ещё раз подтверждает гомологию первых четырёх последовательностей. Пятый и шестой- широкие, также можно считать блоками, дающими основание предполагать гомологию первых шести последовательностей. Седьмой блок также имеет право на существование. Его можно начать с столбца 194.
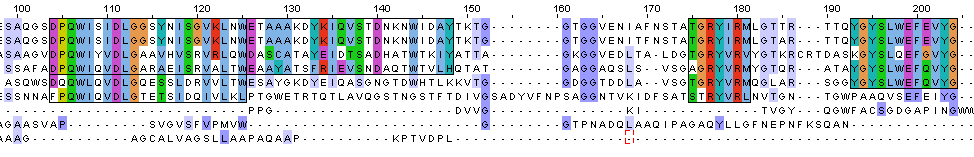
Подводя итог, можно сказать, что первые пять последовательностей, скорее всего, гомологичны. Шестую последовательность я бы считал условно гомологичной, а последние три, судя по блокам, гомологичными считать нельзя. При выполнении оценки я не смотрел на E-value. Оказалось, что шестая последовательность- glycosyl hydrolase, имеет E-value 0.001, что мало для досоверного выравнивания. Последовательности, отнесённые мной к негомологичным имеют E-value: 1.0 (xylosidase), 1E-23 (carbohydrate-binding protein), 1E-150 (adenylyl cyclase). Большое значение E-value для последних двух выравниваний странно в связи с принадлежностью к негомологичной группе. Возможно, это связано с тем, что все последовательности имеют низкое покрытие, около 16% или меньше. Выравнивание, имеющее низкое покрытие и E-value, равный единице, однозначно негомологичное.
Для выполнения задания сначала была выбрана пара белков из первого задания (сиалидаза из Clostridium beijerinckii и крбонат-связывающий белкок из Clostridium sp. D5). С помощью сервиса BLAST в режиме "Align two or more sequences" было построено выравнивание 3 и карта локального сходства (Карта 1). Из карты видно, что ничего интересного в последовательностях нет: они очень похожи. Заметны лишь делеции в последовательности сиалидазы или инсерции в последовательности крбонат-связывающего белка.
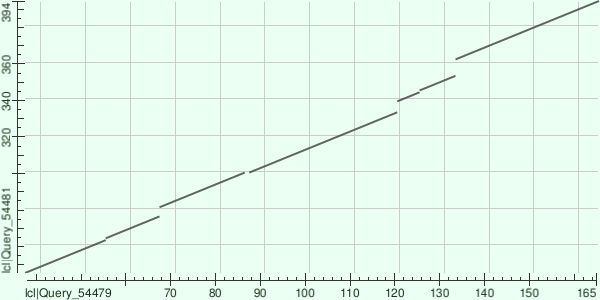



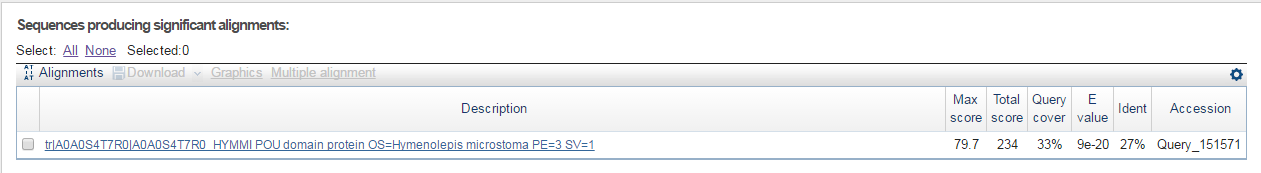
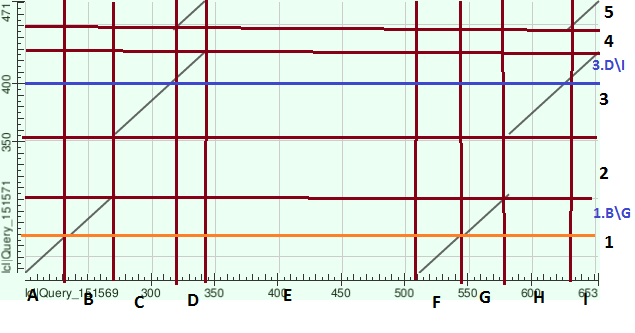
Данные, полученные при выравнивании (Рисунок 6, карта 3), свидетельствуют о наличии двух доменов POU, как на рисунках 4, 5. Видно, что домены идентичны, что может говорить о их дупликации. Заметны одинаковые делеции- индели в более длинной последовательности и два крупный инделя в более короткой последовательности. Попробуем формально описать карту локального сходства. Для этого введём дополнительные обозначения: A, B,.. и 1, 1.B\G, 2, 3, 3.D\I,.. (где 1.B\G и 3.D\I -подпоследовательности) Видно, что участку 1 последовательности из из Larimichthys crocea соответствует два участка второй последовательности, обозначенных A-B и F-G и прерываемых небольшим инделем. Для участков 2, 4 не оказвыается гомологичных последовательностей во втором белке, тогда как участкам 3, 5 соответствуют части последовательности, обозначенные C, D и H, I. Интересно, что участки D и I гомологичны сразу двум частям последовательности из Larimichthys crocea: 3.D\I и 5.

© Кравченко Павел
2017