A- и В- формы ДНК. Структура РНК
Сравнительный анализ канонической ДНК и стеблей тРНК
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
При желании Вы можете насладиться вместе со мной
(В некоторых браузерах не отображается)
Построение моделей структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
Пакет 3DNA позволяет моделировать и проводить анализ нуклеиновых кислот. С помощью программы Putty и пакета 3DNA были построены A-, B- и Z-формы двунитевой ДНК с последовательностью "GATCGATCGATCGATCGATC"
Ссылки на файлы
Сравнение структур 3-х форм ДНК с помощью средств JMol
Упражнение 1
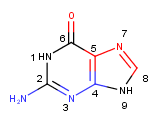
В апплете 1 показан гуанозилмонофосфат, имеющий в составе остатка азотистое основание- гуанин, изображённый на рисунке 1.
При изучении В формы ДНК видно, что нуклеотиды имеют атомы, направленные в и разделяющие большую и малую бороздки.
В сторону большой бороздки явно обращены атомы g25.n2 ([DG]25:B.N2)
при g25.c2 (DG]25:B.C2) атоме, g25.n3 ([DG]25:B.N3), g25.c4 ([DG]25:B.C4).
В сторону большой бороздки направлены
явно атомы g25.О6 ([DG]25:B.O6) при g25.c6 ([DG]25:B.C6) атоме,
g25.c5 ([DG]25:B.C5), g25.n7 ([DG]25:B.N7), g25.c8 ([DG]25:B.C8).
g25.n1 ([DG]25:B.N1) атом направлен по радиусу спирали при рассмотрении с
торца.
g25.n9 ([DG]25:B.N9)
атом связан N-гликозидной связью с дезоксирибозой.
Упражнение 2
В таблице 1 представленны основные характеристики трёх форм ДНК.
|
A-форма |
B-форма |
Z-форма |
| Тип спирали | Правая |
Правая |
Левая |
| Шаг спирали (А) | 28,03 [DC]12:A.P-[DG]1:A.P |
33,75 [DC]20:A.P-[DA]10:A.P |
43,50 [DG]1:A.P-[DG]13:A.P |
| Число оснований на виток | 11 |
10 |
12 |
| Ширина большой бороздки (А) | 16,81 [DT]11:A.P-[DC]32:B.P |
17,21 [DC]12:A.P-[DA]26:B.P |
16,08 [DC]8:A.P-[DC]32:B.P |
| Ширина малой бороздки (А) | 7,980 [DC]32:B.P-[DT]3:A.P |
11,31 [DA]18:A.P-[DA]26:B.P |
7,20 [DG]11:A.P- [DG]33:B.P |
Сравнение структур 3-х форм ДНК с помощью средств JMol
Упражнение 1. Определение торсионных углов нуклеотидов
Для выполнения заданий практикума были взяты структуры рибосомального комплекса терминации с фактором высвобождения 2 и ДНК-связывающий домен эндонуклеазы интронов. Идентификаторы записей в базе PDB- 1ml5 и 1I3J соответственно. Файлы был преобразован в старый формат .pdb с помощью программы remediator командой "remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb"
С помощью пакета 3DNA были получены торсионные углы структур тРНК и ДНК. В программе Excel были определены средние значения для каждого из
торсионных углов, исключая краевые нуклеотиды. Исходя их полученных данных, можно заключить, что данная тРНК по большинству значений углов совпадает с А-формой ДНК.
Средние значения торсионных углов для каждой из цепей молекул приведены в таблице.
Смамыми деформированными нуклеотидами ДНК,
основываясь на нескольких сильно отличающихся углах, являются: для первой цепи
- 5G, 9C; для второй цепи- 2G, 12G, 13G, 14C.
Смамыми деформированными нуклеотидами РНК,
основываясь на нескольких сильно отличающихся углах, являются: для первой цепи
- 28G. 19A; для второй цепи- 12A, 13G, 24G.
Упражнение 2. Определение структуры водородных связей
В данной тРНК с помощью пакета 3DNA было найдено 4 стеблевых участка, составляющих третичную структуру молекулы. Можно сопоставить полученные данные с пространственной структурой, обратив внимание не рисунок 2[3].
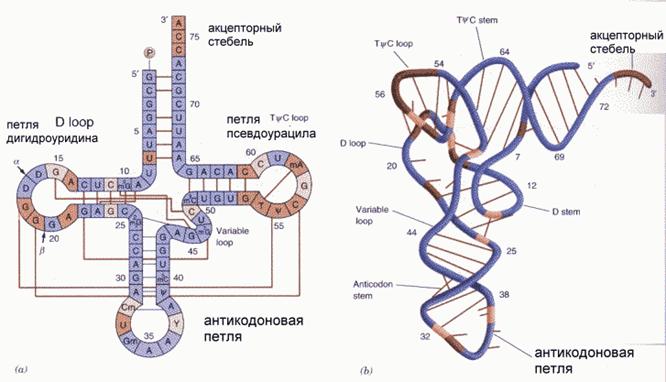
В последовательности нуклеотидов тРНК содержатся сложные участки, стабилизирующие третичную структуру.
Также в тРНК содеоржатся неканонические пары оснований. Так, имеются связи G-U, Псевдоуредин-G, A-C, Псевдоуредин-A, A-G, G-U, некоторые из которых обсуждались в предыдущей работе.
Упражнение 3. Поиск стекинг-взаимодействий
Для выполнения задания практикума была взята другая структура - структура комрлекса глутамил-тРНК синтазы с тРНК. Идентификатор записи в базе PDB- 1G59. Файл был преобразован в старый формат .pdb с помощью программы remediator командой "remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb"
С помощью программ find_pair и analyze был произведён анализ файла 1g59_out.pdb. Для этой цели был использован пайплайн "find_pair -t 1g59_out.pdb stdout | analyze". Из файлов, полученных в результате работы программ, были проанализированны файлы 1g59_out.out и staking.pdb. В первом были отобраны пары нуклеотидов с наибольшей и наименьшей площадями перекрывания. Из второго, с помощью команд "ex_str -8 stacking.pdb step*.pdb" и "stack2img -cdolt step*.pdb step*.ps", были получены файлы .ps формата, найти которые вместе с конвертированными в .png, при желании, Вы можете в директории.
Рисунки 3-5 демонстрируют области с большим интегралом перекрывания между нуклеотидами. А рисунки 6-8 - с ничтожным. Прекрасно видно, что в одном случае нуклеотиды хорошо перекрываются, реализуя стекинг-взаимодействие, тогда как в другом пары либо не взаимодействуют, либо взаимодействуют слабо.
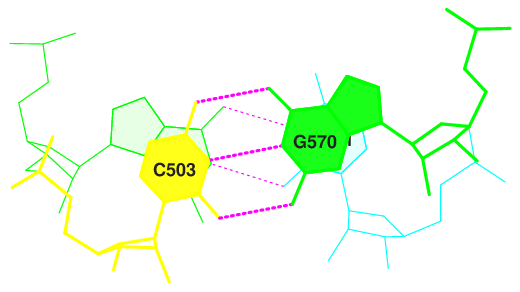
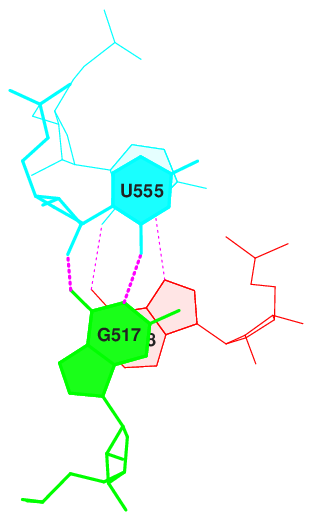
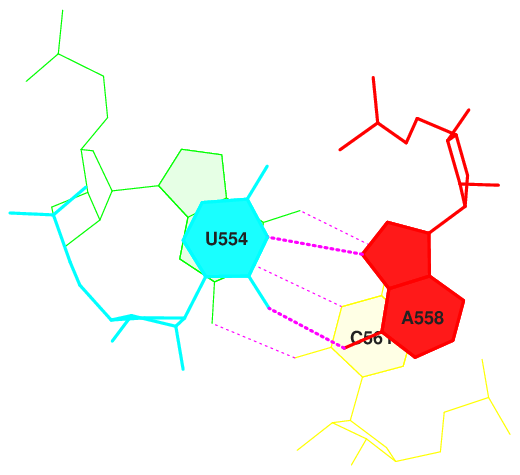
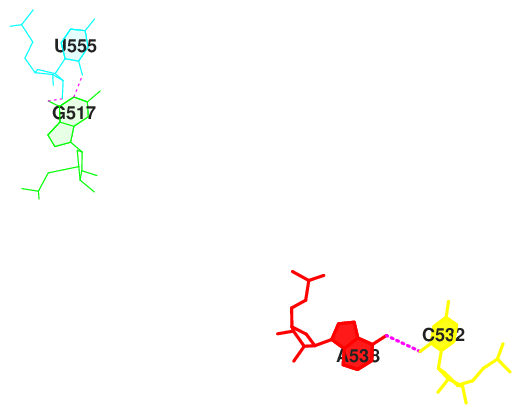
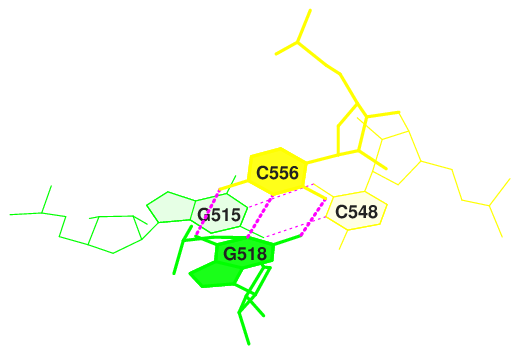
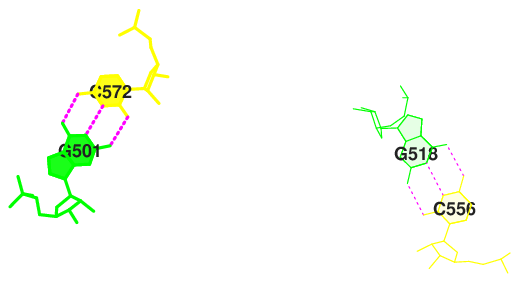
Апплет демонстрирует пространственное расположение описанных нуклеотидных пар в молекуле тРНК. Стоит заметить, что, как правило, пары, хорошо участвующие в стекинг-взаимодействии (выделены жёлтым), и пары, не участвующие в нём (выделены зелёнфм), располагаются рядом. По всей видимости, они обеспечивают поддержение и поворот целостной структуры тРНК.
Ссылки
1. GLUTAMYL-TRNA SYNTHETASE COMPLEXED WITH TRNA(GLU)
3. тРНК
