Филогенетическое дерево
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Построение дерева выбранных организмов
Поиск ортологов CLPX_ECOLI.
Филогенетическое дерево - это дерево, отражающее эволюционные взаимосвязи между различными
видами или другими объектами, предположительно имеющими общего предка.
Филогенетическое дерево состоит из трёх классов объектов: листьев, узлов и, в зависимости от метода построения, корня.
Листья — это конечные вершины отображающий, как правило, некоторый вид организмов, объект исследования.
Каждый узел представляет эволюционное событие: разделение предкового вида на два или более, которые в дальнейшем эволюционировали независимо.
Корень представляет собой предполагаемого общего предка всех рассматриваемых объектов. Ребра филогенетического дерева называют ветвями.
Для выполнения практикума было выбрано семь организмов из предложенной таблицы:
|
Скобочная формула дерева
Дереву можно однозначно сопоставить скобочную формул:
(((((ECOLI, YERPE), PROMH), HAEIN), NEIMA), (RHOS4, RHIEC));
Изображение дерева
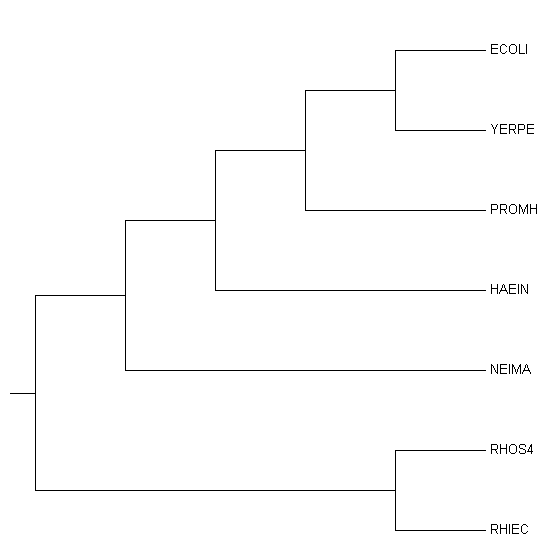 |
Для изображения дерева использовалась программа Mega 7 [1].
Ветви дерева
Дерево содержит нетривиальные ветви:
{ECOLI, YERPE} vs {PROMH, HAEIN, NEIMA, RHOS4, RHIEC}
{ECOLI, YERPE, PROMH} vs {HAEIN, NEIMA, RHOS4, RHIEC}
{ECOLI, YERPE, PROMH, HAEIN} vs {NEIMA, RHOS4, RHIEC}
{ECOLI, YERPE, PROMH, HAEIN, NEIMA} vs {RHOS4, RHIEC}
Реконструкция филогении
Была составлена таблица таксономического положения видов с помощью раздела NCBI Taxonomy. Все выбранные организмы относятня к царству Proteobacteria из домена Bacteria.
|
При взгляде на дерево видно, что некоторые нетривиальные ветви выделяют отдельные таксоны, что говорит о адекватности нарисованного нами дерева, а также указывает на возможную эволюцию бактерий.
Отдельно нетривиальные ветви выделяют следующие таксоны:
Класс Gammaproteobacteria - {ECOLI, YERPE, HAEIN, PROMH} vs {NEIMA, RHOS4, RHIEC}.
Класс Alphaproteobacteria - {RHIEC, RHOS4} vs {HAEIN, PROMH, NEIMA, RHOS4, RHIEC}.
Отряд Enterobacterales - {ECOLI, YERPE, PROMH}.
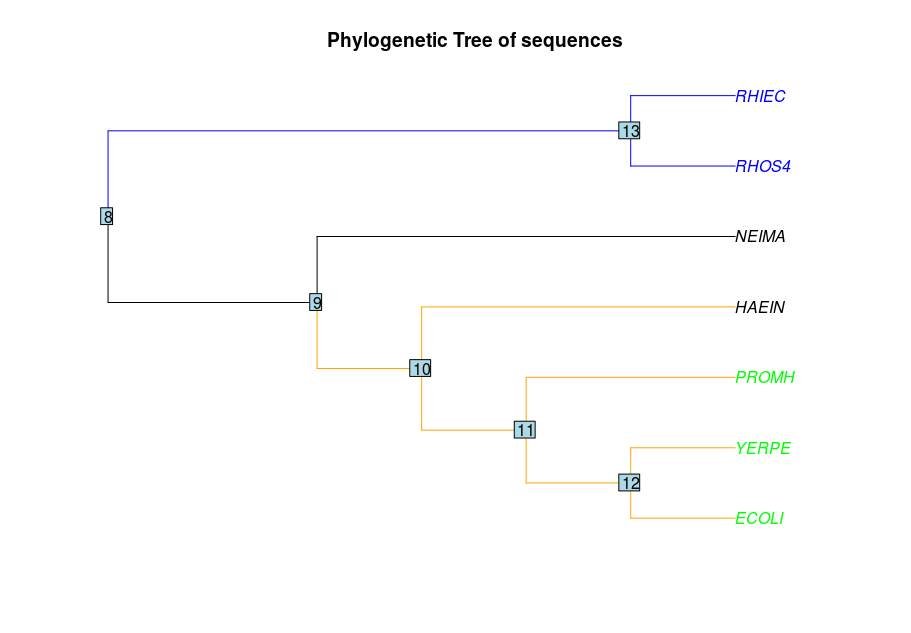 |
Реконструкция филогении
Для выполнения задания в файл RL1.fasta из банка данных Uniprot для отобранных организмов были скачаны последовательности белков семейства RF1.
Последовательности были выровнены с помощью команды muscle -in RL1.fasta -out RL1_aligned.fasta
С помощью различных алгоритмов программы Mega были построены следующие деревья.
Maximum Likelihood
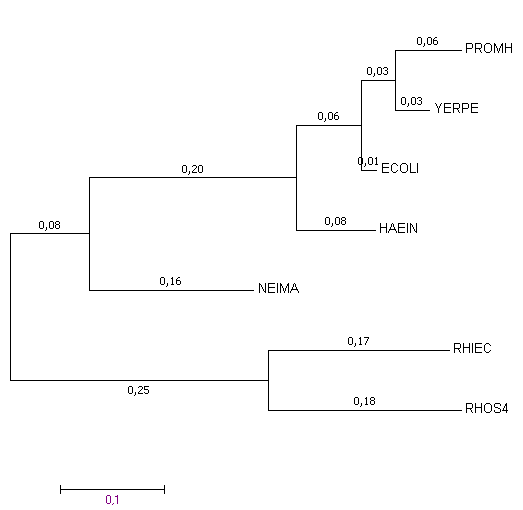 |
Neighbor-joining
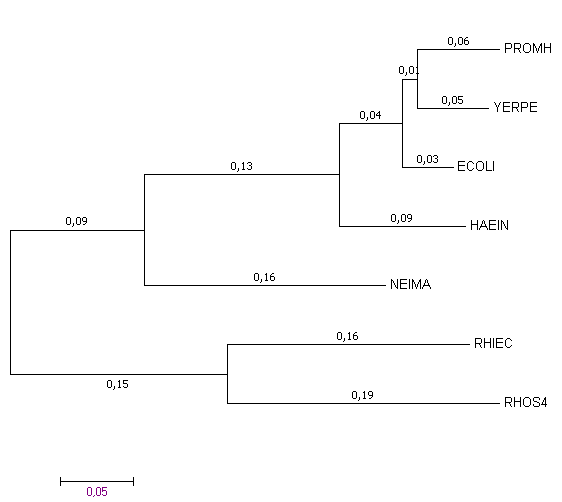 |
Minimum Evolution method
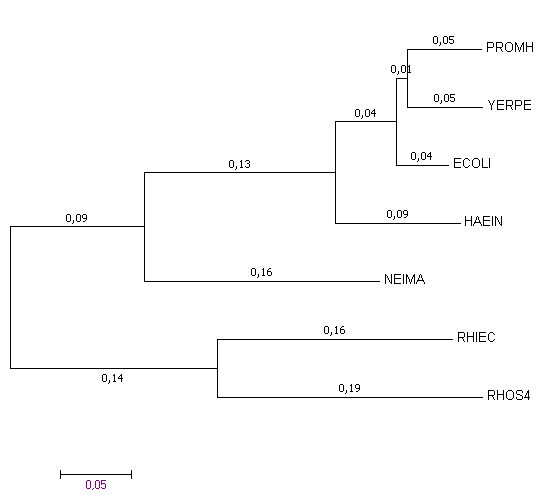 |
Анализируя полученные выравнивания, можно заключить, что три метода, в данном случае, работают практически одинаково. Различие обнаруживается лишь в длинах ветвей. Методы реконструкции хорошо справились со своей задачей. Деревья отличаются от принятого нами за истинное одной перестановкой тривиальных ветвей.
Файлы в формате .newick лежат в директории. Ссылка на дополнительное задание
