Структура белка β-порфириназы из генома бактерии Zobellia galactanivorans
Ниже приведена структура(рис. 1.), полученная при обработке в JMol(http://jmol.sourceforge.net) файла 4ATE, содержащего информацию о пространственной структуре белка β-порфириназы A, скачанного с сайта PDB. Все аминокислоты и низкомолекулярные вещества покрашены в разные цвета. К сожалению, особо важной информации подобное изображение не несет, поэтому размещено на этой странице просто для интереса. Ниже вы сможете найти другие представления данной структуры, помогающие получить более детальную информацию о белке. Чтобы скачать скрипт, с помощью которого получена эта структура, пройдите по этой ссылке.
Внимание! Для того, чтобы скрипт работал корректно, советую после запуска Jmol и открытия файла белка перезагрузить это изображение(File->Reload), так как разные версии JMol имеют разные настройки по умолчанию. Эта рекомендация касается и скриптов, приведенных ниже.
Рис. 1. Структура белка, обработанная в Jmol. Аминокислоты и низкомолекулярные молекулы покрашены в разные цвета
С помощью скрипта, приведенного ниже(смотреть скрипт), получена уже более интересное изображение белка(рис 2).
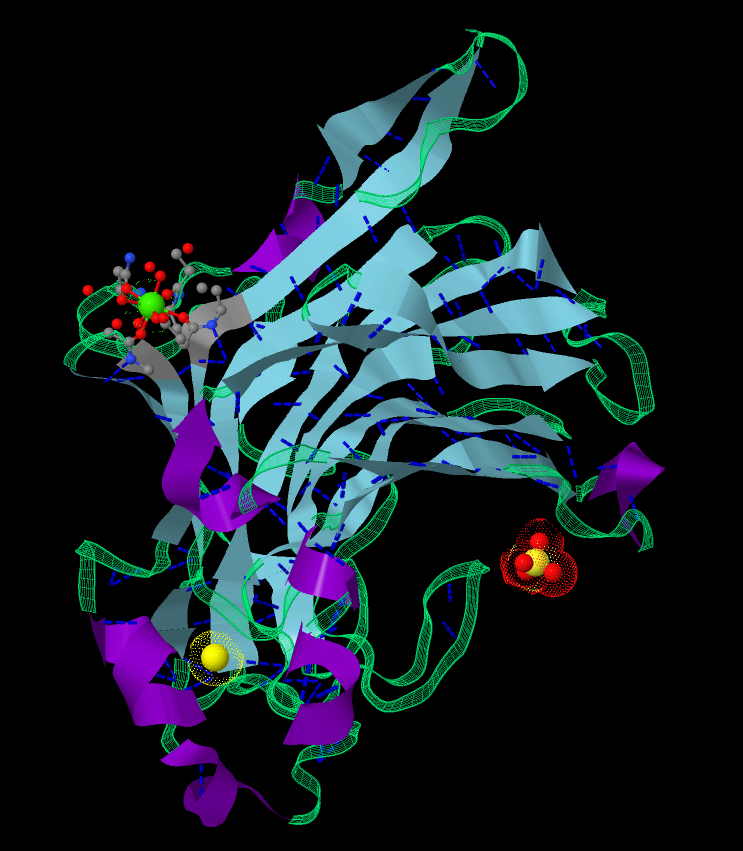
Рис. 2. Структура белка, обработанная в Jmol. α-спирали и β-листы покрашены в разные цвета(соответственно "Darkviolet" и голубой ). К аминокислотам, не входящим в спирали и листы применена команда meshribbon и окрашивание в зеленый цвет. Атом кальция зеленый, хлора - желтый, фтом S-тоже желтый, но он находится в окружении 4 кислородов(красный цвет). Вода не показана. Водородные связи изображены пунктиром и окрашены в синий цвет.
Скрипт, обрабатывающий файл 4ATE.pdb и выдающий в результате изображение, приведенное выше(Рис. 2.)
.select all cpk off cartoons off wireframe off backbone off select protein and not helix and not sheet meshribbon color meshribbon [0,230,117] select sheets or helix cartoons on select sheets color [128,209,227] select helix color Darkviolet select all calculate hbonds hbonds on color hbonds blue translucent 0.2 hbonds 35 select Cl dots spacefill IONIC color yellow define names within (5.0, Ca) select names color cpk cpk 100 wireframe 35 select Ca dots spacefill IONIC color cpk select SO4 dots spacefill IONIC color cpk zoom 55 rotate X 26 rotate Y 52 rotate Z 40
Скачать скрипт можно по этой ссылке
Ниже представлено анимированное изображение белка, полученное при помощи скрипта, упомянутого выше, и скрипта, взятого с сайта http://wiki.jmol.org/index.php/Creating_Movies, а также программы UnFREEz. Текст использованного скрипта приведен ниже(смотреть скрипт)
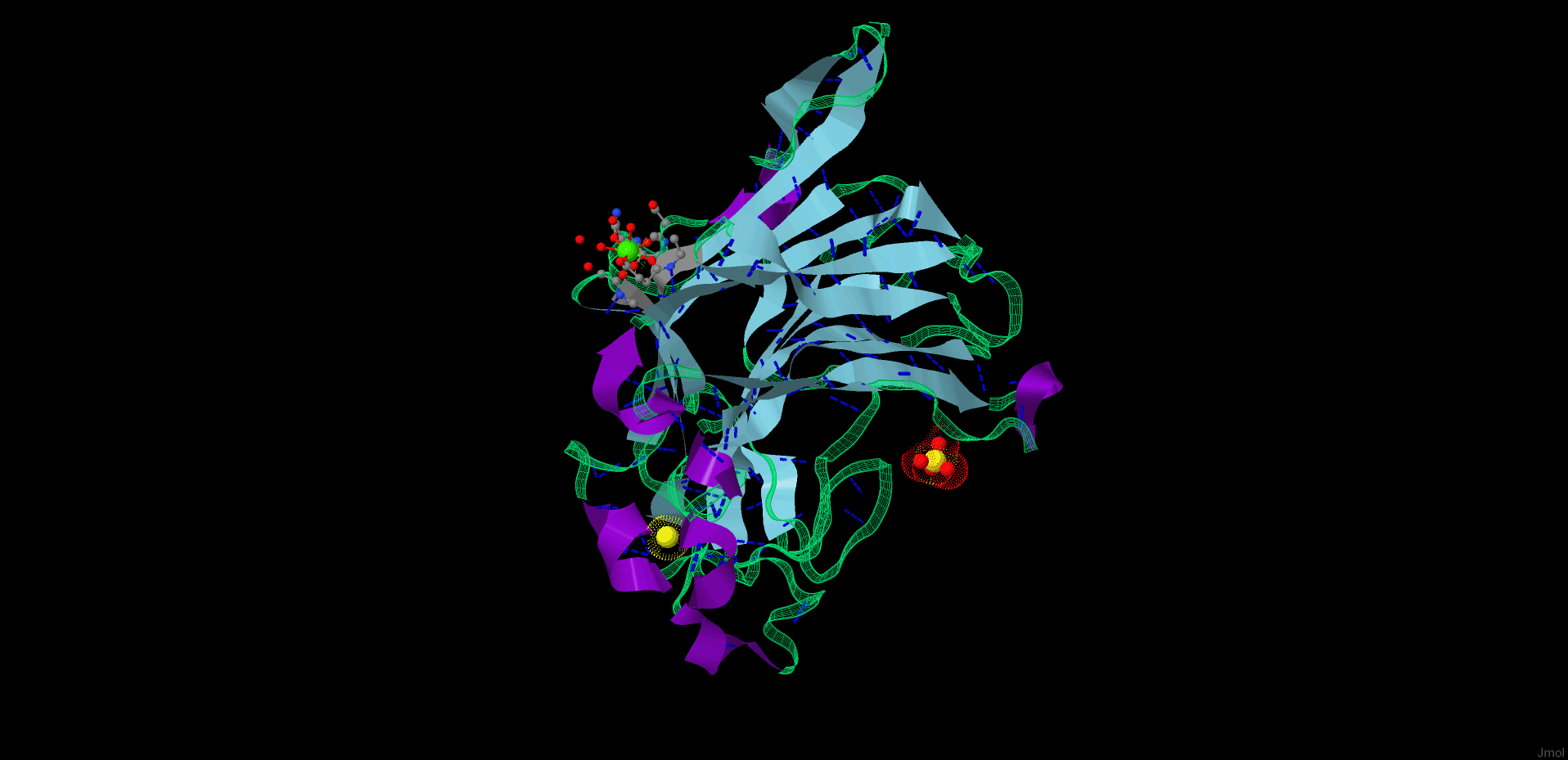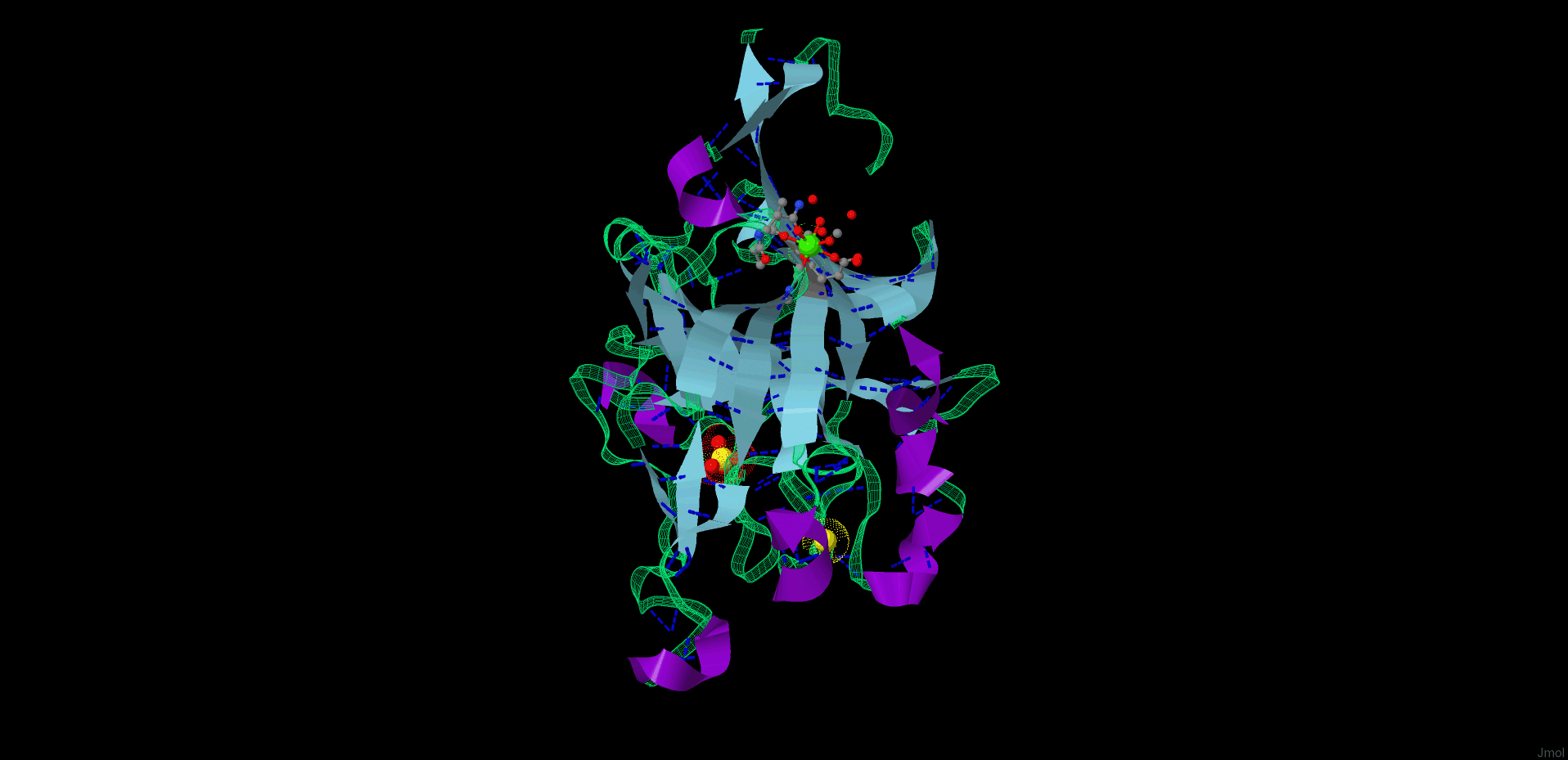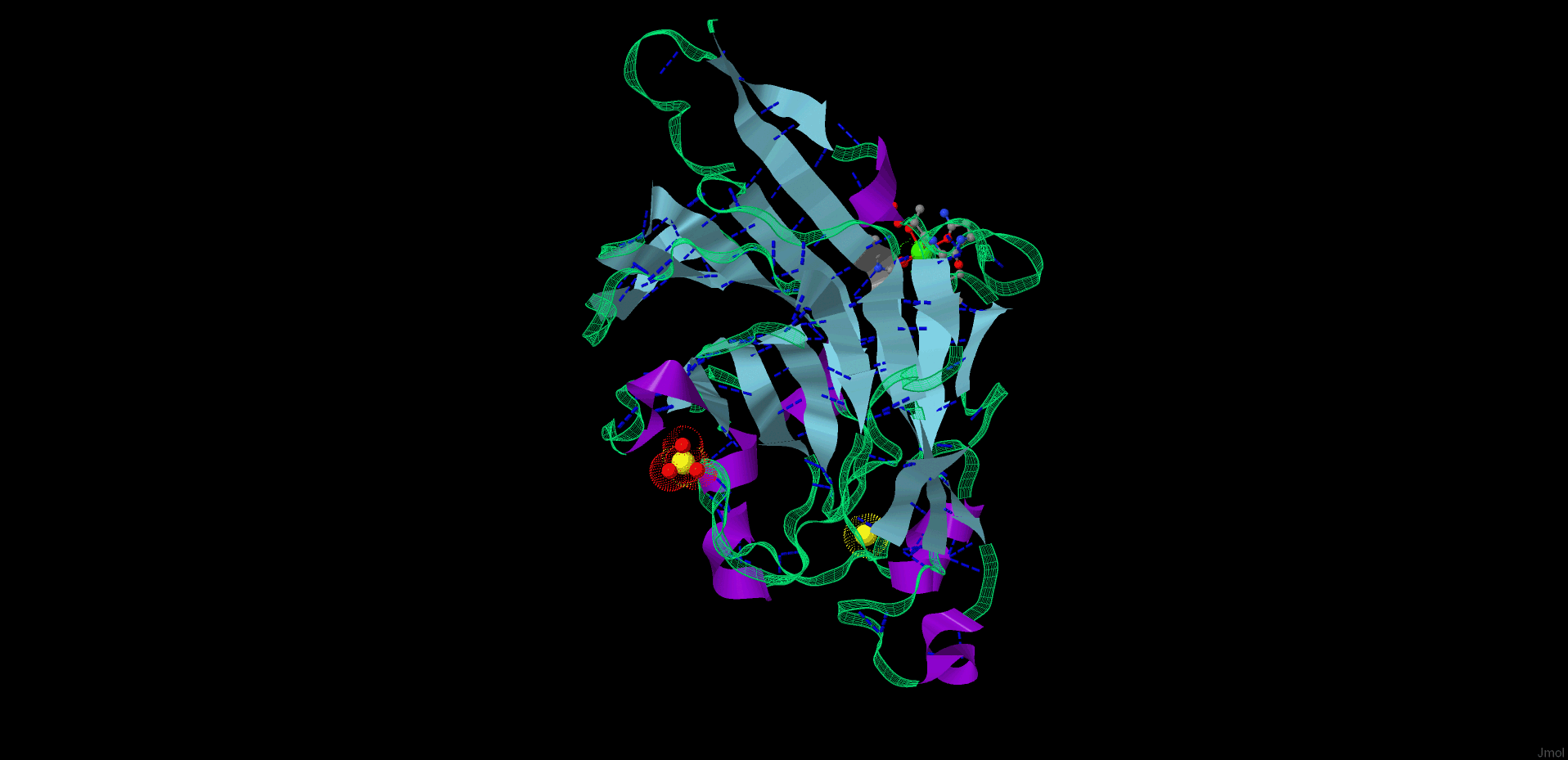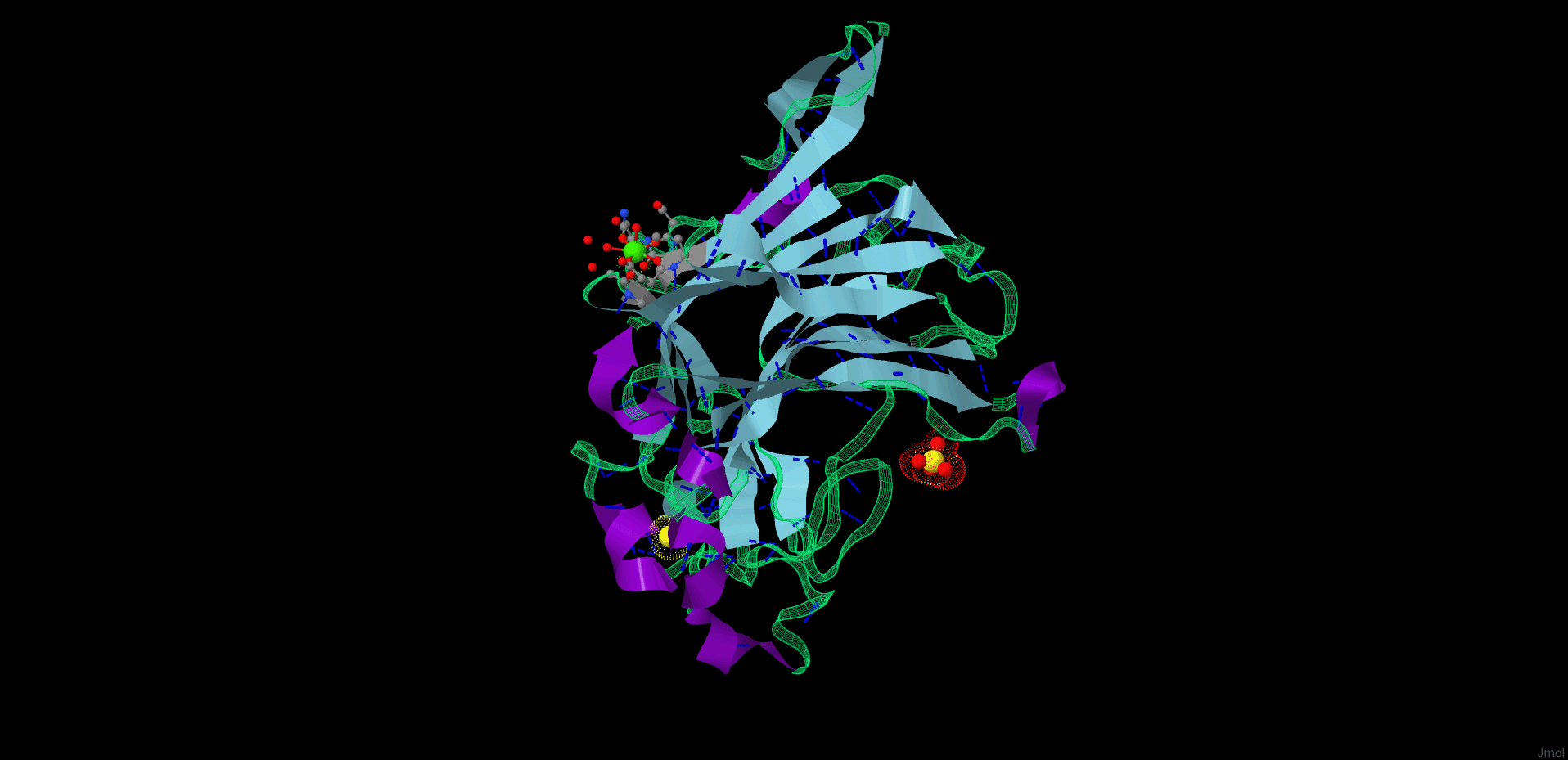
Рис. 3. Анимированное изображение молекулы белка.
Скачать анимированное изображение белка можно по этой ссылке
Скрипт, обрабатывающий файл 4ATE.pdb и выдающий в результате изображение, приведенное выше(Рис. 3.)
.
# goal: a 360 degree spin
~degstep = 5 # or 2, degrees per step
~degrees = 0 # total degrees rotated
while (~degrees < 360)
{
# SPIN
rotate y @~degstep
~degrees += ~degstep
# DISPLAY/WRITE FRAME
refresh
~whereami = "degrees = " + ~degrees
print ~whereami
~fileprefix = "jpg-frames/"
# UNCOMMENT TO GENERATE EXTRA FRAMES REQUIRED FOR WMV.
# COMMENT OUT FOR MP4.
~degreesplus = 1000 + ~degrees
~towrite = ~fileprefix + "m" + ~degreesplus + ".jpg"
# UNCOMMENT TO GENERATE FRAMES FOR MP4 (AND WMV).
#COMMENT OUT TO GENERATE EXTRA FRAMES FOR WMV.
~towrite = ~fileprefix + "m" + ~degrees + ".jpg"
# WRITE THE IMAGE
write image jpg @~towrite
}
Информация об этом белке, полученная с помощью команды grep из файла 4ATE.pdb размещена ниже в двух вариантах - оригинал на английском(таблица 1), и перевод на русский(таблица 2).
Файл с краткой информацией о белке можно скачать по этой ссылке
Таблица 1. Информация из файла 4ATE.pdb в оригинале(на английском).
| Поле | Значение |
| Title | High resolution crystal structure of β-porphyranase a from zobellia galactanivorans |
| Compnd | Mol_id: 1; Molecule: β-porphyranase a; Chain: a; Fragment: catalytic domain, residues 18-277; Synonym: β-porphyranase a\, family gh16; Ec: 3.2.1.178; Engineered: yes |
| Expdta | X-ray diffraction |
| Remark 2 | Resolution. 1.10 angstroms. |
| Hetnam | Ca calcium ion ; SO4 sulfate ion; Cl chloride ion |
| Formul | Ca: Ca 2+ ; SO4: O4 S 2- ; Cl: Cl 1-; HOH: *450(H2 O); |
Таблица 2. Информация из файла 4Ate.pdb, переведенная на русский.
| Поле | Значение |
| Название | Кристаллическая структура белка β-порфираназы, полученной из бактерии Zobellia galactanivorans |
| Информация о молекуле | Идентификатор молекулы: 1; Название молекулы: β-порфираназа a; Цепи: a; Фрагменты: каталитический домен, Аминокислотные остатки: 18-277; Синоним: β-порфираназа а, семейство: gh16; Код фермента: 3.2.1.178; |
| Метод получения структуры | Рентгеноструктурный анализ |
| Разрешение анализа | 1.10 ангстрем. |
| Информация о низкомолекулярных веществах, входящих в состав молекулы | Ca ион кальция ; SO4 сульфат-ион; Cl хлорид-ион |
| Состав | Ca: Атомы: Ca; Заряд: 2+ ; SO4: Атомы: O4 S; Заряд: 2- ; Cl: Атомы: Cl; Заряд: 1-; HOH: Атомы: *450(H2 O) Заряд: 0; |
Сравнение исходного белка с его биологической единицей (biological assembly/unit)
Скачанный мною файл, содержщий биологическую единичу белка, я открыл в Jmol и применил к нему второй скрипт. При простом просмотре отличий незаметно(рис. 4). Чтобы убедиться в правильности своих выводов я использовал программу Structure alignment.
Ниже представлен результат сравнения молекулы и биологической единицы белка. Сравнение проводилось с помощью программы Structure alignment.
Легко увидеть, что они ничем не отличаются друг от друга. На рис. 5 представлен результат работы программы в текстовом режиме, на рис. 6 представлена графическая интерпретация.
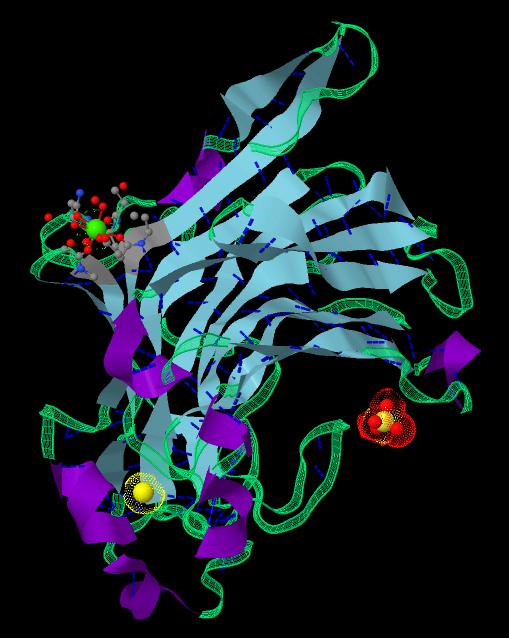
Рис. 4. Изображение биологической единицы белка, полученное при обработке в JMol(http://jmol.sourceforge.net)

Рис. 5. Результат работы Structure alignment(текстовый режим).
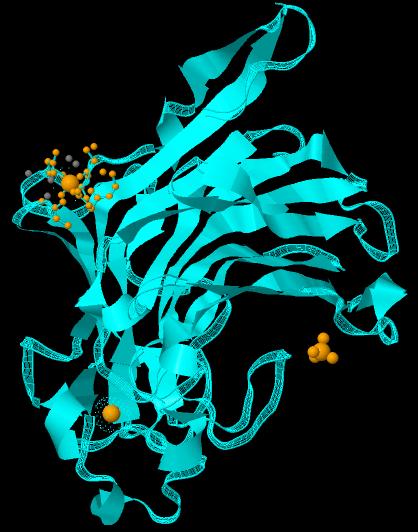
Рис. 6. Результат работы Structure alignment(графический режим).
На рис.7 и рис.8 результаты работы моего скрипта красящего белок и его модификации(подробнее о скрипте здесь). Так же вам интересен может быть этот скрипт.

Рис. 7. Результат работы скрипта.
Рис. 8. Результат работы модифицированного варианта скрипта.
Больше информации об этом белке можно узнать, пройдя по этой ссылке
Так же вас может заинтересовать эта статья
Огромное спасибо Дарье Дибровой за помощь в создании анимированного изображения белка и Ирине Хвень(http://kodomo.fbb.msu.ru/~partyhard/index.html) за то, что помогла разобраться с Structure alignment.
