Практикум 11
Выбор домена, подсемейства и подготовка данных для работы
Краткая информация по выбранному домену:
- AC: PF18933
- Название: PsbP-like protein
- Описание и функции: является родственным к семейству PsbP (белки тилакодной субъединицы фотосисистемы 2; DOI: 10.1007/s11120-008-9359-1)
- Seed: 5
- Full: 238
- Файл со всеми последовательностями: PF18933-full-238.fasta
Для построения профиля я выбрал архитектуру, представленную белком K7VUZ8. В ней было 156 последовательностей. Я выровнял их алгоритмом MAFFT, затем вырезал участок, содержащий два домена, и выровнял его еще раз (сначала через MAFFT, а потом через MUSCLE, так как мне не понравилось начало нового выравнивания). Я удалил те последовательности, из-за которых возникали длинные индели в участках доменов, и установил порог для Remove redundancy 90%. После всех этих преобразованией в конченой выборке осталось всего 96 белков.
- файл выравнивания белков, содержащих домен, в формате fasta
- последовательности белков с выбранным доменом
- последовательности без выбранной архитектуры
- файл со всеми белками, обладающими выбраной архитектурой
- файл с отобранными для построения профиля белками (arch_redact.fa)
Работа с HMM
Ниже представлены команды, с помощью которых я создал и откалибровал профиль (длина 525), а также провел поиск по PF18933-full-238.fasta:
- hmm2build hmm2_profile arch_redact.fa
- hmm2calibrate hmm2_profile
- hmm2search --cpu=1 hmm2_profile full-seq-238.fasta > hmm2_res.txt
Результат работы HMM2 для тестовой выборки и отрицательного контроля.
Анализ полученных данных
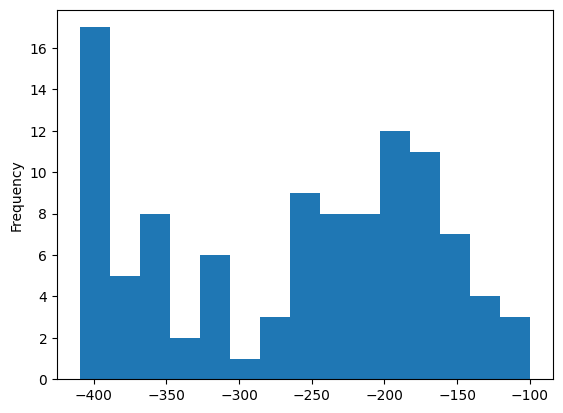
Я записал таблицу из hmm2_res.txt в отдельный файл и отредактировал её, чтобы облегчить дальнейшую работу (ссылка на таблицу). Аналогичная таблица была сделана для отрицательного контроля. Далее используя python, я добавил к этой таблице столбцы: has_architecture и in_profile, где 1 соответствует True, а 0 - значению False (ссылка на блокнот в Colab). Работу с новой таблицей я продолжил в excel, где посчитал значения чувствительности и специфичности, по которым построил ROC-кривую. По данному графику (см. Рис. 1) и гистограмме весов последовательностей я установил порог веса равным -270. Итоговая таблица.