1. Поиск гипотетических гомологов изучаемого белка в разных банках
Таблица 1. Результаты поиска гипотетических гомологов белка CLPQ_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) | |||
| Accession | P39070.1 | 1YYF_D | NP_389497.1 |
| E-value | 4e-129 | 3e-130 | 2e-127 |
| Вес (в битах) | 367 | 367 | 367 |
| Процент идентичности | 100% | 100% | 100% |
2. Число находок с E-value < 10–10 |
314 | 7 | 1823 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 344 | 13 | 1862 |
| Accession | A3DN21.1 | 1Q5Q_H | YP_004109282.1 |
| E-value | 0.97 | 0.17 | 0.96 |
| Вес (в битах) | 32.7 | 32.0 | 37.4 |
| % идентичности | 26% | 33% | 26% |
| % сходства | 44% | 50% | 45% |
| Длина выравнивания | 175 | 70 | 121 |
| Координаты выравнивания (от-до, в запросе и в находке) | запрос:1-161, находка:13-174 | запрос:7-74, находка:1-66 | запрос:20-139, находка:11-114 |
| Число гэпов | 27 | 6 | 18 |
В ходе работы BLAST по алгоритму blastp исходный белок P39070 был найден в базах данных Swiss-Prot и "nr", а его структура в PDB, но отличаются идентификаторы, т.к. белок является частью большего белка, и существует несколько записей в базах данных, относящихся к последовательности этого белка, причем для некоторых его последовательность является подпоследоваетельностью.
Число возможных гомологои вильно различается от бызы к базе, причиной этого служит размер баз, т.к. не все белки, включенные в nr входят в (имеют четкое описание) Swiss-prot и ещё меньше их них входят в PDB (т.е. у ещё меньшего к-ва определена структура).
Всего находок по всем базам(nr,swiss-prot,PDB) 2329, E-value самой последней находки 9.8, чилсло находок было лиметировани сандартным значением E-value в 10 едениц, т.к. предельный размер выдачи был изменен, для просмотра всей выдачи вцелом.
2. Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Таблица 2. Результаты поиска гипотетических гомологов белка CLPQ_BACSU в базе данных swiss-prot в других таксонах
| Поиск по Swiss-Prot | |||
Находка в 'Actinobacteria' (другой отдел того же царства бактерий) |
|||
| Номер находки в списке описаний | 1 | ||
| Accession | A1R6Q7.2 | ||
| E-value | 8e-05 | ||
| Вес (в битах) | 40.8 | ||
| % идентичности | 33% | ||
| % сходства | 50% | ||
| Длина выравнивания | 75 | ||
| Координаты выравнивания (от-до, в запросе и в находке) | запрос:2-74, находка:42-112 | ||
| Число гэпов | 6 | ||
Описание выполнения работы: Поиск произведен по базе данных "swiss-prot", т.к. именно она является самой рецензируемой, а соответственно в большинстве случаев результат поиска по этой базе более адекватен.
E-value достаточно мало, что бы быть уверенным, что найденный результат не случаен, а идентичность явно превышает порог случайного сходства.
Вывод:найденный белок действительно является гомологом.
3. BLAST двух последовательностей
Карта локлального сходстава парного выравнивания последовательности белка CLPQ_BACSU и его гомолога из отдела Actinobacteria: белка PSB_ARTAT, при E-value равном 10 и использовании матрицы замен BLOSUM62
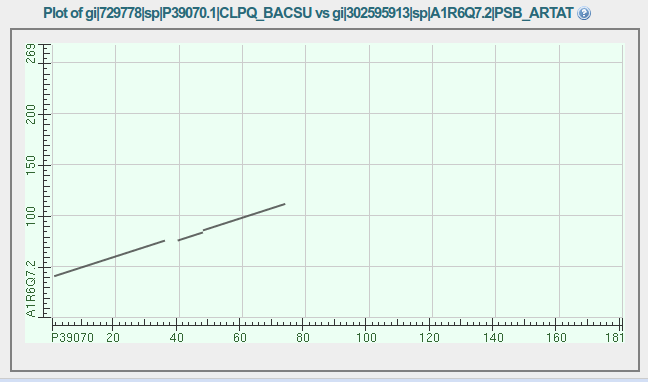
Карта локлального сходстава парного выравнивания последовательности белка CLPQ_BACSU и его гомолога из отдела Actinobacteria: белка PSB_ARTAT, при E-value равном 0.01 и использовании матрицы замен BLOSUM62
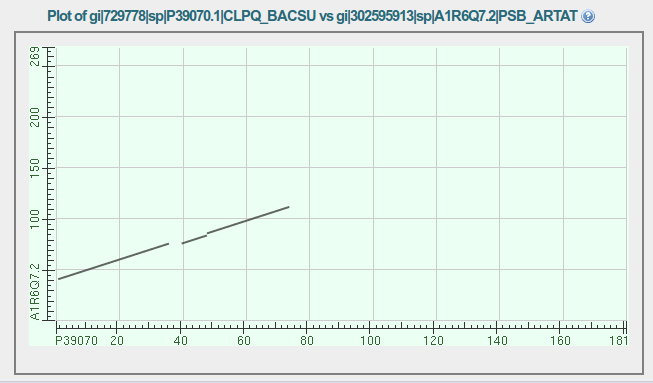
4. Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам при использовании матрицы BLOSUM45.
Таблица 3. Результаты поиска гипотетических гомологов белка CLPQ_BACSU в базе данных swiss-prot в других таксонах при использовании матрицы BLOSUM45.
| BLOSUM45 | |
| Таксон, где найдено | Actinobacteria |
| Номер находки в списке описаний | 1 |
| Accession | A1R6Q7.2 |
| E-value | 3e-05 |
| Вес (в битах) | 41.7 |
| % идентичности | 37% |
| % сходства | 50% |
| Длина выравнивания | 73 |
| Координаты выравнивания (от-до, в запросе и в находке) | запрос:2-74, находка:42-112 |
| Число гэпов | 2 |
Вывод:Сам результат (т.е. найденных белок) остался прежним, но изменился результат выравнивания.
Различие между матрицами аминокислотных замен BLOSUM62 и BLOSUM45 состоит пороге кластеризации,
т.е. в том проценте сходства (который и указывается в названии матрицы замен) при привышении которого выравнивания,
обладающие таким процентом сходства, кластеризуются (т.е. фактически приравниваются к одному) в рамках матрицы BLOCKS,
в результате обработки которой и формируется матрица замен. Таким образом при выравнивании сходных последовательностей стоит использовать матрицы с меньшим процентом идентичности, после которого начинается кластеризация, а для выравнивания сильно различных последовательностей стоит использовать матрицы с большим числом.
В результате изменения коэффициетов стоимости замен аминокислот в матрице замен немного были изменены результаты хода
алгоритма выравнивания на каждом его шаге.
Из за этого поменялось E-value, и вес выравнивания (а как результат и ibentity):
| сравниваемая величина | BLOSUM62 | BLOSUM45 |
| E-value | 8e-05 | 3e-05 |
| идентичность,% | 33 | 37 |
| сходство,% | 50 | 50 |
5. Сравнение реализации веб-интерфейсов BLAST на сервере NCBI, EMBL-EBI и UniProt.
Таблица 3.Результаты сравнения веб-интерфейсов BLAST
| NCBI | EMBL-EBI | UniProt | |
| Очень тонкая настройка запроса | да | нет | совсем нет |
| Usability | Тяжел на превый взгляд | Всё весьма лаконично | Минимализм во всем |
| Представленность различных БД | есть всё | есть почти всё | что-то есть, но далеко не всё |
| Представление данных | Наглядно, поэтапно | Наглядно, настраиваемо | Чуть более наглядно, но менее настраиваемо |
| Ввод по идентификаторам | да | не нашел | да |
| Скачивание полученных данных | Не всё настраиваемо | Всё настраиваемо | Есть уже настроенное, и даже хорошо |
Вывод: Для работы с белками удобно подойдет веб-интерфейс Uniprot (решается большинство задач, особенно связнных с белками), для более сложных задач, требующих некоторой более тонкой настройки лучше подходит веб-интерфейс EMBL-EBI. Сервер NCBI можно описать словами "можно сделать что угодно и как угодно", т.е. доступна очень тонкая настройка всего, что только можно настроить в принципе, это радует, но не всегда нужно (если не нужно, то см. перые два сервера).
