Создание репрезентативной выборки.
Алгоритм создания выборки:- Определяется таксоноспецифичность работы алгоритма BLAST, в частности исключается родной филум Firmicutes, т.к. очевидно что именно в нем изучаемы белок будет слишком хорошо представлен, в результате чего репрезентативности выборки может не быть как таковой, т.к. фактически выборка слишком зашумлена данными из этого филума на фоне других.
- Определение оптимальных параметров для работы алгорима, а именно: колличество хитов, e-value и на самом деле уровень покрытия, но т.к. на прямую ограничение по нему не задать, то о будет косвенно учтен при задании лимитирующего e-value
- Осмысленное повторение пунктов 1 и 2 для случая эукариотичной выборки
Таблица 1.
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов | Реальное количество хитов |
| По прокариотам | BLASTP | Refseq | не влкючать Firmicutes не включать Eukaryota | 7e-24 | 5000 | 1361 |
| По эукариотам | BLASTP | Refseq | включать Eukaryota | 1e-7 | 100 | 38 |
Дополнительно, в виду весьма большого к-ва дынных был задействован сервис NCBI Batch Entrez.
Таблица 2 Выборка гомологов.
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Не найдено | ||
| Bacteria | Acidobacteriales | Granulicella tundricola MP5ACTX9 | 5 |
| Aquificae | Mesorhizobium loti MAFF303099 | 12 | |
| Deferribacterales | Flexistipes sinusarabici DSM 4947 | 4 | |
| fusobacteria | Fusobacterium ulcerans ATCC 49185 | 5 | |
| green sulfur bacteria | Chloroherpeton thalassium ATCC 35110 | 12 | |
| proteobacteria | Yersinia enterocolitica subsp. enterocolitica 8081 Thalassolituus oleivorans MIL-1 Escherichia coli KTE4 Salmonella enterica subsp. enterica serovar Gallinarum str. 287/91 Wolbachia endosymbiont of Drosophila simulans wNo Labrenzia alexandrii DFL-11 Octadecabacter antarcticus 307 Octadecabacter arcticus 238 Herbaspirillum frisingense GSF30 Taylorella asinigenitalis 14/45 Candidatus Burkholderia kirkii UZHbot1 Azoarcus sp. KH32C Helicobacter heilmannii ASB1.4 Helicobacter pylori OK310 Helicobacter pylori CCHI 33 Helicobacter pylori CPY1662 Desulfotignum phosphitoxidans DSM 13687 Desulfovibrio africanus PCS Desulfocapsa sulfexigens DSM 10523 |
1164 | |
| Eukaryotes | apicomplexans | Plasmodium cynomolgi strain B | 9 |
| green plants | Ostreococcus tauri | 9 |
Таким образом были отобраны 26 белковых последовательностей (список gi). Их таксономия более наглядно представленна на иллюстрации 1.
Иллюстрация 1. Дерево взаимоотношений отобранных гомологов.

В результате выполнения отбора гомологов получен FASTA файл содержащий 26 гомологов и исходный белок и FASTA файл подготовленый для проведения множественного выравнивания.
Множественное выравнивание
Было проведено множественное выравнивание 26 гомологов и самого белка CLPQ_BACSU программой muscle при стандартных настройках.
Тут и далее в работе будет применена следующая цветовая схема выравнивания:Минимальная ("нейтральная") аминокислота: бирюзовый
Полярные незаряженные аминокислоты:светло-коричневый
Отрицательно заряженные аминокислоты: синим
Положительно заряженные аминокислоты: красным
Ароматические аминокислоты: малиновым
Серусодержащие аминокислоты: желтым
Алифатические аминокислоты: темно-зеленым
Илллюстрация 2. Множественное выравнивание без обработки.
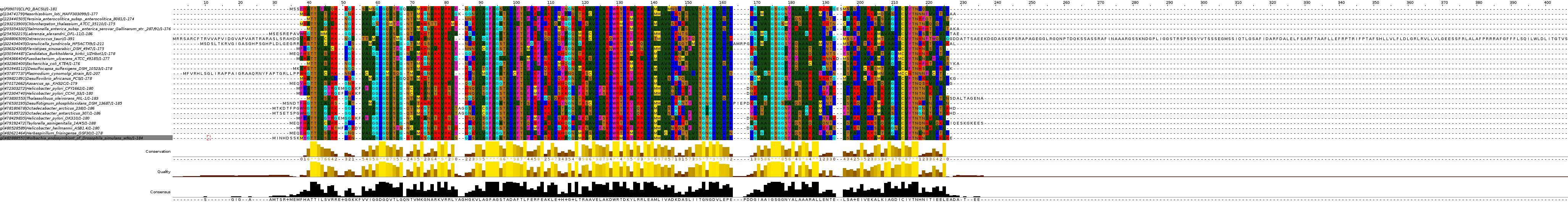
После этого была проведена некоторая визуализация выравнивания: цветовую обработку имеют только те участки, идентичность в которых превышает 55%
Илллюстрация 3. Множественное выравнивание с небольшой обработкой.

Просе этого выравнивание было дополнено указанием дополнительной информации о структуре и функциях:
- поле structure :альфа спирали обозначены красным, бета листы обозначены зеленым
- поле ligand: сами места связывания с лигандом обозначены L
- поле BLOCKS: символом B обозначены гомологичные блоки
- поле Conservation и поле Consensus: показывают консервативность позиции белка.
Выравнивание с этой обработкой явно показывает очень сильную гомологию, особенно в зоне BLOCKS.
Илллюстрация 4. Множественное выравнивание после обработки.

Результат множественного выравнивания
В целом выравнивание получилось очень хорошее (Илюстрация 4.), т.к существует большое колличество консервативных участков на протяжении всего выравнивания.
Многие консервативные участки по положению совпадают с некоторыми элекментами вторичной структуры, а именно с альфаспиралями. Особо сильных совпадений же по бета-листам не наблюдается. Это связано с фунцкциональной важностью вторичной структуры и, как следствие, из-за этого и происходит консервативность.
Особо плохих участков в выравнивании не наблюдается, в в иду хорошей подготовки выборки.
В основном, "колонки-гэпы" не встречаюстя в местах, формирующих вторичную структуру. За исключением бета-листов, что никоем образом не отражается функционально, т.к. в целом для белка не важно будет ли бета-лист чуть длинее или короче.
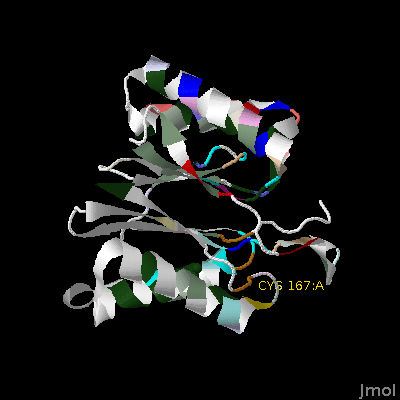
Это хорошо проглядывается на изображении трехмерной структуры белка, где окрашивание повторяет окрашивание множественного выравнивания ( Иллюстрация 5.)
Илллюстрация 5. Белок CLPQ_BACSU. Модель, раскрашивание идентично такому в множественном выравнивании, желтым обозначен консервативный участок связвывания лиганда.
Выборка блоков была проведена на основе стабильных консервативных участнов, судя по выравниванию, они в основном совпадают с форменными элементами вторичной структуры.
Консервативность участков, связывающих лиганды сильно высока: два из трех (Cys167 и Thr170) вообще не меняются на протяжении всей выборки, а у теретего остатка происходит смена Ala<->Gly, что по сути практически никак не влияет на связывание лиганда.
Вывод: Очевидно, при такой степени консервативности можно с легостью рассуждать, что на основе данного множественного выравнивания можно строить фукнциональные предсказания для других белков.
