1. Доменная структура белка CLPQ_BACSU по данным Pfam
Таблица 1. Доменная структура белка CLPQ_BACSU по данным Pfam
Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов |
Положение в последовательности белка CLPQ_BACSU | Клан |
| 1. | PF00227 | Proteasome | Семейство доменов названо по виду белков: протеасом, которые выполняют функцию расщепления коротких пептидов и пренадлежат к классу гидролаз, т.е. расщепляют путем каталитической реакции гидролиза | 3–180 | Клан NTN (CL0052) содержит 14 семейств: AAT, Asparaginase_2, CBAH, DUF1933, DUF3700, G_glu_transpept, GATase_2, GATase_4, GATase_6, GATase_7, Penicil_amidase, Peptidase_C69, Phospholip_B, Proteasome |
2. Данные о домене Proteasome
Таблица 2. Данные о домене Proteasome по данным Pfam
| Во сколько разных архитектур входит домен? | Этот домен входит в 55 архитектур |
| Для какого числа белков, содержащих домен, известна последовательность? | Последоваетльность известна для 10806 белков, среди которых число содержащих только этот домен 7199. |
| Для какого числа разных белков, содержащих домен, определена пространственная структура (домена или всего белка)? | Пространственная структура определена для 60 уникальных белков. |
| Выравнивание "seed" фрагментов белков, соответствующих домену | Доступно для скачивания в формате MSF |
| Оценка достоверности | Как явно видно из визуализации построенного множественного выравнивания, белки содержащие этот домен явно обладают гомологией. |
3. Описание встречаемости в различных таксонах доменной архитектуры вида  , состоящей из доменов PF10584 (Proteasome_A_N) и PF00227 (Proteasome)
, состоящей из доменов PF10584 (Proteasome_A_N) и PF00227 (Proteasome)
Таблица 3. Представленность домена PF10584 в организмах разных таксонов
|
Таксон
|
Количество белков с доменом PF10584.
|
|
| Эукариоты | Зеленые растения | 340 |
| Грибы | 892 | |
| Животные | 1028 | |
| Остальные эукариоты | 443 | |
| Археи | 187 | |
| Бактерии | 0 | |
| Вирусы | 0 | |
Таблица 4. Представленность домена PF00227 в организмах разных таксонов
|
Таксон
|
Количество белков с доменом PF00227.
|
|
| Эукариоты | Зеленые растения | 764 |
| Грибы | 1989 | |
| Животные | 2792 | |
| Остальные эукариоты | 1011 | |
| Археи | 429 | |
| Бактерии | 3590 | |
| Вирусы | 0 | |
Вывод:Не смотря на кажущуюся функциональную схожесть доменов, явно наблюдается некоторое различие в их распределении по таксонам.
Так, домен PF10584 никак не представлен среди бактерий, в отличие от домена PF00227 для которого, фактически, бактерии являются основной группой.
Кроме этого, наблюдаются некоторые сходные черты: распределение доменов по царствам эукариот весьма хорошо коррелируют между собой, кроме этого оба домена никак не представлены среди вирусов.
4. Сравнение описание мотивов в разных банках семейств, по данным InterPro.
Иллюстрация 1. Схема разметки всех мотивов на основании данных InterPro
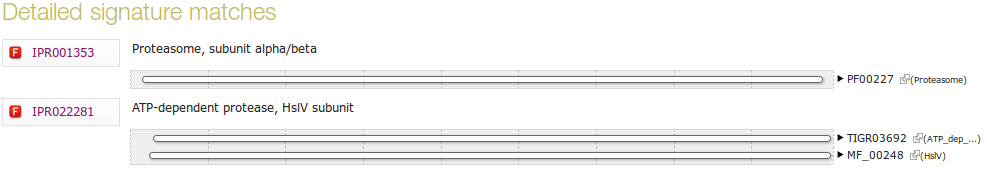
| Как называется самый короткий мотив? В каком банке он описан? |
(Family IPR001353) ,
Proteasome, subunit alpha/beta
Описан в банке Pfam |
| Как называется самый длинный мотив? В каком банке он описан? |
(Family IPR022281) ,
ATP-dependent protease, HslV subunit Описан в базе HAMAP |
| Какие структурные подписи интегрированы в InterPro? | Никаких 
|
| Отличаются ли границы структурных доменов от границ доменов Pfam? Если да, то как? | не определить, не хватает данных |
