Программа getorf пакета EMBOSS
При помощи программы enthret получена записть из банка embl с идентификатором D89965. Процесс получения записи и сама запись из базы данных представлена в Таблице 1.
Получение записи embl:D89965
enthret embl:D89965 task_1_embl_D89965.txt
Таблица 1. Получение записи из бд embl с идентификатором D89965 и её содержание.
Далее были найдены и транслированы ORF. Трансляция проведена по стандартной таблице, подобраны только ORF длины 30 аминокислотных остатков и больше, а также начинающиеся со старт-кодона (или начала последовательности) и заканчивающиеся стоп-кодоном (или концом последовательности). Процесс и результат поиска представлен в Таблице 2.
Получение записи embl:D89965
getorf task_1_embl_D89965.txt -table 0 -minsize 90 -find 1 -outseq task_1_orfs.fasta
task_1_orfs.fasta
Таблица 2. Процесс получения и сами транслированные по стандартной таблице ORF из записи embl:D89965.
Для определения, какая из найденных ORF полностью и частично соответствует записи FT из embl:D89965 была запущена последовательность команд представленая в Таблице 3. В Таблице 3 так же представлены файлы полученные в результате работы данных команд.
Получение ORF и её белковой последовательности, поиск её среди полученных командой getorf.
extractfeat task_1_embl_D89965.txt -type CDS -join -outseq task_1_CDS_nucl.fasta
coderet task_1_embl_D89965.txt -nocds -mrna -norest -translationoutseq task_1_CDS_translated.fasta
blastp -query task_1_CDS_translated.fasta -subject task_1_orfs.fasta -outfmt 6 -evalue 1e-7 -out task_1_blastp.txt
task_1_CDS_nucl.fasta
task_1_CDS_translated.fasta
task_1_blastp.txt
Таблица 3. Процесс идентификации какой именно из найденных ORF соответствует CDS указанная в записи embl:D89965.
Как видно из Таблицы 2 CDS указанной в embl:D89965 соответствует 3-я найденная ORF (в task_1_orfs.fasta обозначается D89965_3 )
Для определения какой из полученных ORF соответствует запись в Swiss-Prot проведем последовательность команд представленную в Таблице 3. Так же в Таблице 3 представлен результат выполнения этих команд.
Для определения, какая из найденных ORF полностью и частично соответствует записи FT из embl:D89965 была запущена последовательность команд представленая в Таблице 3. В Таблице 3 так же представлены файлы полученные в результате работы данных команд.
Получение белковой последовательности из Swiss-Prot. Поиск соответствующей ей ORF.
seqret sw:P0A7B8 task_1_sw_prot.fasta
blastp -query task_1_sw_prot.fasta -subject task_1_orfs.fasta -outfmt 6 -evalue 1e-7 -out task_1_blastp_sw_orf.txt
task_1_sw_prot.fasta
task_1_blastp_sw_orf.txt
Таблица 4. Процесс идендификации ORF которой соответствует белок в Swiss-prot, на который ссылается embl:D89965
Как видно из результатов Таблицы 4 ORF которой частично соответствует белок в Swiss-prot, на который ссылается embl:D89965 является 5-я найденная ORF (в task_1_orfs.fasta обозначается D89965_5 ). При этом найденный белок отностся к E.coli, в то время как данные ORF относятся к Rattus norvegicus.
Это можно объяснить тем, что в процессе пробоподготовки к секвенированию скорее всего произошло загрязнеие образца посторонними мРНК (т.к. секвенировались мРНК эпителия желудка, то очень вероятно, что там встретилась кишечная палочка.) После чего в автоматическом режиме, скорее всего путем blast'a по базе, была проведена аннотация записи embl:D89965.
Файлы-списки
Для получения fasta-файла с последовательностями алкогольдегидрогеназ организмов: CLOBE,DROTE,ARATH,CERCO,KLUMA,DROMU,METM5,DROLE,RAT,RABIT,NEUCR,MAIZE,MAIZE,STAAN,STAES,CLOAB,DROME,MAIZE,HUMAN,DROHY
была выполнена определенная последовательность команд, представленная в Таблице 5.Получение всех алкогольдегидрогеназ из Swiss-Prot
seqret sw:adh*_* adh.fasta
Организмы для фильтрации
Фильтрация usa по организмам
infoseq adh.fasta -only -usa -out stdout|grep -f organisms.txt > task_2_usa_list.txt
task_2_usa_list.txt
Получение всех последовательностей алкогольдегидрогеназ принадлежащих огранизмам из списка
seqret @task_2_usa_list.txt org_filtered_adh.fasta
Таблица 5. Процесс получения fasta-файла, содержащего алкогольдегидрогеназы определенных организмов.
EnsEMBL
При помощи сервиса SRS, что быстро и надежно, выяснили, что белку sw:GUSP1_HUMAN соответствует mRNA из EMBL:BT006760. Далее программой seqret получена последовательность кодирующая данный белок , которая представлена в Таблице 6.
Получение последовательности, кодирующей GUSP1_HUMAN
seqret embl:BT006760 GUSP1_HUMAN_cds.fasta
GUSP1_HUMAN_cds.fasta
Таблица 6. Процесс получения последовательности, кодирующей GUSP1_HUMAN.
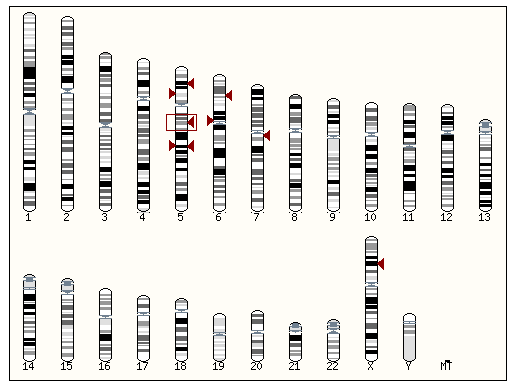
Рис. 1 Кариотип человека, на котором отмечены локусы к которых довольно большое количество хитов поиска последовательности, кодирующей белок GUSP1_HUMAN. Наиболее вероятная локализация показана прямоугольником (5-я хромосома).
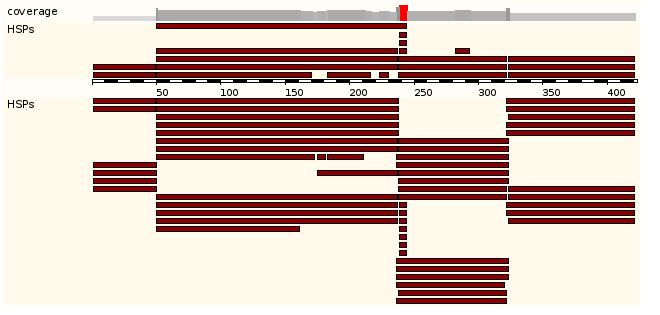
Рис. 2 Выравнивание хитов против последовательности кодирующей GUSP1_HUMAN.
При нажатии на [C] около какого-либо хита мы перейдем в режим Contig View. В этом режиме (полностью интерактивном) мы можем рассмотреть детальное расположение хита в хромосоме (Рис. 3), в контиге (Рис. 4) и детальное окружение хита (Рис. 5).
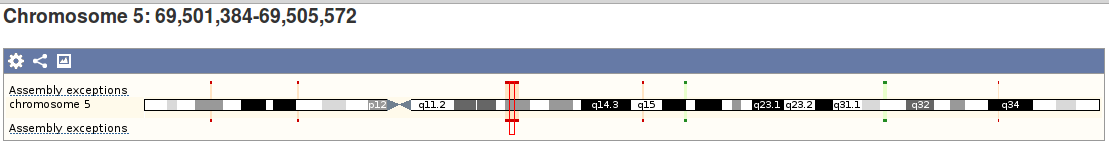
Рис. 3 Расположение лучшего хита на 5-й хромосоме человека.

Рис. 4 Выравнивание хитов против последовательности кодирующей GUSP1_HUMAN.
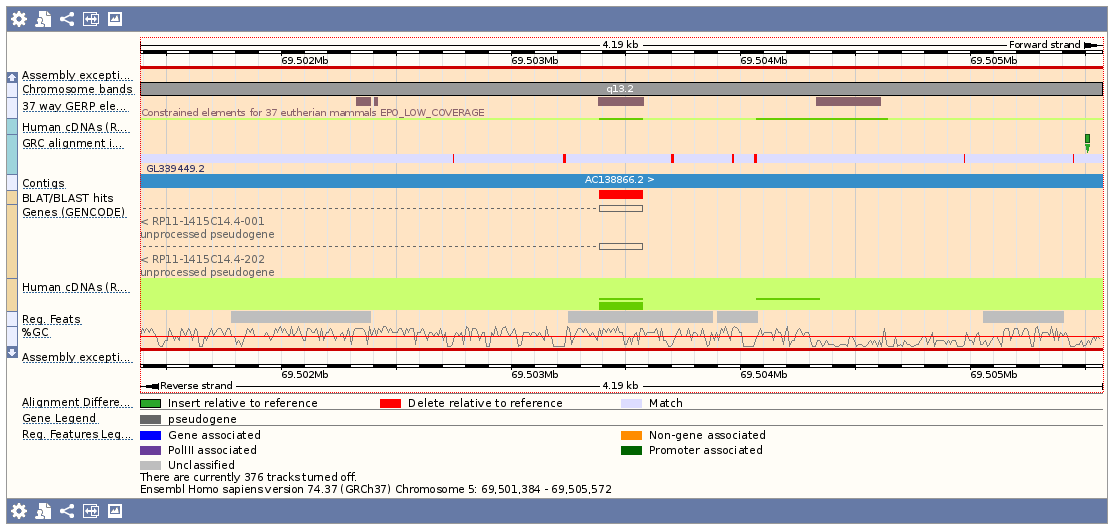
Рис. 5 Детальное окружение хита.
Сервис EnsEMBL довольно сильно информативен, как и другие биоинформатические сервисы он является как аггрератором биологических данных, так и весьма серьезным инструментом для работы с ними.
Отдельно следует упомянуть сервис EnsEMBL просмотра поорганизменной информации о кариотипе.
Кроме этого в данный сервис включено довльно большое колличество средств для сравнительной геномики (поиск по геномам, получение данных о вариативности и т.д.)
Так же даный сервис содержит другие различные средства: сравнение экспресии генов по тканям, анализ фенотипических данных / данных о генетических заболеваниях.
